Word2Vec 모델 기반의 단어 유사도를 이용한 새로운 약물-부작용 관계 예측
초록
약물의 부작용은 사람들의 건강에 위험을 초래하며, 심각한 경우 죽음에 이르게 할 수도 있다. 이러한 위험성 때문에 약물의 알려지지 않은 부작용을 찾아내는 일은 매우 중요하다. 본 연구에서는 텍스트 마이닝을 이용하여 기존에 알려진 약물의 부작용 이외의, 새로운 부작용을 예측할 수 있는 방법을 제안한다. 워드 임베딩의 한 모델인 Word2Vec 모델을 이용하여 생물학적 문헌에 언급되는 약물과 부작용을 벡터로 매핑하고, 이 벡터를 이용하여 약물 쌍과 부작용 쌍의 유사도를 계산한다. 약물 쌍과 부작용 쌍의 유사도를 기반으로 벡터로 매핑 된 값이 존재하는 모든 약물-부작용 쌍의 관계 점수를 구하였으며 이 관계 점수가 높을수록 약물-부작용 쌍이 실제로 관련이 있는 것으로 예측한다.
Abstract
Side effects of drugs pose a risk to people's health, and in severe cases can lead to death. Because of these risks, it is very important to identify unknown side effects of drugs. In this study, we propose ways to predict new side effects of drugs in addition to the side effects which have been previously known using text mining. Using the Word2Vec model which is one of word embedding models, drugs and side effects mentioned in biological literature are mapped to vectors, and similarities of drug pairs and similarities of side effect pairs are being calculate using these vectors. Using these similarities, we calculate relationship scores for all drugs and all side effects which could be mapped to vectors. Higher score of this relationship predicts that the drug-side effect pair is actually related.
Keywords:
text mining, data mining, Word2Vec, side effect prediction, bioinformaticsⅠ. 서 론
매년 전 세계의 수많은 사람들이 심각한 약물 부작용으로 인해 사망하는 것으로 보고되고 있다. 예를 들어, 미국에서는 매년 약 2백만 건의 심각한 부작용이 보고되어 약 10만 명이 사망한다고 한다. 이는 미국의 4번째 주요 사망 원인으로 추정된다[1].
이러한 부작용의 위험성은 약물의 개발 실패 원인 중 1/3을 차지한다[2]. 하지만 약물이 시중에 출시되었음에도 불구하고 사용 승인을 받기 위한 임상 실험에서 발견되지 않은 많은 부작용들이 존재한다[3]. 그렇기 때문에 약물이 시장에 상용화된 후에도 이미 알려진 부작용 이외의 새로운 부작용이 나타나는지에 대한 감시와 새롭게 나타날 수 있는 부작용에 대한 예측이 중요하다. 하지만 생물학적인 실험 방법을 통한 약물 부작용의 발견은 비용과 시간이 많이 소모된다[4]. 이러한 문제를 보완하기 위하여 컴퓨터를 이용한 실험이 활발하게 이루어지고 있다. 컴퓨터를 이용한 실험은 약물에서 발견될 수 있는 부작용에 대한 범위를 줄여 실험 결과를 빠르게 도출하여 실험에 소요되는 비용과 시간을 줄일 수 있도록 도와준다.
Yamanishi의 연구에서는 약물의 화학적 구조와 생물학적 정보인 단백질 타겟을 통합해 특성(Feature)으로 사용한 커널 회귀(Kernel regression) 모델을 적용하여 약물의 부작용을 예측했다[5]. Zhao의 연구에서는 fingerprint의 유사성, 구조의 유사성, ATC 코드, 문헌의 관계, 타겟 단백질로 이루어진 약물의 5가지 특성을 랜덤 포레스트(Random forest)를 이용해 분류하여 약물의 부작용을 예측하는 모델을 구축하였다[6]. Sukyung의 연구에서는 화학적 구조, 타겟 유전자 정보와 같은 약물의 정보 외에도 다양한 적응증 데이터베이스로부터 수집한 약물의 표현형 정보를 특징으로 사용하여 기계학습 기법을 이용한 약물 부작용의 예측 모델을 구축하였다[7].
이 밖에도 지난 몇 년간 양이 빠르게 증가하고 있는 생물학적 문헌을 바탕으로 텍스트 마이닝(Text mining)을 이용한 연구들이 활발하게 이루어지고 있다[8]. 텍스트 마이닝은 컴퓨터를 이용한 실험 방법 중 하나로[9], 비정형 데이터(Unstructured data)로부터 기존에 알려지지 않은 정보를 도출해내는 방법이다[10]. Min의 연구에서는 텍스트 마이닝의 방법 중 개체명 인식(Named entity recognition)과 관계추출을 이용하여 문장의 ‘약물-단백질’, ‘단백질-단백질’, ‘단백질-부작용’의 관계를 추출해냈다. 추출한 ‘약물-단백질’ 관계와 ‘단백질-부작용’ 관계 사이에 공통된 ‘단백질-단백질’ 관계가 존재할 경우 경로로 연결하여 새로운 부작용을 추론하였다[11].
본 연구는 텍스트 마이닝 기법 중에서도 단어를 벡터로 바꿔주는 워드 임베딩(Word embedding)을 이용한 약물 쌍과 부작용 쌍의 유사도를 기반으로 한 새로운 약물-부작용 관계 예측 방법을 제안한다. 본 연구는 먼저 생물학적 문헌의 초록을 문장의 형태로 바꾸는 전처리를 진행하였다. 전처리한 초록의 문장들을 이용하여 워드 임베딩 모델 중에서 Word2Vec 모델의 학습을 진행하였다. 그 후 학습된 Word2Vec 모델로부터 얻은 약물 쌍과 부작용 쌍의 코사인 유사도를 계산하였다. 계산된 유사도를 이용하여 모든 약물-부작용 쌍에 대하여 관계 점수를 계산하고 점수의 높고 낮음을 통하여 약물-부작용 쌍의 관계를 추론할 수 있다.
본 연구에서는 약물-부작용 쌍의 관계 점수를 이용한 관계 추론의 정확도를 AUC(Area Under the ROC Curve)로 보였다. 또한 Fisher’s exact test를 이용하여 알려지지 않은 약물-부작용 쌍의 관계 예측이, 통계적으로 유의미함을 검증하였다. 비교 실험을 통하여 본 연구에서 사용된 Word2Vec 모델 기반의 유사도를 사용한 방식이 단어의 동시 출현 기반의 유사도를 사용한 방식보다 정확도 면에서 뛰어난 것을 확인하였다.
본 논문의 구성은 다음과 같다. 2장에서는 본 연구에 사용된 관련된 연구와 기법에 관하여 소개한다. 3장에서는 본 연구의 시스템 개요와 연구에 사용된 데이터에 대한 설명과 실험 과정이 기술되어 있으며, 4장에서는 실험에 대한 결과와 비교 실험, 예측에 대한 검증을 기술하였다. 5장에서는 본 연구에 대한 결론과 향후 연구 방향을 기술하였다.
Ⅱ. 관련 연구
2.1 Word2Vec
자연어 처리의 기법 중에는 단어를 벡터로 매핑 시켜주는 워드 임베딩이라는 방식이 있다. Word2Vec는 워드 임베딩 방식 중에서도 널리 쓰이는 모델 중 하나이다. 워드 임베딩은 비슷한 분포를 가진 단어는 유사한 뜻을 가진다는 분산 가설(Distributional hypothesis)을 기반으로 한다. 워드 임베딩을 통한 단어의 벡터 화는 신경망 모델을 이용하여 이루어졌다. Word2Vec는 기존 워드 임베딩 모델의 학습 방법보다 간단한 계산 복잡도를 가지고 있어, 기존의 방법에 비해 효율적으로 학습을 가능하게 한 모델이다.
Word2Vec의 학습 알고리즘에는 2가지 모델이 있다. 첫 번째는 문맥을 바탕으로 단어를 예측하는 CBOW(Continuous Bag Of Words) 모델과 단어를 바탕으로 문맥을 예측하는 skip-gram 모델이 있다[12].
본 연구에서는 약물과 부작용을 벡터로 매핑 하기 위하여 CBOW 모델이 사용되었다.
2.2 코사인 유사도
코사인 유사도는 텍스트 마이닝 기법에서 벡터로 표현된 단어나 문서의 유사도를 구할 때 주로 사용되는 유사도 계산 방법이다.
코사인 유사도는 두 벡터간의 코사인 각도를 이용해서 구하는 벡터간의 유사도를 의미한다. 코사인 유사도는 -1 과 1 사이의 값을 가지며 값이 1에 가까울수록 유사도가 높다. 코사인 유사도를 구하는 식은 식 (1)과 같다.
| (1) |
본 연구에서는 약물 간 유사도와 부작용 간 유사도를 계산하는데 사용되었다[13].
2.3 AUC 계산
본 연구에서 계산된 약물과 부작용의 점수의 예측 성능을 측정하기 위하여 ROC(Receiver Operating Characteristic) 곡선과 AUC를 사용하였다.
실제가 참인 경우(True)인데 참이라고 예측한 경우(Positive)를 TPR(True Positive Rate)라고 한다. 실제가 거짓(False)인데 참이라 잘못 예측한 경우(Positive)를 FPR(False Positive Rate)라고 한다. ROC곡선은 TPR과 FPR을 이용하여 그려지는 그래프이다. ROC 곡선의 수직축은 TPR, 수평축은 FPR을 나타낸다.
AUC 값은 ROC 곡선 아래의 면적이다. AUC 값이 1.0에 가까울수록 분류의 정확도가 높다고 할 수 있고, 0.5 이하일 경우 일반적으로 분류의 의미가 없다고 판단한다[14].
본 연구에서는 관계 점수가 계산된 모든 약물-부작용 쌍과 알려진 약물-부작용 관계를 이용하여 AUC 값을 구하여 관계 점수를 이용한 예측의 정확도를 측정하였다.
2.4 Fisher’s exact test
Fisher’s exact test는 표1과 같이 두 그룹에 대하여 작성된 분할표를 바탕으로 두 그룹간의 연관성이 있는지 알아보고자 할 때 쓰이는 검정 방법이다.
분할표에 대한 유의확률 p는 식 (3)과 같이 구할 수 있다. 이 식에서 x, y, z, w는 표 1에서의 각 셀의 값을 의미하고, n은 식 (2)와 같이 모든 셀의 값의 합을 의미한다.
| (2) |
| (3) |
유의확률 p가 0.05 미만인 경우, 두 집단이 통계적으로 관련이 있다고 판단한다[15].
본 논문에서의 연구 방법으로 새로운 약물-부작용 쌍의 예측을 통계적으로 검증하기 위하여 Fisher’s exact test가 사용되었다.
Ⅲ. 연구 방법
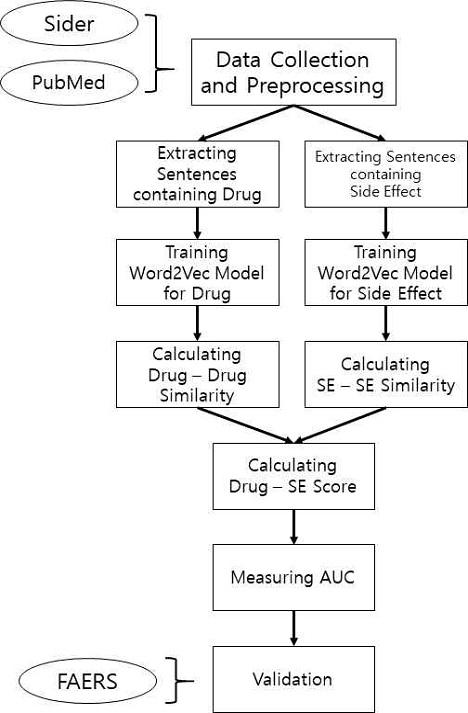
본 연구의 전체 시스템 개요는 그림 1과 같다. 먼저, 연구에 사용하기 위한 데이터를 PubMed와 SIDER로부터 수집 및 정제하였다.
Word2Vec 모델의 학습에 사용하기 위하여, SIDER로부터 수집한 약물과 부작용의 이름이 한 번이라도 언급된 문장들을 정제된 문헌 데이터로부터 추출하였다.
추출된 문장으로 Word2Vec 모델을 학습시키고 그로부터 얻은 약물과 부작용의 벡터를 바탕으로 약물 쌍과 부작용 쌍의 유사도를 계산한다. 약물 쌍과 부작용 쌍의 유사도와 기존의 알려진 약물-부작용 관계를 이용해서 모든 약물과 부작용의 쌍에 대하여 점수를 계산한다.
위에서 구한 약물과 부작용의 쌍의 관계에 대한 점수가 높을수록 약물과 부작용이 실제로 관련이 있을 가능성이 높다고 예측한다. 이 예측의 정확도를 측정하기 위하여 AUC 값을 계산한다. 그 후 예측이 통계적으로 유의한지 확인하기 위하여 검증을 진행한다.
3.1 데이터 수집
본 연구는 문헌을 기반으로 이루어지기 때문에 많은 양의 문헌 데이터가 필요하다. 본 연구에서는 생물학 문헌을 제공하는 데이터베이스인 PubMed를 사용하여 19년 12월 16일에 업데이트된 19,936,675개의 논문의 초록을 수집하였다[16]. 수집한 초록은 문장 단위로 정제하여 사용하였다.
SIDER는 시판되는 약물의 부작용 정보를 기록하는 데이터베이스이다. 본 논문에서는 SIDER 4.1 버전에서 약물-부작용 관계 158,095쌍을 수집하였다. SIDER에서 제공되는 약물의 이름은 FDA로부터 제공되는 약물의 라벨이 사용되었고, 부작용의 명칭은 MedDRA(Medical Dictionary for Regulatory Activities)의 용어가 사용되었다. 수집한 약물-부작용 관계에서 한 번이라도 등장한 약물의 이름 1,344개, 부작용의 명칭 6,123개를 약물과 부작용의 사전으로 쓰기 위하여 추출하였다[17].
3.2 Word2Vec 모델 학습
약물과 부작용을 벡터로 매핑하는 Word2Vec 모델의 학습을 위하여, PubMed로부터 수집하고 정제된 문장들로부터 SIDER에서 수집한 약물 혹은 부작용이 한 번 이상 언급된 문장들을 각각 추출했다.
약물을 벡터로 매핑 할 때 사용되는 모델의 학습을 위하여 약물이 한 번 이상 언급된 문장 88,489,956개가 사용되었다.
부작용을 벡터로 매핑 할 때 사용되는 모델의 학습을 위하여 부작용이 한 번 이상 언급된 문장 40,140,939개가 사용되었다.
각 모델은 학습에 사용한 문장에서 한 번이라도 언급된 모든 단어를 대상으로 학습되었다. 그 결과 SIDER를 통하여 얻은 약물과 부작용의 목록 중 학습에 사용된 문장에서 한 번이라도 언급된 적이 있는 약물 1,289개와 부작용 4,776개의 벡터가 생성되었다.
3.3 약물 쌍과 부작용 쌍의 유사도 계산
Word2Vec 모델로부터 벡터를 얻을 수 있는 약물 1,289개와 부작용 4,776개를 대상으로 모든 약물 쌍과 부작용 쌍의 코사인 유사도 계산을 진행하였다.
유사도 계산 결과 약물 쌍의 유사도 1,660,232개와 부작용 쌍의 유사도 22,805,400개를 얻었다.
3.4 약물-부작용의 관계 점수 계산
벡터로 매핑된 값이 존재하는 약물과 부작용의 모든 쌍에 대하여 앞에서 구한 유사도들을 이용하여 약물-부작용의 관계 점수를 계산한다.
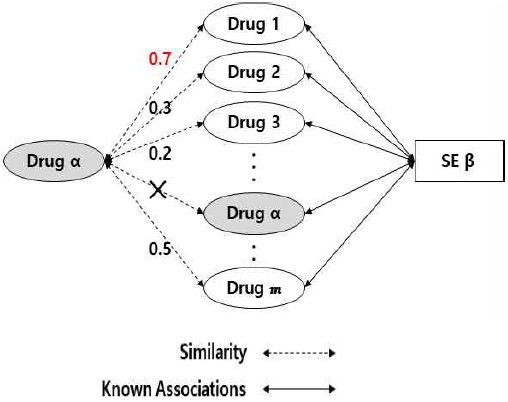
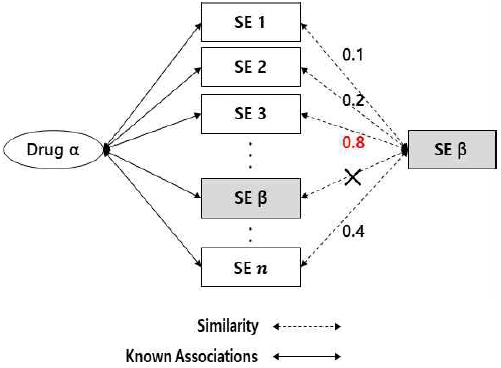
그림 2와 그림 3은 알려지지 않은 관계에 있는 특정 약물 Drug α와 특정 부작용 SE β의 관계 점수 계산 과정에 대한 예시이다. 부작용 SE β와 관계가 있다고 알려진 약물들과 약물 Drug α의 유사도 중에서 가장 높은 값을 유사도 simx라고 하고, 약물 Drug α와 관계가 있다고 알려진 부작용들과 부작용 SE β의 유사도 중에서 가장 높은 값을 유사도 simy라고 한다.
그림 2는 식 (4)와 같이 유사도 simx에 대한 계산 과정을 나타냈다. 부작용 SE β와 관계가 있다고 알려진 약물의 목록 ‘Drug 1, Drug 2 … Drug m’ 이 있다. 알려진 약물의 목록 중에서 약물 Drug α와 유사도가 제일 높은 약물 1과 약물 Drug α의 유사도가 유사도 simx가 된다. 만약 알려진 약물의 목록에 약물 Drug α가 있을 경우 유사도 계산에서 약물 Drug α는 제외한다.
그림 3은 식 (5)와 같이 유사도 simy에 대한 계산 과정을 나타냈다. 약물 Drug α와 관계가 있다고 알려진 부작용 목록 ‘SE 1, SE 2 … SE n’ 이 있다. 알려진 부작용 목록 중에서 부작용 SE β와 유사도가 제일 높은 SE 3과 부작용 SE β의 유사도가 유사도 simy가 된다. 만약 알려진 부작용의 목록에 부작용 SE β가 있을 경우 유사도 계산에서 부작용 SE β는 제외한다.
| (4) |
| (5) |
| (6) |
식 (6)과 같이 위에서 구한 유사도 simx와 유사도 simy를 곱하면 약물 Drug α와 부작용 SE β의 관계의 점수가 된다.
벡터로 매핑된 값이 존재하는 약물 1,289개와 부작용 4,776개에 대하여 위의 계산 방법을 이용하여 모든 약물-부작용 쌍의 점수를 계산했다. 그 결과 총 6,109,753개의 약물-부작용 쌍에 대한 점수를 구하였다. 계산 된 점수가 높을수록 약물과 부작용이 실제로 관계가 있을 가능성이 높은 것으로 예측한다.
3.5 AUC 측정
위에서 계산된 6,109,753개의 모든 약물-부작용 쌍의 점수를 이용한 약물과 부작용의 관계 예측의 정확도를 검증하기 위하여 AUC 값을 측정하였다.
SIDER에서 제공하는 알려진 약물-부작용 관계 158,095쌍 중에서 약물-부작용 쌍에 계산에 사용된 약물 1,289개, 부작용 4,776개와 관련이 있는 140,411쌍을 사용하였다. 점수가 계산된 모든 약물-부작용 쌍의 목록 중 알려진 약물-부작용 관계에 해당하는 쌍은 True, 해당하지 않는 쌍은 False로 클래스 값을 부여하였다. 모든 약물-부작용 쌍을 점수 순으로 정렬한 후 그에 대한 단일 ROC곡선을 생성하였다.
생성된 ROC 곡선과 AUC 값을 이용하여 약물-부작용 쌍이 실제로 관계가 있을지 예측을 하기 위한 진단 기준치(Cut-off point)를 구하였다. 약물-부작용의 관계 점수가 진단 기준치보다 높을 경우 실제로 약물과 부작용이 관계가 있을 것으로 예측한다.
Ⅳ. 결 과
4.1 성능 평가
본 연구에서 제안된 방법의 약물-부작용 관계 예측이 정확한지 검증하기 위하여 AUC 값을 측정한 결과는 0.891이 나왔다.
예측을 위한 진단 기준치는 민감도와 특이도의 합이 제일 높은 수치가 나오는 1.631(민감도 0.7874 / 특이도 0.8436)인 지점인 0.4368이다. 약물과 부작용의 관계 점수가 0.4368보다 높을 경우 실제로 관계가 있을 것으로 예측한다.
4.2 비교 실험
본 연구에서 제안 방법의 성능 평가를 위하여 동시출현 빈도를 이용한 약물-부작용 점수의 계산을 이용한 단어의 동시 출현을 이용한 두 가지 비교 실험을 진행하였다.
비교 실험에서 사용된 데이터는 앞에서 수집한 데이터를 사용하였다. 사용된 문헌의 데이터는 PubMed로부터 수집한 19,936,675개의 논문의 초록을 사용하였다. 약물과 부작용의 목록은 SIDER로부터 얻은 약물의 이름 1,344개, 부작용의 명칭 6,123개를 사용하였다.
첫 번째는 단어의 동시 출현 빈도를 이용한 방법이다. 수집한 약물의 목록과 부작용의 목록을 바탕으로 약물의 동시 출현 빈도와 부작용의 동시 출현 빈도를 각각 구하였다. 그 후 약물의 동시 출현 빈도와 부작용의 동시 출현 빈도를 Min-Max 정규화를 통하여 정규화시킨 값을 각각 약물 쌍의 유사도와 부작용 쌍의 유사도로 사용했다. Min-Max 정규화에 사용되는 식은 식 (7)과 같다. 식 (7)에서 모든 출현 빈도 값 중에서 최댓값을 xmax, 최솟값을 xmin이라고 한다. 각각의 모든 출현 빈도 x에서 xmin을 빼준 값을 xmax에서 xmin를 뺀 값으로 나누면 x를 Min-Max 정규화 시킨 값 X가 나오게 된다.
| (7) |
두 번째는 단어의 동시 출현을 이용한 방법이다. 각 약물과 부작용이 문장에 동시에 출현했을 경우 1, 아닐 땐 0으로 채워진 약물-부작용의 동시 출현에 관한 매트릭스를 생성한다. 그 후 약물-부작용의 동시 출현 매트릭스를 기반으로 식 (8)과 같이 자카드 계수를 이용하여 약물 쌍의 유사도와 부작용 쌍의 유사도를 측정했다[12]. 식 (8)을 이용해서 약물쌍의 유사도를 구할 경우 A와 B가 동시 출현 매트릭스에서 각각 다른 약물에 해당하는 벡터라고 할 때 두 벡터의 합집합이 분모가 되고, 교집합이 분자가 된다. 자카드 계수는 0 과 1 사이의 값을 가지며 값이 1에 가까울수록 유사도가 높다.
| (8) |
위의 두 가지 방법을 통하여 구한 약물의 유사도와 부작용의 유사도를 이용하여 본 연구의 방법으로 모든 약물-부작용 쌍의 관계에 대한 점수를 계산하였다.
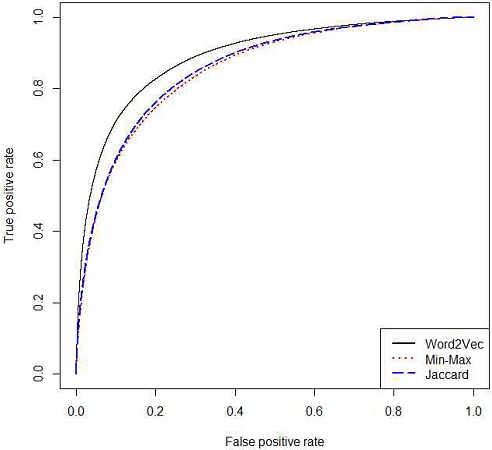
그림 4는 비교 실험의 결과를 이용하여 그려진 ROC 곡선들이다. 비교 실험들의 AUC값은 표 2와 같다.
비교 실험에서 사용된 단어의 동시 출현을 이용한 방법은 약물, 부작용의 같은 문장에서의 출현 여부 또는 빈도를 이용하여 약물, 부작용을 벡터로 매핑하는 방법이다. 단어의 동시 출현 방법은 사용한 약물, 부작용의 개수가 많아질수록 벡터의 차원이 늘어나게 된다. 게다가 이렇게 만들어진 벡터의 차원들은 대부분의 값이 0을 가지고 있기 때문에 벡터의 정보가 희소한 표현(Sparse representation)을 가지게 된다. 이러한 희소 벡터는 약물, 부작용의 특성을 벡터로 제대로 표현하지 못해 약물 쌍과 부작용의 쌍의 유사도가 비교적 정확하게 계산되지 못한다.
본 연구의 약물, 부작용을 Word2Vec를 이용하여 벡터로 매핑하는 방법은 단어의 동시 출현 방법보다 훨씬 적은 벡터의 차원을 가지고 있고, 각각의 차원들이 모두 정보를 가지고 있기 때문에 약물 쌍과 부작용의 쌍의 유사도를 보다 정확하게 계산할 수 있다.
Word2Vec를 이용한 본 연구의 AUC 값이 두 가지 비교 실험(Min-max, Jaccard)의 AUC 값보다 좋게 나온다는 것을 확인할 수 있다.
4.3 검증
검증을 위하여 연구 방법에서 사용되지 않은 데이터베이스인 FAERS(FDA Adverse Event Reporting System)로부터 알려진 약물-부작용 관계를 추출하였다. FAERS는 FDA에 제출된 시판된 약물에 대한 부작용 보고서이다[18].
본 연구에서는 FAERS에서 제공된 2012년 4분기부터 2020년 2분기까지의 데이터를 사용했다. FAERS에서, 본 연구에서 사용된 약물, 부작용과 관련이 있는 724,556개의 알려진 약물-부작용 관계를 추출하였다.
본 연구에서 계산된 6,109,753개의 모든 약물-부작용 쌍의 점수를 기반으로 한 약물-부작용 관계에 대한 예측과 FAERS에서 추출된 724,556개의 알려진 약물-부작용 관계를 바탕으로 검증을 위한 분할표를 작성하였다. 본 연구에서 계산된 모든 약물-부작용 쌍의 목록 중에서 FAERS를 통해 알려진 약물-부작용 쌍은 722,261개, 알려지지 않은 약물-부작용 쌍은 5,387,492개이다. FAERS를 통해 알려지지 않은 약물-부작용 쌍은 실제로 부작용이 존재하지 않는지는 알 수 없으며 추후 실험 혹은 시판된 약물에 대한 감시를 통해 새로운 약물-부작용 관계가 나타날 수 있다.
알려진 약물-부작용 쌍과 알려지지 않은 약물-부작용 쌍의 개수의 불균형을 해결하기 위하여 다음과 같은 과정을 진행하였다. 모든 약물-부작용 쌍에서 알려진 약물-부작용 쌍의 개수만큼 무작위로 골라낸 알려지지 않은 약물-부작용 쌍과 알려진 약물-부작용 쌍을 Fisher’s exact test를 위한 약물-부작용 쌍의 목록으로 사용하였다. 이와 같은 과정을 통해 만들어진 약물-부작용 쌍의 목록으로 FAERS와 본 연구를 통한 예측에 대한 Fisher’s exact test를 진행하였다. 이 과정을 100회 반복한 뒤 모든 Fisher’s exact test의 결과의 평균을 계산하였다. 그 결과 유의확률 p는 2.2e-16미만, odds ratio는 5.3418이 나왔다. 유의확률이 0.05 미만임으로 본 연구에서 예측된 약물-부작용 관계가 유의성이 있음을 알 수 있다.
Ⅴ. 결론 및 향후 과제
본 연구에서는 약물끼리의 유사도, 부작용끼리의 유사도, 기존에 알려진 약물-부작용 관계를 이용하여 새로운 약물-부작용 관계에 대한 예측을 진행하였다. Word2Vec 모델을 이용하여 약물끼리의 유사도와 부작용끼리의 유사도를 구하였다. 이를 기반으로 본 연구의 약물-부작용 관계 점수 계산 방법을 이용한 약물-부작용 관계 예측을 실시하였다. 약물-부작용 관계 예측의 AUC 값 측정과 Fisher’s exact test를 진행한 결과 본 연구의 관계 예측 방법이 통계적으로 유의미함을 확인했다. 이를 이용하여 약물에서 발생할 수 있는 부작용의 범위를 줄여 약물의 부작용에 대한 생물학적 실험에 소용되는 시간과 비용을 절약할 수 있을 것으로 기대한다.
본 연구에서는 약물과 부작용의 관계 예측 시 부작용의 위험성은 고려하지 않았다. 본 연구에서 예측된 약물과 부작용의 관계 점수의 상위권에 있는 부작용은 체중 증가, 체중 감소와 같은 부작용들이 있는데 이는 비교적 심각하지 않고 흔히 발생할 수 있는 부작용들이다. 향후 연구에서는 부작용의 위험성을 고려하여 심각한 위험을 가진 부작용에는 가중치를 부여하는 방식으로 약물의 사용 시 발생할 수 있는 부작용 중 심각한 위험성을 지닌 부작용을 우선적으로 예측할 수 있도록 하는 연구를 진행할 예정이다.
Acknowledgments
이 논문은 2018년도 정부(미래창조과학부)의 재원으로 한국연구재단(No.2018R1A2B6006223)의 지원을 받아 수행된 연구임
References
-
Kathleen M. Giacomini, Ronald M. Krauss, Dan M. Roden, Michel Eichelbaum, Michael R. Hayden, and Yusuke Nakamura, "When good drugs go bad", Nature, Vol. 446, pp. 975-977, Apr. 2007.
[https://doi.org/10.1038/446975a]

- Tony Kennedy, "Managing the drug discovery/development interface", Drug discovery today, Vol. 5, No. 9, pp. 409-414, Sep. 1997.
-
Nicholas P. Tatonetti, Patrick P. Ye, Roxana Daneshjou, and Russ B. Altman, "Data-Driven Prediction of Drug Effects and Interactions", Science Translational Medicine, Vol. 4, No. 125, pp. 125ra31, Mar. 2012.
[https://doi.org/10.1126/scitranslmed.3003377]

-
Nicholas P. Tatonetti, Tianyun Liu, and Russ B. Altman, "Predicting drug side-effects by chemical systems biology", Genome Biol, Vol. 10, No. 238, Sep. 2009.
[https://doi.org/10.1186/gb-2009-10-9-238]

-
Yoshihiro Yamanishi, Edouard Pauwels, and Masaaki Kotera., "Drug side-effect prediction based on the integration of chemical and biological spaces", Journal of chemical information and modeling, Vol. 52, No. 12, pp. 3284-3292, Dec. 2012.
[https://doi.org/10.1021/ci2005548]

-
Zhao Xian, Chen Lei, and Lu Jing, "A similarity-based method for prediction of drug side effects with heterogeneous information", Mathematical biosciences, Vol. 306, pp. 136-144, Dec. 2018.
[https://doi.org/10.1016/j.mbs.2018.09.010]

-
Sukyung Seo, Taekeon Lee, and Youngmi Yoon, “Prediction of Drug Side Effects Based on Drug-Related Information”, Journal of KIIT, Vol. 17, No. 12, pp. 21-28, Dec. 2019.
[https://doi.org/10.14801/jkiit.2019.17.12.21]

-
K. Bretonnel Cohen and Lawrence Hunter, "Getting Started in Text Mining", PLoS Computational Biology, Vol. 4, pp. e20, Jan. 2008.
[https://doi.org/10.1371/journal.pcbi.0040020]

-
Wilco W. M. Fleuren and Wynand Alkema, "Application of text mining in the biomedical domain", Mehods, Vol. 74, pp. 97-106, Mar. 2015
[https://doi.org/10.1016/j.ymeth.2015.01.015]

- Ah-Hwee Tan, "Text Mining: The state of the art and the challenges", Proceedings of the Pacific Asia Conf on Knowledge Discovery and Data Mining PAKDD'99 workshop on Knowledge Discovery from Advanced Databases, pp. 65-70, 1999.
-
Min Song, Seung Han Baek, Go Eun Heo, and Jeong-Hoon Lee, "Inferring Drug-Protein–Side Effect Relationships from Biomedical Text", Genes, Vol. 10, No. 2, pp. 159, Feb. 2019.
[https://doi.org/10.3390/genes10020159]

- Tomas Mikolov, Kai Chen, Greg Corrado, and Jeffery Dean, "Efficient Estimation of Word Representations in Vector Space", [online] arXiv preprint arXiv:1301.3781, , Sep. 2013.
- Anna Huang, "Similarity measures for text document clustering", In Proceedings of the 6th New Zealand Computer Science Research Student Conference, pp. 49–56, 2008.
- Jiawei Han, Micheline Kamber, and Jian Pei, "Data Mining: concepts and techniques third edition", Morgan Kaufmann Publisher Inc, pp. 373-377, 2011.
- Keith M. Bower, "When to use Fisher’s Exact Test", American Society for Quality, Six Sigma Forum Magazine, Vol. 2, No. 4, pp. 35-37, 2003.
- Kathi Canese and Sarah Weis, "PubMed: The Bibliographic Database", The NCBI Handbook [internet], Mar. 2013 [accessed: Dec. 29, 2019]
-
Michael Kuhn, Ivica Letunic, Lars Juhl Jensen, and Peer Bork, "The SIDER database of drugs and side effects", Nucleic acids research, Vol. 44, No. D1, pp. D1075–D1079, Jan. 2016.
[https://doi.org/10.1093/nar/gkv1075]

- The food and drug administration adverse event reporting system (FAERS), https://www.fda.gov/drugs/surveillance/questions-and-answers-fdas-adverse-event-reporting-system-faers, . [accessed: Sep. 28, 2020]

2019년 2월 : 가천대학교 컴퓨터공학과(공학사)
2019년 3월 ~ 현재 : 가천대학교 IT융합공학과(석사)
관심분야 : 데이터마이닝, 텍스트 마이닝, 바이오인포매틱스

2020년 2월 : 가천대학교 전자공학과(공학사)
관심분야 : 데이터마이닝, 텍스트 마이닝, 바이오인포매틱스

1981년 : 서울대학교 자연과학대학(학사)
1983년 : 오하이오 주립대학 수학과(학사 수료)
1987년 : 스탠포드대학교 컴퓨터 과학과 졸업(이학석사)
2008년 : 연세대학교 컴퓨터과학과졸업(공학박사)
1987년 5월 ~ 1993년 5월 : IntelliGenetics Inc., California, USA, Software Engineer
1995년 2월 ~ 현재 : 가천대학교 컴퓨터공학과 교수
관심분야 : 데이터베이스 시스템, 데이터 마이닝, 바이오인포매틱스, 소셜미디어 데이터 마이닝