안저사진 및 EMR 데이터 기반 멀티모달 녹내장 진단 모델
초록
녹내장은 심각한 시각 장애를 유발하는 질환으로, 심할 경우 실명까지 이어질 수 있다. 따라서 녹내장을 조기에 발견하는 것은 중요하다. 본 연구에서는 이미지 데이터인 안저사진과 수치형 정형 데이터인 EMR(Electronic Medical Records) 데이터를 기반으로 한 멀티모달 녹내장 진단 모델을 제안한다. 멀티모달은 여러 가지 데이터 유형 또는 모달리티를 결합하여 작동하는 모델을 의미한다. 본 연구에서는 EMR 데이터를 이미지로 변환하는 기법을 적용하여 이미지 피처를 추출하였다. 안저사진에 대해서도 전처리 후 이미지 피처를 추출하였다. 추출된 두 피처 벡터는 결합되어 앙상블 학습을 통해 최종 결과를 도출하였다. 본 모델의 성능을 검증한 결과, 약 93.31%의 분류 정확도를 달성하였다. 이것은 싱글모달 대비 약 1.21%, 다른 멀티모달 모델들과 비교했을 때도 약 0.44%가 향상된 결과이다.
Abstract
Glaucoma is a condition that can cause serious visual impairment and can even lead to blindness in severe cases. Therefore, early detection of glaucoma is important. In this study, we propose a multimodal glaucoma diagnosis model based on fundus photography, which is image data, and Electronic Medical Records(EMR) data, which is numerical structured data. Multimodal refers to a model that operates by combining multiple data types or modalities. In this study, image features were extracted by applying a technique to convert EMR data into images. Image features were also extracted from fundus photographs after preprocessing. The two extracted feature vectors were combined to produce the final result through ensemble learning. As a result of verifying the performance of this model, a classification accuracy of approximately 93.31% was achieved. This is an improvement of about 1.21% compared to single modal and about 0.44% compared to other multimodal models.
Keywords:
glaucoma, multimodal, image conversion of structured data, fundus image, EMR dataⅠ. 서 론
녹내장(Glaucoma)은 시신경 내 망막 신경절 세포(RGC, Retinal Ganglion Cell)와 축삭이 소실되어 시신경 병증으로 이어지는 안구 질환이다. 이러한 질환은 시야의 점진적인 손실로 이어질 수 있으며, 치료하지 않을 경우 실명에 이를 수도 있다[1][2]. 세계보건기구(WHO)에 따르면 녹내장은 시각 장애와 실명의 주요 원인 중 하나이다[3]. 세계 인구 중 40세 이상에서 녹내장의 연령 표준화된 유병률은 약 3~5%로 보고되며, 2040년까지 1억 1200만 명으로 증가할 것으로 예상되고 있다[4]. 일부 개발도상국에서는 녹내장의 90%가 발견되지 않고 있다는 연구가 있었으며[5], 미국에서는 녹내장을 앓고 있는 인구 중 약 절반이 녹내장을 앓고 있다는 사실을 모르고 있다는 연구가 있다[6].
녹내장의 진단에는 안저사진((Fundus image), 시야검사(Visual field test), 광간섭단층촬영(Optical coherence tomography)와 같은 다양한 검사 도구가 사용되고 신체 계측 정보, 안압검사등을 기록한 EMR(Electronic Medical Records) 자료도 활용된다. 임상의는 다양한 검사결과와 EMR 자료를 종합하여 녹내장 여부를 판정한다.
최근 머신러닝 기술을 녹내장 진단에 적용하고자 하는 시도가 활발히 이루어지고 있으나 대부분 단일 이미지 데이터나 EMR 과 같은 단일 형태의 싱글모달 자료를 학습하여 진단 모델을 만드는 연구가 주류를 이루고 있다[7]-[12]. 그러나 보다 신뢰성있는 진단모델을 만들기 위해서는 인간 의사와 같이 다양한 자료를 종합하여 녹내장에 대한 진단을 내릴 수 있는 멀티모달 진단 모델의 개발이 요청되나 소수의 연구만이 진행된 실정이다[13][14].
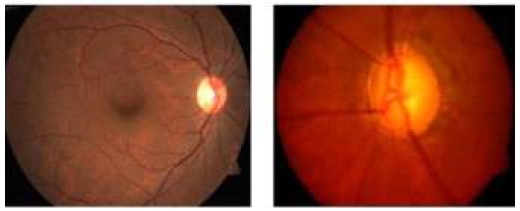
멀티모달 학습에서는 이질적 유형의 테이터를 통합 학습하기 위해 여러 가지 전략을 사용하는데, 녹내장 진단에 적합한 멀티모달 학습 방법의 개발이 요청되고 있다. 본 연구에서는 녹내장 조기 진단의 정확도와 신뢰도를 향상시키기 위해 이미지 데이터인 안저사진과 정형 수치 데이터인 EMR 자료를 통합하여 진단하는 멀티모달 진단 모델을 제안한다. 안저사진은 그림 1과 같이 안저 촬영을 통해 얻은 눈의 망막, 시신경, 망막 주변 혈관의 이미지이다.
안저사진을 통해 망막 질환, 혈관 이상, 시신경유두(Optic disc)의 상태 등을 평가함으로써, 눈의 건강 상태와 다양한 질병의 존재를 진단하는데 도움이 된다[15].
EMR은 녹내장 진단과 관련된 다양한 검사 결과 수치(나이, 시력, 혈압, 몸무게, 안압 등)들을 테이블 형태로 저장하고 있으며 환자의 상태를 파악하는 기초 자료로 활용된다.
Ⅱ. 제안 모델과 실험 방법
2.1 데이터셋
본 연구는 2012년 1월부터 2020년 11월까지 경상국립대학교(GNU) 병원에서 수집된 녹내장 환자의 데이터를 활용하여 실험을 수행하였다. 데이터셋은 훈련 데이터(Train data)와 테스트 데이터(Test data)로 나뉘며 5-Fold Cross Validation 기법으로 검증하였다(표 1).
2.2 안저사진의 전처리
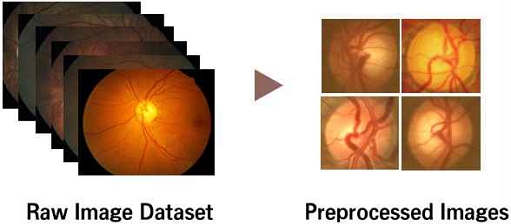
먼저 환자들로부터 수집된 원본 안저사진 데이터셋을 그림 2와 같이 전처리하는 단계를 진행한다. 전처리 과정의 첫 단계로는 분석에 적합한 이미지를 원본 데이터셋에서 선별한다.
이어서 선별된 안저사진들은 시신경유두 영상 분할 과정을 거쳤다. 마지막으로 이미지의 밝기, 색상 등을 조절하는 작업을 수행한다.
이후, 합성곱 신경망(CNN, Convolutional Neural Network)을 이용하여 피처를 추출하고, GAP, FC layer 등을 포함한 피처 변환(Feature transformation) 과정을 통해 2차원 피처 벡터를 생성한다.
2.3 EMR 데이터 전처리
EMR 원본 데이터로부터 피처 선택과정을 거처 총 10개의 주요 피처를 선택하였고, 샘플 데이터는 표 2와 같다.
이어서 각 피처의 값을 0과 1 사이의 값으로 변환하기 위해, 최소-최대 정규화(Min-max normalization) 과정을 거친 후, 정형데이터를 이미지 형태로 변환한다.
본 연구에서 사용한 정형데이터의 이미지 변환 방법은 M. F. Aslan et al.(2021), Y. Celik et al.(2022)와 같은 연구들[16][17]에서 사용한 방법을 기반으로 한다. 먼저 각 데이터 피처를 나타내기 위해 224×224 해상도를 가진 RGB채널의 Grid 이미지를 생성한다. 그리고 생성된 이미지를 그림 3과 같이 데이터 피처의 수에 맞게 비대칭적인 방식으로 영역을 분할한다. 본 연구에서는 정형데이터의 각 피처를 이미지의 영역에 할당할 때, 피처의 중요도가 높을수록 영역이 넓고 인접 영역이 많은 곳에 배치하는 방법을 채택하였다. 그리고 Red, Green, Blue 등과 같이 서로 구별되는 색상들을 인접한 영역 간 색상의 중복을 피하면서 각 영역에 할당하여 피처들을 구분한다. 마지막으로, 0에서 1 사이로 정규화된 각 피처의 값에 따라 각 영역의 색상의 밝기 강도가 0에서 255 사이로 조정된다. 그림 4는 정형데이터를 이미지 형태로 변환한 예시를 보여준다.
이후, 변환된 이미지 데이터는 CNN 모델을 통해 피처 추출 과정을 거치며 GAP 등의 피처 변환 과정을 통해 2차원 피처 벡터를 생성한다.
2.4 데이터 통합 및 모델 구축
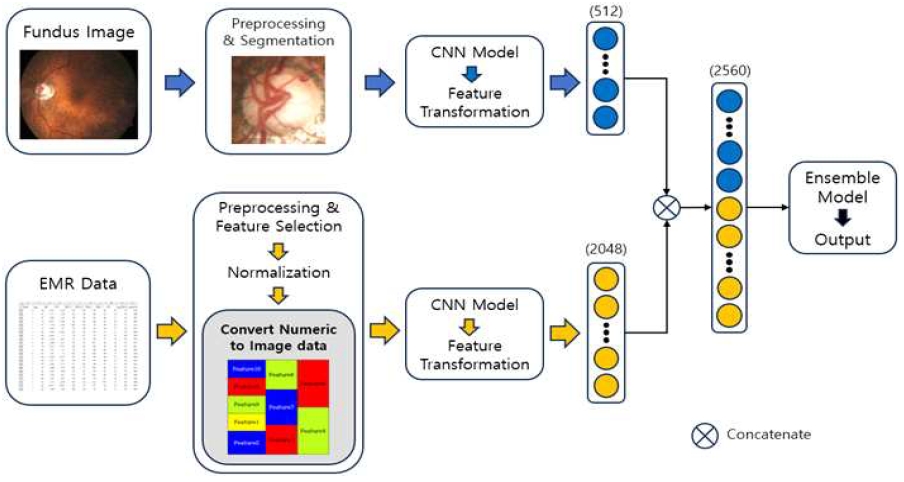
그림 5는 본 연구에서 제안하는 멀티모달 학습의 개요를 보여준다. 전처리 및 시신경유두 영역의 분할 과정을 거친 안저사진과 전처리 및 이미지 형태로 변환된 EMR 데이터는 각각 ResNet, VGG 등과 같은 많이 사용되는 CNN 모델을 통해 전이 학습을 활용하여 피처를 추출한다. 이후 GAP, FC layer 등의 피처 변환 과정을 통해 적절한 2차원 피처 벡터를 생성한다.
안저사진과 EMR 데이터에서 추출된 2차원 피처 벡터는 Concatenate 방법을 통해 하나의 통합된 피처 벡터로 결합한다. 결합된 피처 벡터는 XGBoost 앙상블 학습 모델을 사용하여 최종적인 녹내장 예측 모델을 구축하는 데 사용된다.
2.5 평가 지표
본 연구에서는 모델의 성능 평가를 위해 Accuracy, Precision, Recall, F1-score, AUC 및 Specificity와 같은 성능 지표들을 사용하였다.
| (1) |
| (2) |
| (3) |
| (4) |
| (5) |
2.6 싱글모달 비교 모델
본 연구의 멀티모달 모델과의 비교를 위해, 싱글모달 모델들에 대한 실험도 진행하였다. 안저사진을 기반으로 한 모델은 먼저 전처리 및 CNN 모델을 통해 피처 벡터로 변환된 후, GAP와 FC layer들을 통해 분류되었다. EMR 데이터를 기반으로 한 모델도 전처리 및 피처 선택 과정을 거친 후, 그래디언트 부스팅 방법인 XGBoost를 활용하여 최종 결과를 도출하였다. 표 3은 싱글모달 모델의 최고 결과를 보여준다.
표 3에 따르면, 안저사진 기반의 싱글모달 모델은 73.25%의 정확도를 보였고, EMR 데이터 기반의 싱글모달 모델은 92.10%의 정확도를 보였다. 이에 따라, 더 높은 정확도를 보인 EMR 데이터 기반의 싱글모달 모델이 제안하는 멀티모달 모델과 비교하는 데 사용되었다.
2.7 멀티모달 비교 모델
본 연구의 멀티모달 모델과의 비교를 위해, 멀티모달 녹내장 진단 모델인 P. Mehta et al.(2021)의 연구[13]와 W. S. Lim et al.(2022)의 연구[14]를 재현한 모델들에 대한 실험도 진행하였다. P. Mehta et al.(2021)의 연구에서는 황반 OCT 이미지 및 안저사진으로부터 추출된 피처를 인구통계학적 및 임상 데이터와 결합하여 그래디언트 부스팅 방법인 XGBoost를 통해 분석을 진행하였다. W. S. Lim et al.(2022)의 연구에서는 안저사진에서 회귀모델을 통해 예측한 RNFL 두께, C/D vertical ratio와 같은 피처와 Self-Attention을 적용한 안저사진에서 추출한 피처 및 검사 데이터를 결합 후, Random Forest, SVM 등과 같은 머신러닝 알고리즘으로 분석을 수행하였다. 본 연구에서는 각각의 연구에서 제안한 방식을 사용하였으나 부재한 데이터 제외, RNFL 두께 등의 일부 수치들은 제안한 방식처럼 직접 추출하거나 예측하지 않고 기존 OCT 검사에서 획득한 데이터를 그대로 사용하였다. 또한 머신러닝 알고리즘으로 모두 XGBoost 방법을 채택하였다.
Ⅲ. 실험 결과
3.1 제안모델의 성능 평가
제시된 실험 결과는 경상국립대학교 병원 데이터셋에 대해 제안된 멀티모달 모델의 성능을 비교 분석한 것이다. 제안한 방법은 안저사진과 EMR 데이터를 각각 알려진 CNN 모델로 학습한 뒤 이를 통합하는 것이다. 그런데 발표된 CNN 모델들이 다양하기 때문에 이들 모델들의 조합을 모두 테스트하기에는 시간적 제약이 따른다. 따라서 본 연구에서는 안저사진을 위한 CNN 모델은 성능이 높게 나온 ResNet50로 고정 사용하고, EMR 데이터를 위한 CNN 모델은 3개 모델(ResNet, VGG, DenseNet)의 여러 버전들을 테스트하여 어떤 모델이 우수한 성능을 보이는지 확인하였다. 표 4는 EMR의 CNN모델 중 성능이 높게 나온 DenseNet201, ResNet50, ResNet101에 따른 멀티모달 결과이다.
표 4에 따르면, DenseNet201이 93.31%의 가장 높은 정확도를 달성하여 제안 모델에서 채택하였다. 다시 말해서 안저사진을 위한 CNN 모델은 ResNet50을, EMR 데이터를 위한 CNN모델은 DenseNet201을 사용할 때 제안한 멀티모달 모델은 가장 높은 성능을 보였다. 그러나 보다 다양한 CNN 모델들의 조합을 테스트 한다면 더 높은 성능을 보이는 조합도 찾을 수 있을 것이다.
3.2 제안모델과 이전 모델의 성능 비교
본 절에서는 제안된 멀티모달 모델과 이전 연구에서 소개된 모델들을 경상국립대학교(GNU) 병원 데이터셋에 적용한 결과를 비교 분석한다.
표 5에 따르면, 제안된 멀티모달 모델은 싱글모달 비교 모델에 비해 모든 평가 지표에서 보다 우수한 성과를 보였으며, 정확도(ACC.)는 0.9353으로 싱글모달의 92.10에 비해 1.21% 향상되었다.
또한 제안된 모델은 멀티모달 비교모델인 P. Mehta와 W. S. Lim모델의 정확도 92.87에 비해 약 0.44% 더 향상되었다. 정밀도(Prec.)와 특이도(Spec.)에서는 다른 멀티모달 비교모델들에 비해 다소 낮은 성능을 보였지만, F1 Score와 AUC 등의 다른 성능 평가 지표들에서는 가장 높은 성능을 보였다.
Ⅳ. 결 론
본 연구에서는 녹내장 진단을 위해 안저사진과 EMR 데이터를 가지고 정형데이터를 이미지 데이터로 변환하는 기법을 활용한 새로운 멀티모달 러닝 분류 모델을 제안하였다. 제안한 방법은 싱글모달 방식에 비해 정확도뿐만 아니라 다른 모든 성능 평가 지표에서 우위의 성능을 나타내며, 다른 멀티모달 비교 모델들과 비교했을 때도 일부 지표를 제외하고는 더 높은 성능을 보였다.
제안된 모델은 녹내장 진단을 위한 새로운 형태의 멀티모달 러닝 방법을 제시하였으며, EMR 정형 데이터를 이미지로 변환하는 방법을 녹내장 진단을 위해 처음 적용하였다는 점과, 이러한 시도가 기존 멀티모달 녹내장 진단 모델들 보다 다소나마 우수한 성능을 보였다는 점에서 그 의미가 있다. 그리고 다양한 CNN 모델들의 조합을 안저사진과 이미지로 변환된 EMR 데이터에 적용하여 테스트한다면 보다높은 성능 향상이 기대된다. 또한 본 연구에서 제안한 접근 방법은 일반화가 가능하므로 다른 질환의 진단 모델 개발에의 적용도 시도해볼 수 있다.
Acknowledgments
이 논문은 2023년도 정부(과학기술정보통신부)의 재원으로 정보통신기획평가원의 지원을 받아 수행된 연구임(RS-2023-00222191, 분산된 데이터에 대한 논리적 데이터 통합과 복합분석을 지원하는 데이터 패브릭 기술개발)
References
-
B. M.. Davis, L. Crawley, and M. F. Cordeiro, "Glaucoma: the retina and beyond", Acta neuropathologica, Vol. 132, pp. 807-826, Aug. 2016.
[https://doi.org/10.1007/s00401-016-1609-2]

- American Academy of Ophthalmology, "What Is Glaucoma? Symptoms, Causes, Diagnosis, Treatment", https://www.aao.org/eye-health/diseases/what-is-glaucoma, [accessed: Dec. 04, 2023]
- World Health Organization, "Blindness and vision impairment", https://www.who.int/news-room/fact-sheets/detail/blindness-and-visual-impairment, . [accessed: Dec. 10, 2023]
-
Y. Lin, et al., "The Global Burden of Glaucoma: Findings from the Global Burden of Disease 2019 Study and Predictions by Bayesian Age–Period–Cohort Analysis", Journal of Clinical Medicine, Vol. 12, No. 5, pp. 1828, Feb. 2023.
[https://doi.org/10.3390/jcm12051828]

-
J. Chua, et al., "Prevalence, risk factors, and visual features of undiagnosed glaucoma: the Singapore Epidemiology of Eye Diseases Study", JAMA ophthalmology, Vol. 133, No. 8, pp. 938-946, Aug. 2015.
[https://doi.org/10.1001/jamaophthalmol.2015.1478]

-
Eye Diseases Prevalence Research Group, "Prevalence of open-angle glaucoma among adults in the United States", Archives of ophthalmology, Vol. 122, No. 4, pp. 532-538, Apr. 2004.
[https://doi.org/10.1001/archopht.122.4.532]

-
M. Biarnés, et al., "Classifying glaucoma exclusively with OCT: comparison of three clustering algorithms derived from machine learning", Eye, Vol. 38, No. 5, pp. 841-846, Oct. 2024.
[https://doi.org/10.1038/s41433-023-02785-5]

-
C. Li, et al., "Assessing the external validity of machine learning-based detection of glaucoma", Scientific reports, Vol. 13, No. 1, pp. 558, Jan. 2023.
[https://doi.org/10.1038/s41598-023-27783-1]

-
M. Raju, K. P. Shanmugam, and C.-R. Shyu, "Application of Machine Learning Predictive Models for Early Detection of Glaucoma Using Real World Data", Applied Sciences, Vol. 13, No. 4, pp. 2445, Feb. 2023.
[https://doi.org/10.3390/app13042445]

-
R. Verma, L. Shrinivasan, and B. Hiremath, "Machine learning classifiers for detection of glaucoma", IAES International Journal of Artificial Intelligence, Vol. 12, No. 2, pp. 806, Jun. 2023.
[https://doi.org/10.11591/ijai.v12.i2.pp806-814]

- H. Kim, H. Yoon, and Y. Kim, "Extraction of Abnormal Object Using Object Classification of Brain Images for HCI-based Medical Information System", Journal of Korean Institute of Information Technology, Vol. 10, No. 1, pp. 217-232, Jan. 2012.
-
H. Kim, K. Kim, and J. Y. Kang, "A Study on Impact of Medical Facility Accessibility on the Elderly Population Growth: A Comparative Analysis using Regression Analysis and Machine Learning Techniques", Journal of Korean Institute of Information Technology, Vol. 20, No. 5, pp. 113-121, May 2022.
[https://doi.org/10.14801/jkiit.2022.20.5.113]

-
P. Mehta, et al., "Automated detection of glaucoma with interpretable machine learning using clinical data and multimodal retinal images", American Journal of Ophthalmology, Vol. 231, pp. 154-169, Nov. 2021.
[https://doi.org/10.1016/j.ajo.2021.04.021]

-
W. S. Lim, et al., "Use of multimodal dataset in AI for detecting glaucoma based on fundus photographs assessed with OCT: focus group study on high prevalence of myopia", BMC Medical Imaging, Vol. 22, No. 1, pp. 1-14, Nov. 2022.
[https://doi.org/10.1186/s12880-022-00933-z]

-
U. Raghavendra, et al., "Deep convolution neural network for accurate diagnosis of glaucoma using digital fundus images", Information Sciences, Vol. 441, pp. 41-49, May 2018.
[https://doi.org/10.1016/j.ins.2018.01.051]

-
M. F. Aslan, K. Sabanci, and A. Durdu, "A CNN-based novel solution for determining the survival status of heart failure patients with clinical record data: numeric to image", Biomedical Signal Processing and Control, Vol. 68, pp. 102716, Jul. 2021.
[https://doi.org/10.1016/j.bspc.2021.102716]

-
Y. Celik, et al., "Improving inertial sensor-based activity recognition in neurological populations", Sensors, Vol. 22, No. 24, pp. 9891, Dec. 2022.
[https://doi.org/10.3390/s22249891]


2022년 2월 : 단국대학교 소프트웨어학과(공학사)
2024년 2월 : 단국대학교 인공지능융합학과(공학석사)
관심분야 : 데이터 분석, 머신러닝, 디지털 헬스케어, 멀티모달 러닝

2002년 2월 : 경상대학교 의학과(의학사)
2014년 2월 : 경상대학교 의학과(의학박사)
2020년 4월 ~ 현재 : 경상국립대학교 의과대학 부교수
관심분야 : 머신러닝, 질환 진단 및 임상시험용 모델 개발

1989년 2월 : 서강대학교 컴퓨터학과(공학사)
1991년 2월 : 서강대학교 컴퓨터학과(공학석사)
2001년 8월 : 서강대학교 컴퓨터학과(공학박사)
2003년 9월 ~ 현재 : 단국대학교 소프트웨어학과 교수
관심분야 : 데이터 분석, 머신러닝, 디지털 헬스케어