약물-질병 이분 네트워크를 통한 약물 재창출
초록
지난 몇 년간 새로운 약물이 개발되는 비율은 매우 감소하였다. 이를 해결하려는 방법 중 약물 재창출이 주목받고 있다. 약물 재창출이란 기존에 검증된 약물의 새로운 적응증을 찾는 것으로 약물 개발의 시간, 비용적 문제를 해결할 수 있다. 본 연구에서는 새로운 약물-질병 관계를 예측하기 위하여 약물 관련 데이터와 질병 관련 데이터를 통해 약물 간 유사도, 질병 간 유사도를 측정하고, 약물-질병 이분 네트워크를 구축한다. 이분 네트워크에서 공통된 적응증을 갖는 약물들을 통해 새로운 약물-질병 관계를 구한다. AUC(Area Under the ROC Curve) 값을 측정하여 본 연구 방법이 기존의 GBA(Guilt By Association) 방법보다 더 좋은 결과를 얻는 것을 보였다. 또한 피셔의 정확성 검정을 통해 본 연구에서 예측한 후보 약물-질병 관계가 KEGG 데이터베이스에 통계적으로 유의하게 포함된 것을 확인하였다.
Abstract
The rate of developing new drugs has decreased significantly over the past few years. Drug repositioning is drawing attention as a way to solve this problem. Drug repositioning can solve the time and cost of drug development by finding new indications for previously proven drugs. In this study, drug-related data and disease-related data are used to measure drug-to-disease similarities, and to establish a drug-to-disease bifurcation network. New drug-disease relationships are sought through drugs with common indications in bipartite networks. Measuring AUC(Area Under the ROC Curve) values, this study method showed better results than GBA (Guilt By Association). Fischer's exact test confirmed that the predicted candidate drug-disease relationship in this study was statistically significant in the KEGG database.
Keywords:
drug repositioning, network biology, bipartite network, data mining, bioinformaticsⅠ. 서 론
신약 개발은 많은 비용과 시간이 드는 작업이지만, 성공률이 매우 낮다. 새로운 약을 개발하는데 평균적으로 13.9년이 걸리며 성공률은 2%로 낮은 수치를 보인다[1]. 이러한 신약 개발의 시간적, 비용적 문제를 해결하는 방법으로 약물 재창출(Drug Repositioning)이 있다. 약물 재창출이란 기존에 알려진 약물들의 새로운 적응증을 찾아 기존과 다른 용도로 사용되는 것이다[2]. 대표적인 사례로 실데나필과 미녹시딜 약물이 있다. 두 약물은 고혈압 치료제로 개발되었으나, 실데나필은 발기부전, 미녹시딜은 탈모에 대한 새로운 적응증을 발견하여 발기부전 치료제, 탈모 치료제로 쓰이고 있다[3].
많은 약물 재창출 연구 중 네트워크를 활용한 연구들이 주목받고 있다. 네트워크 기반의 연구들은 생체분자들 간의 상호 작용을 강조하고 네트워크 개념을 강조한다[4]. Chiang의 연구에서는 유사한 두 질병에 대하여, 하나의 질병을 치료하는 약물은 나머지 하나의 질병 또한 치료할 가능성이 크다는 GBA(Guilt By Association) 접근 방식을 사용하여 새로운 약물-질병 관계를 예측하였다[5]. Martínez의 연구는 약물, 질병, 단백질의 정보를 통합한 네트워크 기반의 DrugNet 방법으로 약물-질병의 우선순위를 구하였다[6]. Gottlieb의 연구에서는 약물 간 유사도, 질병 간 유사도를 통해 PREDICT 알고리즘으로 새로운 약물-질병 관계를 예측하였다[7]. Zeng은 9개의 다양한 네트워크의 정보를 통합하여 딥러닝 기반의 약물 재창출을 진행하였다[8]. 이러한 기존 연구들은 약물의 화학적, 생물학적 유사도만 고려하여 진행하였다. 하지만 화학적 구조의 유사도가 약물 작용의 유사성을 의미하는 것은 아니다[9]. 이전 연구에 따르면, 다른 구조를 가진 약물들이 유사한 작용을 하는 경우가 있다[10].
본 연구에서는 약물 관련 데이터와 질병 관련 데이터를 통하여 약물 간 유사도, 질병 간 유사도를 각각 계산하였다. 추가적으로 약물의 구조적 유사도가 낮지만 질병에 대한 작용이 유사한 경우를 고려하기 위하여 알려진 약물-질병 치료 관계를 활용하였다.
본 논문은 2절에선 연구에서 사용한 주요 데이터와 성능 평가를 위해 사용한 기법을 소개한다. 3절에서는 본 연구에서 사용한 데이터, 연구 방법과 비교 실험에 대해 기술한다. 4절에서는 실험 결과와 비교 실험과의 성능 평가를 기술하며, 5절에서는 향후 연구에 대해 기술한다.
Ⅱ. 관련 연구
2.1 ATC code
ATC(Anatomical Therapeutic Chemical) 코드는 WHO에서 의약품을 분류하기 위해 사용하는 코드이다. 약물을 화학적 특성, 작용 부위에 따라 5단계로 분류하여 7자리의 코드로 표현한다[11]. 1단계는 약이 적용되는 부위에 따라 14개의 그룹 중 하나가 부여된다. A의 경우 소화관 및 대사, B의 경우 혈액, 조혈기관에 적용되는 약물임을 의미한다. 2단계는 약효, 3단계는 약물학적 특성, 4단계는 약물의 화학적 특성, 5단계는 개별 성분명에 따라 부여한다. 약물의 작용부위에 따라 ATC 코드가 다르므로 한 약물이 여러 개의 ATC 코드를 가질 수 있다. 아스피린의 경우 혈전성 방지 목적일 때는 B01AC06, 진통 해열제의 목적으로 먹는 경우 N02BA01의 ATC 코드를 가진다. 이처럼 ATC 코드는 약물의 화학적, 약물학적 등의 특성을 나타내는 코드이므로 두 약물의 코드가 동일하면 같은 효능을 가졌다고 볼 수 있다.
본 연구에서는 ATC 코드가 겹칠수록 두 약물이 유사하다는 가정하에 DrugBank 데이터베이스에서 제공하는 ATC 코드 정보를 사용하여 약물 간 유사도를 구하였다. 계층적 구조를 가진 코드이므로 이를 고려하기 위해 B01AC06 = {B, B01, B01A, B01AC, B01AC06} 와 같이 코드를 계층별로 나누어 사용하였다.
2.2 GO
GO(Gene Ontology)는 유전자를 분자 기능, 세포 성분, 생물학적 과정과 관련하여 설명하는 것이다. 분자 기능의 경우 분자 수준에서의 촉매, 결합 활동을 설명하며 세포 성분은 세포 내 구조에서의 위치를 나타내며 생물학적 과정은 분자 기능에 의해 달성되는 생물학적 목표를 의미한다 [12]. 시토크롬c의 경우 분자 기능은 산화 환원 효소 활동, 세포 성분은 미토콘드리아, 생물학적 과정은 산화 인산화로 나타낸다.
본 연구에서는 질병의 생물학적 과정을 고려하기 위해 질병 관련 유전자의 GO를 사용하였다. 두 질병의 질병관련유전자 사이에 겹치는 GO가 많으면 그 두 질병이 유사하다는 가정 하에 자카드 유사도를 구하였다.
2.3 AUC
본 연구 결과의 성능을 측정하기 위해 AUC(Area Under the ROC Curve) 값을 사용하였다. AUC는 ROC 곡선 아래의 영역을 의미한다.
ROC(Receiver Operating Characteristic) 곡선은 이진 분류 문제에 대한 모델의 성능을 평가하기 위해 사용되는 그래프이다. ROC 곡선의 x축은 False인 데이터를 모델이 True라고 잘못 예측한 FPR(False Positive Rate) 이며 y축은 True인 데이터를 모델이 True라고 맞게 예측한 TPR(True Positive Rate) 이다. TPR이 1에 가까울수록, 곡선이 좌측 상단에 가까울수록 성능이 좋은 모델이다. AUC는 위에서 구한 ROC 곡선의 아래 면적으로 1에 가까운 값이 나올수록 성능이 좋은 모델임을 나타낸다.
Ⅲ. 연구 방법
3.1 실험 데이터
본 연구에서는 새로운 약물-질병 관계를 찾기 위해 약물, 질병과 관련된 데이터와 기존에 알려진 약물-질병 관계 데이터를 사용하였다.
약물 목록은 DrugBank 데이터베이스에서 DrugBank 아이디로 수집하였다. DrugBank는 약물, 약물 표적 유전자, 약물 상호 작용 정보, 화학 구조식 등 약물과 관련된 데이터를 제공하는 데이터베이스이다[13].
약물의 유사도를 측정하기 위해 본 연구에서는 약물의 화학적 구조, ATC 코드, 부작용 데이터를 사용하였다.
약물의 화학적 구조는 PubChem 과 DrugBank에서 수집하였다[14]. PubChem은 화학 구조, 물리적 특성, 생물학적 활동 및 독성 데이터 등 생물 의학 연구를 위한 정보를 제공하는 데이터베이스이다. 약물의 화학적 구조는 약물의 흡수, 분포와 같은 특성을 결정하며 약물 분자의 활성도에 영향을 미친다[15]. SMILES(Simplified Molecular Input Line Entry System) 형태의 1,878가지 약물의 화학적 구조를 수집하였다.
약물 ATC 코드는 DrugBank에서 3,149가지 약물의 ATC 코드를 수집하였다.
약물 부작용 데이터는 SIDER 데이터베이스에서 수집하였다. SIDER는 약물의 부작용에 대한 정보를 제공하는 데이터베이스이다[16]. 1,009개의 약물과 5,357개의 부작용으로 이루어진 107,878개의 약물-부작용 관계를 수집하였다.
질병 목록은 CTD(Comparative Toxicogenomics Database) 데이터베이스에서 MeSH 아이디로 수집하였다. CTD는 문헌 기반으로 화학 물질, 유전자, 질병 등 이들 간의 관계를 수동으로 수집한 데이터베이스이다[17].
질병의 유사도를 측정하기 위해 본 연구에서는 질병관련 유전자, GO, MeSH 데이터를 사용하였다.
질병관련 유전자는 DisGeNet에서 수집하였다. DisGeNet은 질병 관련 유전자 데이터, 질병-질병 연관 데이터 등을 제공하는 데이터베이스이다[18]. 7,665개의 질병에서 16,407개의 질병 관련 유전자를 수집하였다.
질병 MeSH 트리 구조 데이터는 National Library of Medicine에서 수집하였다[19]. MeSH 트리는 질병에 대한 정보들로 이루어진 계층적 구조를 가진다. 질병 중 유방암의 경우 C04.588.180 = {C04, C04.588, C04.588.180} 의 MeSH 트리 구조를 가진다. ATC 코드와 동일하게 계층적 구조를 가졌으므로 이를 고려하여 계층별 유사도를 구한 후 평균값을 최종 MeSH 트리 구조 유사도로 사용하였다. 2,195개의 질병에 대한 MeSH 트리 구조를 수집하였다.
R 패키지 ClusterProfiler에서 제공하는 enrichGO 함수를 통해 질병 관련 유전자의 GO 데이터를 수집하였다[20]. ClusterProfiler는 유전자 및 유전자의 기능을 분석할 수 있는 R 패키지이다. 3,348개의 질병에 대한 47,098개의 GO 데이터를 수집하였다.
본 연구에서는 네트워크 구축에 사용할 알려진 약물-질병 관계를 CTD에서 수집하였다. 2,573개의 약물과 2,288개의 질병으로 이루어진 25,894개의 약물-질병 관계를 수집하였다.
본 연구 방법을 검증하기 위해 KEGG MEDICUS 데이터베이스에서 약물-질병 관계를 수집하였다. KEGG는 분자 상호작용 경로, 질병, 약물, 유전자, 효소 등의 다양한 생물학적 데이터를 제공하는 데이터베이스이다[21]. 569개의 약물과 305개의 질병으로 이루어진 1,477개의 약물-질병 관계를 수집하였다.
3.2 연구 개요
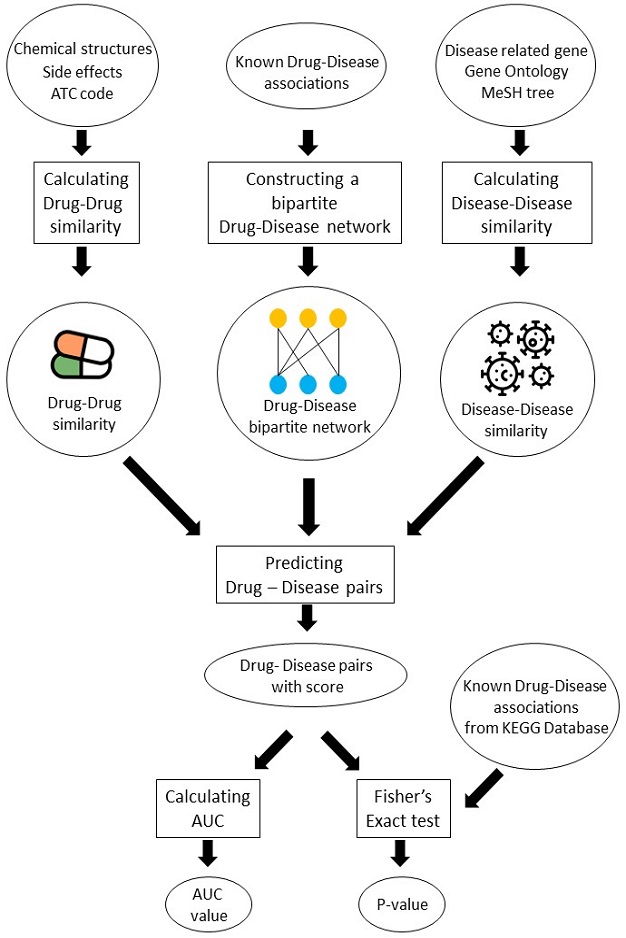
그림 1은 본 연구의 전체적인 개요를 나타낸다. 먼저, 약물의 화학적 구조, ATC 코드, 부작용 데이터를 사용하여 약물 간 유사도를 측정한다.
질병 또한 질병관련 유전자, GO, MeSH 용어 데이터를 사용하여 질병 간 유사도를 측정한다. CTD에서 얻은 약물-질병 관계를 통해 약물-질병 네트워크를 구축하고, 새로운 약물-질병 후보군을 추출한다. 앞에서 구한 약물 간 유사도, 질병 간 유사도를 통해 약물-질병 후보군의 점수를 부여한다. 후보군을 검증하기 위해 Fisher’s exact test를 진행하며, 성능 평가를 위해 AUC값을 측정하였다.
3.3 약물-약물, 질병-질병 유사도 측정
본 연구에서는 약물의 화학적 구조, ATC 코드, 부작용 데이터를 이용하여 약물 간 유사도를 측정하였다. ATC 코드, 부작용은 자카드 계수를 사용하여 유사도를 계산하였다. 자카드 계수의 공식은 식 (1) 과 같다.
| (1) |
∣A⋃B∣는 서로 다른 두 약물 A와 B가 가지고 있는 속성의 합집합의 크기이다. ∣A⋂B∣는 두 약물이 공통으로 가지고 있는 속성의 개수이다. 자카드 계수는 0과 1 사이의 값을 가지며 1에 가까울수록 두 약물이 유사하다는 것을 의미한다. 약물의 화학적 구조는 타니모토 계수를 사용하여 유사도를 계산하였다. 타니모토 계수는 자카드 계수와 같이 0과 1 사이의 값을 가지며 1에 가까울수록 두 약물이 유사하다는 것을 의미한다. 약물-약물 유사도를 구할 때 세 가지 속성에 대해 모두 값이 존재하는 약물만 사용하였다. 최종 약물 유사도 값은 세 가지 속성의 유사도 값들의 평균값을 사용하였다.
질병관련 유전자, GO, MeSH 트리 구조를 이용하여 질병 간 유사도를 구하였다. 약물 간 유사도와 동일하게 자카드 계수를 사용하여 유사도를 계산하였다. 질병-질병 유사도를 구할 때 약물과 동일하게 세 가지 속성에 대해 모두 값이 존재하는 질병만 사용하였다. 최종 질병 유사도 값은 세 가지 속성의 유사도 값들의 평균값을 사용하였다.
3.4 약물-질병 관계 예측
CTD에서 얻은 알려진 약물-질병 관계를 이용하여 이분 약물-질병 네트워크를 구축한다. 같은 그룹에 속한 노드들 사이에는 간선이 존재하지 않으며 약물 노드와 질병 노드를 연결하는 간선만 존재한다. 질병과 관계가 없는 약물 노드는 제외하고 실험을 진행하였다.
약물의 작용을 고려하여 새로운 약물-질병 관계를 도출하기 위해, 공통된 적응증을 가진 약물 쌍을 추출한다. 추출된 약물 쌍을 구성하는 약물 각각의 고유한 적응증과 나머지 하나의 약물에 대하여 약물 재창출을 시도한다.
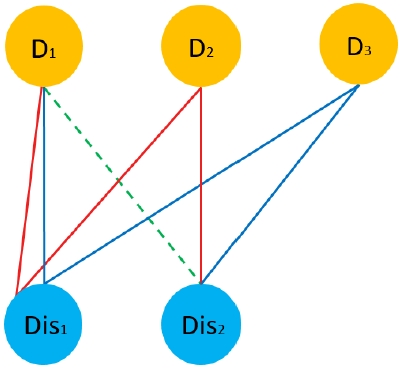
그림 2는 약물-질병 네트워크로, 실선은 CTD에서 수집한 약물-질병의 알려진 치료 관계이며 점선은 예측하고자하는 약물-질병 관계를 나타낸다. 약물 목록을 D, 질병 목록을 Dis라고 한다. 그림 2와 같이 공통된 적응증 Dis1을 가진 약물 쌍 D1과 D2에 대해, D2의 고유한 적응증인 Dis2와 약물 D1 사이의 관계를 약물 간, 질병 간 유사도를 기반으로 정량화한다.
| (2) |
약물 Di와 질병 Disj의 점수 계산식은 식 (2)와 같다. DRsim은 Di와 공통된 적응증을 갖는 약물 의 유사도이며, DISsim은 약물 쌍의 공통된 적응증과 Disj의 질병 간 유사도이다.
기존의 GBA 방법과 마찬가지로 약물-약물, 질병-질병의 유사도가 높을 경우 높은 점수를 부여하며, 유사도가 낮더라도 두 약물이 공통된 적응증을 많이 가질수록 높은 점수를 부여한다. 따라서 점수가 높을수록 해당 약물이 질병을 치료할 가능성이 높음을 의미한다. 그림 2의 경우 D1-Dis2의 점수는 D1-D2 유사도와 Dis1-Dis2 유사도를 곱한 값과 D1-D3 유사도와 Dis1-Dis2 유사도를 곱한 값의 합이다.
3.5 후보 약물-질병 관계 검증
위 방법을 통해 약물-질병 네트워크로부터 총 740,039개의 새로운 약물-질병 관계를 얻었다. CTD에서 얻은 약물-질병 관계를 통해 새롭게 얻은 약물-질병에 True와 False를 부여한다. 새롭게 얻은 관계가 CTD의 약물-질병 목록에 존재한다면 True, 존재하지 않는다면 False를 부여한다.
3.6 비교 실험
본 연구 방법의 성능을 평가하기 위해 비교 실험으로 GBA을 진행하였다.
GBA 방법은 두 약물이 유사하면 한 약물이 치료하는 질병을 다른 약물도 치료할 수 있다는 가정을 두어 실험하는 것이다. 반대로 두 질병이 유사하면 한 질병을 치료하는 약물이 다른 질병도 치료할 수 있다고 가정한다. D1이 Dis1을 치료한다고 알려져 있고 D1과 유사도가 가장 높은 약물이 D3이라면, D3과 Dis1을 새로운 약물-질병 관계로 정의한다. D3-Dis1의 점수는 D1-D3 의 유사도 값이다. 질병도 약물과 동일하게, Dis3이 D3을 치료한다고 알려져 있고 Dis3과 유사도가 가장 높은 질병이 Dis1이라면, D3과 Dis1을 새로운 약물-질병 관계로 정의하며 점수는 Dis1-Dis3의 유사도 값이다. 최종적인 약물-질병 관계 점수는 위에서 구해진 약물의 유사도에 질병 유사도를 곱한 값이다. 점수가 높을수록 해당 약물이 질병을 치료할 가능성이 크다는 것을 의미한다. 모든 약물, 질병에 대해 이 과정을 수행한다. 본 연구와 동일하게 약물과 질병은 세 가지 속성에 대해 모든 값이 존재하는 객체만 사용하였다.
위 방법을 통해 718,930개의 약물-질병 관계를 얻었다. 본 연구와 동일하게 CTD에 해당 약물-질병 관계가 존재하면 True, 존재하지 않으면 False를 부여한다.
Ⅳ. 결 과
4.1 성능 평가
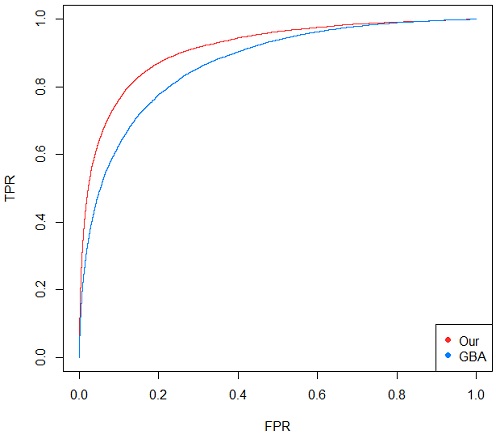
본 연구 방법의 성능을 평가하기 위해, GBA방법과 본 연구의 AUC 값을 구하였다. GBA는 두 약물이 유사하면 치료하는 질병을 공유할 것이라고 가정하는 방법이다. 본 연구에서 구한 약물-질병 관계에서 True는 11,974개 False는 727,565개이며 GBA는 True가 10,383개 False가 708,047개로 이 결과를 통해 그림 3과 같은 ROC곡선을 그렸다. x축은 FPR, y축은 TPR이며 빨간색은 본 연구방법, 파란색은 GBA의 ROC 곡선이다.
표 1은 ROC곡선을 통해 얻은 본 연구 방법과 GBA의 AUC 값으로, 본 연구의 AUC가 GBA보다 0.0448 더 높은 결과를 얻었다.
AUC 값이 1에 가까울수록, ROC 곡선이 좌측 상단에 가까울수록 성능이 좋다는 것이므로 본 연구의 방법이 GBA보다 성능이 좋다는 것을 볼 수 있다. 결론적으로, 질병을 공유하는 약물은 또 다른 질병을 공유할 것이라는 본 연구의 방법이 공유하는 질병을 고려하지 않은 GBA방법보다 약물-질병 관계를 예측을 적절하게 수행하는 것을 보인다.
4.2 후보 약물-질병 관계 검증
본 연구 방법에서 얻은 후보 약물-질병 관계를 검증하기 위해, KEGG MEDICUS에서 수집한 약물-질병 관계를 통해 fisher’s exact test를 진행하였다. 본 연구의 기준값(Cut-off Value)을 구하기 위해, ROC 곡선에서 Youden’s 지수를 구하고, 이 값을 기준으로 약물-질병 관계 점수가 지수 값 이상은 True, 미만은 False를 새로 부여하였다. Youden’s 지수 계산법은 식 (3)과 같다[22]. 검증 실험 결과, p-value < 2.2e-16의 값으로 0.05 이하의 값을 갖는다. 따라서 본 연구 방법이 통계적으로 유의미하게 후보 약물-질병 관계를 추출함을 보인다.
| (3) |
표 2는 본 연구에서 예측한 상위 5개의 약물-질병 목록이다. Paclitaxel, Cyclophosphamide 두 약물과 유방암 간의 관계는 KEGG 데이터베이스에 존재하는 것을 확인하였다. 또한 Doxorubicin 약물이 유방암과 전립선암을 치료한다는 연구가 진행되었으며[23][24] 이외에도 많은 종양에 대한 치료제로 사용된다[25]. 이를 통해 본 연구의 방법이 새로운 약물-질병 관계를 예측하는 데 효과적임을 보인다.
Ⅴ. 결론 및 향후 과제
본 연구에서는 약물의 화학적 구조, ATC 코드, 부작용 데이터를 통해 약물 간 유사도, 질병 관련 유전자, MeSH 용어, GO를 통해 질병 간 유사도를 구하였다. CTD에서 얻은 약물-질병 관계를 통해 약물-질병 네트워크를 구축하여 새로운 약물-질병 관계를 얻었다. GBA의 방법과 AUC를 비교한 결과, 본 연구의 방법의 AUC가 더 높은 결과를 보였다. 또한 Fisher’s exact test를 통해 후보 약물-질병 관계가 유의미한 관계임을 확인하였다.
향후 연구에서는 다른 데이터베이스에서 연구에 사용한 약물 관련 데이터, 질병 관련 데이터를 추가적으로 수집하여 연구에 사용할 수 있는 약물, 질병 개수를 늘려 더 정확한 약물-약물 유사도, 질병-질병 유사도를 구해 약물-질병 점수를 계산할 예정이다. 또한 약물의 표적 유전자, 효소, 수용체와 같은 약물 관련 데이터를 추가하여 다양한 특성을 고려하여 실험을 진행할 예정이다.
Acknowledgments
이 논문은 2018년도 정부(미래창조과학부)의 재원으로 한국연구재단(No.2018R1A2B6006223)의 지원을 받아 수행된 연구임. (NRF-2018R1A2B6006223)
References
-
Y. Yeu, Y. Yoon, and S. Park, "Protein localization vector propagation: a method for improving the accuracy of drug repositioning", Molecular bioSystems, Vol. 11, No. 7, pp. 2096-2102, Jul. 2015.
[https://doi.org/10.1039/C5MB00306G]

-
S. Shin, Y. Sin, G. Jang, and Y. Yoon, "Co-occurrence Based Drug-disease Relationship Inference with Genes as Mediators", The Journal of Korean Institute of Information Technology, Vol. 16, No. 11, pp. 1-9, Nov. 2018.
[https://doi.org/10.14801/jkiit.2018.16.11.1]

-
Y. Y. Li and S. J. Jones, "Drug repositioning for personalized medicine", Genome Med, Vol. 4, p. 27, Mar. 2012.
[https://doi.org/10.1186/gm326]

-
Z. Wu, Y. Wang, and L. Chen, "Network-based drug repositioning", Mol. BioSyst, No. 9, pp. 1268-1281, Sep. 2013.
[https://doi.org/10.1039/c3mb25382a]

-
Chiang AP and Butte AJ, "Systematic evaluation of drug-disease relationships to identify leads for novel drug uses", Clinical pharmacology and therapeutics, Vol. 86, No. 5, pp. 507-510, Nov. 2009.
[https://doi.org/10.1038/clpt.2009.103]

-
V. Martínez, C. Navarro, C. Cano, and W. Fajardo and A. Blanco, "DrugNet: Network-based drug–disease prioritization by integrating heterogeneous data", Artif Intell Med, Vol. 63, No. 1, pp. 41–49, Jan. 2015.
[https://doi.org/10.1016/j.artmed.2014.11.003]

-
A. Gottlieb, G. Y. Stein, E. Ruppin, and R. Sharan, "PREDICT: a method for inferring novel drug indications with application to personalized medicine", Mol Syst Biol, Vol. 7, No. 1, p. 496, Jun. 2011.
[https://doi.org/10.1038/msb.2011.26]

-
Xiangxiang Zeng, Siyi Zhu, Xiangrong Liu, Yadi Zhou, Ruth Nussinov, and Feixiong Cheng, "deepDR: a network-based deep learning approach to in silico drug repositioning", Bioinformatics, Vol. 35, No. 24, pp. 5191-5198, Dec. 2019.
[https://doi.org/10.1093/bioinformatics/btz418]

-
N. Nikolova and J. Jaworska, "Approaches to measure chemical similarity – a review", QSAR Comb, Vol. 22, No. 9-10, pp. 1006-1026, Jan. 2004.
[https://doi.org/10.1002/qsar.200330831]

-
Y. Hu and J. Bajorath, "Many structurally related drugs bind different targets whereas distinct drugs display significant target overlap", RSC Adv, Vol. 2, pp. 3481-3489, Jan. 2012.
[https://doi.org/10.1039/c2ra01345b]

- WHO Collaborating Centre for Drug Statistics Methodology, https://www.whocc.no, [accessed: Aug. 28. 2020]
-
Gene Ontology Consortium, "The Gene Ontology (GO) database and informatics resource", Nucleic Acids Research, Vol. 32, No. suppl_1, 1, pp. D258–D261, Jan. 2004.
[https://doi.org/10.1093/nar/gkh036]

-
D. S. Wishart, Y. D. Feunang, A. C. Guo, E. J. Lo, A. Marcu, J. R. Grant, T. Sajed, D. Johnson, C. Li, and Z. Sayeeda et al, "DrugBank 5.0: a major update to the DrugBank database for 2018", Nucleic Acids Research, Vol. 46, No. D1, pp. D1074–D1082, Jan. 2018.
[https://doi.org/10.1093/nar/gkx1037]

-
S. Kim, J. Chen, T. Cheng, A. Gindulyte, J. He, S. He, Q. Li, B. A. Shoemaker, P. A. Thiessen, and B. Yu et al, "PubChem 2019 update: improved access to chemical data", Nucleic Acids Research, Vol. 47, No. D1, pp. D1102–D1109, Jan. 2019.
[https://doi.org/10.1093/nar/gky1033]

-
F. Mao, W. Ni, X. Xu, H. Wang, J. Wang, M. Ji, and J. Li, "Chemical Structure-Related Drug-Like Criteria of Global Approved Drugs", Molecules, Vol. 21, No. 1:75, Jan. 2016.
[https://doi.org/10.3390/molecules21010075]

-
Michael Kuhn, Ivica Letunic, Lars Juhl Jensen, and Peer Bork, "The SIDER database of drugs and side effects", Nucleic Acids Res, Vol. 44, No. D1, pp. D1075-1079, Jan. 2016.
[https://doi.org/10.1093/nar/gkv1075]

-
Allan Peter Davis, Cynthia J Grondin, Robin J Johnson, Daniela Sciaky, Roy McMorran, Jolene Wiegers, Thomas C Wiegers, and Carolyn J Mattingly, "The Comparative Toxicogenomics Database: update 2019”, Nucleic Acids Research, Vol. 47, No. D1, pp. D948–D954, Jan. 2019.
[https://doi.org/10.1093/nar/gky868]

- Janet Piñero, Juan Manuel Ramírez-Anguita, Josep Saüch-Pitarch, Francesco Ronzano, Emilio Centeno, Ferran Sanz, and Laura I Furlong, "The DisGeNET knowledge platform for disease genomics: 2019 update", Nucleic Acids Research, Vol. 48, No. D1, pp. D845–D855, Jan, 2020.
- C. E. Lipscomb, "Medical Subject Headings (MeSH)", Bull Med Libr Assoc, Vol. 88, No. 3, pp. 265-266, Jul. 2000.
-
G. Yu, L. G. Wang, Y. Han, and Q. Y. He, "clusterProfiler: an R package for comparing biological themes among gene clusters", OMICS: A Journal of Integrative Biology, Vol. 16, No. 5, pp. 284-287, May 2012.
[https://doi.org/10.1089/omi.2011.0118]

-
Minoru Kanehisa and Susumu Goto, "KEGG: Kyoto Encyclopedia of Genes and Genomes", Nucleic Acids Research, Vol. 28, No. 1, pp. 27–30, Jan. 2000.
[https://doi.org/10.1093/nar/28.1.27]

-
R. Fluss, D. Faraggi, and B. Reiser, "Estimation of the Youden Index and its associated cutoff point", Biom J., Vol. 47, No. 4, pp. 458–472, Mar. 2005.
[https://doi.org/10.1002/bimj.200410135]

-
Z. Ma, L. Xu, D. Liu, X. Zhang, S. Di, W. Li, and et al, "Utilizing Melatonin to Alleviate Side Effects of Chemotherapy: A Potentially Good Partner for Treating Cancer with Ageing", Oxidative Medicine and Cellular Longevity, Vol. 2020:6841581, May 2020.
[https://doi.org/10.1155/2020/6841581]

-
X. Fan, L. Wang, Y. Guo, X. Xiong, L. Zhu, and K. Fang, "Inhibition of prostate cancer growth using doxorubicin assisted by ultrasound-targeted nanobubble destruction", Int J Nanomedicine, Vol. 2016:11, pp. 3585-3596, Jul. 2016.
[https://doi.org/10.2147/IJN.S111808]

-
S. Eikenberry, "A tumor cord model for doxorubicin delivery and dose optimization in solid tumors", Theoretical biology & medical modelling, Vol. 6, p. 16, Aug. 2009.
[https://doi.org/10.1186/1742-4682-6-16]


2017년 3월 ~ 현재 : 가천대학교 컴퓨터공학과 재학
관심분야 : 데이터마이닝, 네트워크 바이올로지, 바이오인포매틱스

1981년 : 서울대학교 자연과학대학 (학사)
1983년 : 오하이오 주립대학 수학과(학사 수료)
1987년 : 스탠포드대학교 컴퓨터 과학과 졸업(이학석사)
2008년 : 연세대학교 컴퓨터과학과 졸업(공학박사)
1987년 5월 ~ 1993년 5월 : IntelliGenetics Inc., California, USA, Software Engineer
1995년 2월 ~ 현재 : 가천대학교 컴퓨터공학과 교수
관심분야 : 데이터베이스 시스템, 데이터 마이닝, 바이오인포매틱스, 소셜미디어 데이터 마이닝