초분광 영상과 심층 신경망을 이용한 작물 생육상태 분류 기법 : 파프리카 잎 사례 연구
초록
최근 들어 초분광 영상을 이용하여 식물의 생육상태 분석 연구가 이루어지고 있다. 하지만 물리적 요인과 데이터의 복잡성 등 많은 요인이 초분광 영상 분석을 어렵게 한다. 본 논문은 초분광 영상과 DNN을 이용하여 작물의 생육상태 분류 기법을 제안한다. 초분광 영상을 통해 작물의 주요 정보를 얻고 DNN이 효과적으로 동작할 수 있도록 작은 패치 크기로 데이터를 자르고, 회전시키는 등 데이터 전처리 기법을 사용한다. 실험에서는 파프리카 잎을 정상 잎, 해충 피해를 입은 잎 등 4종류의 잎과 배경으로 분류하였으며, 실험 결과 90.9%의 정확도를 보였다. 제안된 기법은 데이터 생성 방법이 DNN에 부담을 주지 않으며, 기존의 RGB 영상으로는 판단하기 어려운 다양한 생육 상태를 분류할 수 있다.
Abstract
Recently, the analysis research of plant's growth condition is done with the use of hyperspectral image. However, there are many factors such as physical factors and complexity of data make the hyperspectral image analysis difficult. This study presents the classification technique of plant's growth condition using hyperspectral image and DNN(Deep Neural Network). Major information of plants is acquired through hyperspectral image, and the preprocessing is followed for the information to be used for DNN learning. The preprocessing is used by cutting the data in small patch size and rotating it for the models to be operated effectively. In the experiment, paprika leaves are divided into four types of leaves and backgrounds such as normal and damaged by harmful insects, and the result of the experiment showed 90.9% of accuracy. The presented technique has advantages that the data generation method does not affect DNN and can classify various growth conditions that are difficult in the existing RGB image.
Keywords:
hyperspectral images, deep neural network, growth condition, image classification, paprika, disease/pestⅠ. 서 론
작물의 생산량에 직간접적으로 영향을 미치는 많은 요인들 중 건강 상태를 모니터링하고 병해충을 탐지하는 것은 매우 중요하다. 이는 질병으로 인한 피해를 줄이고 성장을 효과적으로 촉진시킬 수 있어 작물을 관리하고 개선하는데 매우 유용하다[1].
일반적으로 병해충 진단 방법은 파괴 및 비파괴 분석 방법으로 분류할 수 있다. 파괴 분석 방법은 샘플을 화학적으로 분석하는 기술로 분석을 위한 시간과 비용이 많이 소모되며 무엇보다 작물이 파괴되는 단점이 있다[2]. 이러한 이유로 최근에는 비파괴 분석법을 적용하여 생육 특성을 측정하고 분석한다[3]. 비파괴 분석법은 재료가 갖는 물리적인 성질을 이용하여 시험 대상물을 손상시키거나, 파괴하지 않고 내외부의 상태를 파악하는 방법으로 일반적으로 분광, 형광, 열화상, 초분광 등의 스펙트럼 기반 영상이 작물 병해충 탐지에 가장 많이 사용되는 비파괴 분석법 중 하나이다[4]. 특히 최근에는 초분광 영상(Hyperspectral Imaging, HSI)을 이용하여 식물의 생육상태 분석, 병해충 탐지, 모델링 등 다양하게 활용되고 있다[3]-[6].
식물이 다양한 원인에 의해 스트레스를 받으면 잎 엽록소 함량이 감소하거나, 잎 세포 구조가 변하는 등의 생물리학적인 변화를 겪는다. 초분광 영상은 식물의 스펙트럼 반사율에서 이런 미묘한 변화를 감지하는 이점이 있다. 하지만 높은 공간 해상도와 스펙트럼 정보로 인한 물리학적 요인과 데이터의 중복, 차원의 저주 등과 같은 다양한 요인이 초분광 영상의 분석을 어렵게 한다[7].
최근 심층 신경망(Deep Neural Network, DNN)이 활발히 연구되면서 자동으로 특징을 추출하고 효과적으로 학습할 수 있는 지도학습 분류기를 구성할 수 있게 되었다[8]. DNN이 효과적으로 분류하기 위해서는 데이터의 양이 중요하다. 특히 지도학습 딥러닝은 과잉적합 문제를 피하기 위해 많은 레이블링된 데이터가 필요하다[9].
우리나라 수출 농산물의 주요 작물인 파프리카는 주로 칼슘과 붕소 흡수가 저해되어 잎과 과실의 끝이 검게 썩는 잎마름병과, 해충에 의해 잎 표면에 작고 흰 반점이 나타나 피해가 심하면 잎이 말라 죽는 질병 등이 있다[10]-[12]. 결국 질병의 증상은 파프리카 잎에 주로 나타나며 이를 조기에 발견하는 것은 파프리카가 잘 성장하는데 매우 중요하다.
본 논문에서는 초분광 영상과 DNN을 이용하여 작물의 생육상태 분류기법을 제시하고자 한다. 생육 상태와 병해충을 검출하기 위해 주요 질병 발생 위치인 잎을 초분광 카메라로 데이터를 취득한다. 그리고 데이터의 양을 늘려 DNN이 효과적으로 동작하도록 다양한 데이터 전처리 기법을 사용한다. 실험에서는 파프리카 잎을 정상 잎, 해충 피해를 입은 잎 등 4종류와 배경으로 분류하였으며, 실험 결과 90.9%의 정확도를 보였다.
논문의 나머지 부분은 다음과 같이 구성되어 있다. 2장에서는 작물 생육상태 진단에 적용되는 초분광 영상 및 기계학습 기술을 기술한다. 3장은 본 논문에서 제안하는 작물 생육상태 분류 기법을 제시하며, 데이터 전처리 방법과 DNN 모델에 대해 설명한다. 4장에서는 실험을 통한 파프리카 생육상태 분류 기법의 효과를 보여주며, 마지막으로 5장에서 논문을 마치고 향후 연구에 대해 논의한다.
Ⅱ. 관련 연구
2.1 초분광 영상 기반 생육상태 관련 연구
엽면적 지수(LAI, Leaf area index)는 한 군락의 총 엽면적을 그 군락의 재배 면적으로 나눈 값으로 식물의 광합성 생산과 증발산 등 식물의 성장을 평가하기 위한 중요 지표 중 하나이다.
Liang[6]등은 LAI 값을 추정하기 위해 하이브리드 역전 기법 연구를 수행하였다. 초분광 시뮬레이션 데이터 세트를 기반으로 43개의 식생지수(VI, Vegetation Indices)를 분석하여 LAI 값을 추정하기 위한 최적의 VI를 식별하였다. 하지만 VI 지수는 건강한 작물과 병해충에 걸린 작물을 분류하도록 설계되지 않았다[13].
Ke[14]등은 초분광 영상에 기반을 둔 LAI 추정 접근법을 통계 모델 접근법, 물리적인 모델(캐노피 반사 모델), 하이브리드 역전 모델의 3가지 유형으로 분류하였다. 마찬가지로, LAI는 단위 면적당 엽면적의 분포를 확인할 수 있는 지표로서 잎의 병해충을 조기에 판단하기에는 무리가 있다.
다른 연구로는 초분광 영상으로 정보를 추출하기 위해 유전 알고리즘을 사용하여 GP-SVI(Genetic Programming-Spectral Vegetation Index)를 제안하였다[15]. 이 기법은 작물의 생물리학적 변수를 설명하는 회귀 모델을 옥수수의 캐노피 질소 함량과 스펙트럼 밴드 반사율을 상관시켜 캐노피 질소 변동에 대하여 농부가 비료 공급을 조절할 수 있도록 유도하였다. 이를 통해 토양의 지속적인 관리와 경제적으로 절약할 수 있는 이점을 제공하였다.
2.2 농업 분야의 기계학습 활용 연구
농업 분야는 개인정보가 없으며 활용도가 매우 넓어 예전부터 기계 학습을 적용한 연구가 활발하게 이루어지고 있다. 다양한 연구자들은 수확량에 대한 가상 조건과 작물의 환경·생장 데이터를 기반으로 작물 생장 모델을 제안하였다[16].
Bejo[17]등은 지능형 시스템을 농업 연구의 복잡한 문제에 대해 해결 방법을 제시하고자 가장 일반적인 ANN(Artificial Neural Network) 유형을 제안하였다. 농업분야의 ANN 아키텍처의 기본 원칙, 입력 매개 변수로 다양한 유형의 작물 생장 데이터를 사용하여 수확량 예측을 위한 ANN 적용 방법 등 현재 추세에 대해 제시하였다. 또한 Bose[18]등은 작물의 수확량은 파종부터 수확까지 긴 시간동안 다양한 변수들이 영향을 미치므로, 생물리학적 변수를 통한 시계열 정보로부터 작물 생산량을 추정을 위한 SNN(Spiking Neural Network) 계산 모델을 제안하였다.
Xia[19]등은 병렬 컴퓨팅을 활용하여 두 가지 유채꽃의 종자를 세 가지 다른 물줄기에 노출 시킨 후 이들의 고사에 대하여 초분광 영상을 취득하였다. 그리고 데이터 전처리 과정을 거쳐 ANN과 SVM(Support Vector Machine) 알고리즘으로 유채 종자 수분 탐지 모델을 제안하였다. 이는 작물과 수분간의 상관관계를 분석하고 실제 생산 시나리오에 적용할 수 있는 방법을 제공하였다.
2.3 초분광 영상 기반 작물 병해충 판단 연구
SVM은 작물 병해충 탐지를 위해 자주 사용되는 기계 학습 알고리즘이다. Rumpf[20]등은 초분광 영상으로 작물의 질병을 조기에 발견하기 위해 SVM을 사용하였다. 그들은 25가지 VI를 이용하여 초분광 영상에서 일부분의 스펙트럼 대역을 이용하여 질병 분류 모델을 보여주었다. 하지만 초분광 영상은 일반적으로 100개 이상의 스팩트럼 밴드를 구성하고 있어 이를 모두 SVM 알고리즘으로 학습시키기에는 무리가 있다.
Zhu[21]등은 초분광 영상으로 담배 잎의 질병 조기 검출과 분류 기법을 연구하였다. 그들은 VNIR (400-1000nm)대역을 사용하며 모든 픽셀이 아닌 ROI(Region Of Interest)로부터 평균 스펙트럼을 생성하였다. 또한 대부분의 문헌은 VI[20], PCA[21] 및 수동 파장 선택[22]과 같은 접근법을 통해 초분광 영상에서 특징을 추출하였다.
질병을 탐지하기 위해 기계학습을 사용한 여러 연구가 있었지만 초분광 영상이 아닌 RGB 이미지만 사용한 연구도 있었다[23]-[25]. 그리고 딥러닝을 이용한 파프리카의 병해충 자동 검출 기법[26]은 기존에 연구 되었으나 RGB 이미지만 사용하였고, 초분광 영상을 사용한 파프리카 병해충 검출 기법에 대한 연구는 아직 수행되지 않았다. 그 이유는 주로 초분광 영상은 촬영하기 쉽지 않고 사용하는 센서도 일반 카메라 렌즈보다 훨씬 비싸기 때문이다. 일반적으로 딥러닝을 수행하기 위해서는 막대한 양의 데이터가 필요하다. 따라서 초분광 영상을 이용한 딥러닝 분류 모델을 만들기 위해서는 제한된 양의 초분광 영상을 효율적으로 사용해야 한다.
본 논문에서는 딥러닝 모델이 효율적으로 작동하고 과적합 문제를 피하기 위해 초분광 영상을 다양한 기법에 적용시켜 변형 및 가공하고 사용할 수 있도록 제안된다.
Ⅲ. 작물 생육상태 분류 기법
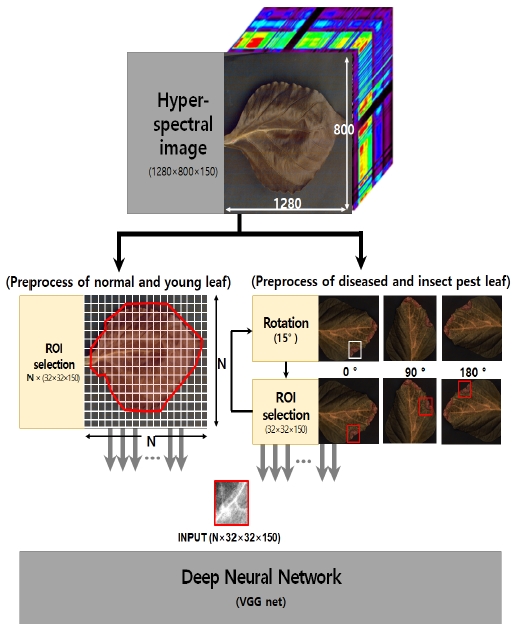
이번 장에서는 초분광 영상과 DNN을 이용한 작물 생육상태 분류 기법을 제안한다. 전체 기법의 전반적인 절차는 그림 1과 같으며, 초분광 영상을 취득하는 단계부터 초분광 영상 전처리, DNN 학습을 위한 데이터 전처리, 그리고 분류 모델인 DNN 구조에 대해 기술한다.
작물 생육상태는 5가지 조건으로 정상 잎, 어린 잎, 질병에 걸린 잎, 해충에 피해를 입은 잎 그리고 배경을 포함한다. 여기서 어린 잎은 작물 상단의 잎이 생긴지 20일 이전이며 그 이후의 잎을 정상 잎으로 분류한다.
3.1 초분광 영상 데이터 취득 단계
2019년 9월 동안 전북 군산시에 위치한 비닐온실에서 파프리카 잎의 초분광 영상을 촬영하였다. 초분광 카메라 시스템은 Snapscan 방식의 센서가 내장된 그림 2의 Snapscan VNIR(IMEC Inc.)를 사용하였고, 할로겐 램프(FOMEX H1000, Halogen 650W, 2Set)가 설치된 실내에서 데이터를 획득하였다.
초분광 영상 촬영 시 조명은 매우 중요한 요소로 조명에 따라 각각의 파장에서 다른 강도의 레벨을 갖는다. 이는 영상 시스템의 전체 스펙트럼 응답에 크게 영향을 미친다.
따라서 응용 분야 요구 사항에 따라 시스템에 사용되는 조명의 스펙트럼 특성을 고려하는 것이 중요하다. 본 연구에서는 식물 잎의 생육상태를 위한 데이터 취득이 목적이므로, 일반적으로 식물 잎의 광합성 색소로 인한 빛 흡수 범위인 VIS(400-700nm)와 잎의 세포 구조와 밀접하게 연관된 NIR(700-1000nm)를 측정하기 위해 할로겐 조명을 사용하였다.
촬영은 잎의 수분 부족을 피하기 위해 5분 전에 수집된 잎에 대해서만 영상을 촬영하였다. 공간 해상도는 1280×800 pixel로 고정하였으며, 데이터는 32 bit/pixel의 디지털 형식으로 저장하였다. 따라서 초분광 영상의 전체 볼륨 크기는 1280(Width)×800(Height)×150(Band)×32(Bit) 이다.
3.2 초분광 영상 데이터 전처리 단계
초분광 카메라 시스템으로 측정한 객체는 아래와 같은 영향을 제거하기 위해 반사율 계산인 화이트 밸런싱을 진행한다.
• 광원의 방출 스펙트럼
• 조명으로 발생하는 스펙트럼 불균일성
• 빛이 통과하는 매체의 영향
• 렌즈 시스템의 오차율
• 렌즈 시스템으로 인한 스펙트럼의 변형
• 시스템 모델과 실제 현실과의 차이
• 센서 자체 특성 등
화이트 밸런싱은 정면에 흰색 확산 반사 대상체를 배치하여 수행한다. 대상체를 측정하고 측정된 값을 반사율 값으로 나누어 측정된 값을 100%로 확대하여 계산한다. 다음 식으로 하나의 흰색 참조 이미지와 두 개의 어두운 참조 이미지를 사용하여 반사율을 계산한다.
| (1) |
• objradi : 관심 객체의 광도
• white ref : 흰색 반사 대상체의 복사량
• dark ref (timeobj) : 관심 객체의 timeobj로 찍은 어두운 참조 이미지
• dark ref (timewhite ref) : 흰색 반사 대상체의 timewhite ref로 찍은 어두운 참조 이미지
• timewhite ref : 흰색 참조 이미지 캡처 시간
• timeobj : 객체 이미지 캡처 시간
3.3 DNN의 입력을 위한 데이터 전처리 단계
잎 상태에 따른 분류를 위해 DNN의 한 종류인 VGG-16의 변형[27]이 사용된다. 딥러닝 학습은 기본적으로 많은 양의 데이터가 필요하다는 것은 잘 알려져 있다. 하지만 초분광 영상은 촬영에 많은 시간이 소요되고, 장비 또한 고가이므로 데이터 세트가 부족할 수 있다. 따라서 본 논문에서는 표 1의 생육 상태에 따른 원본 이미지를 바로 학습 데이터로 사용하지 않고, 그림 3의 데이터 전처리 과정을 거쳐 작은 패치 사이즈로 잘라내어 학습에 필요한 데이터를 확보하였다.
패치를 분류하여 훈련 데이터의 수를 늘릴 수 있지만 병해충을 포함하는 패치는 잎의 작은 영역만 관련 정보를 포함하고 있기 때문에 학습 데이터가 충분하지 않다. 또한 병해충을 포함하지 않은 영역이 해당 패치에 포함될 경우 정상 잎이나 어린잎과 같이 분류할 수 있는 문제가 있다.
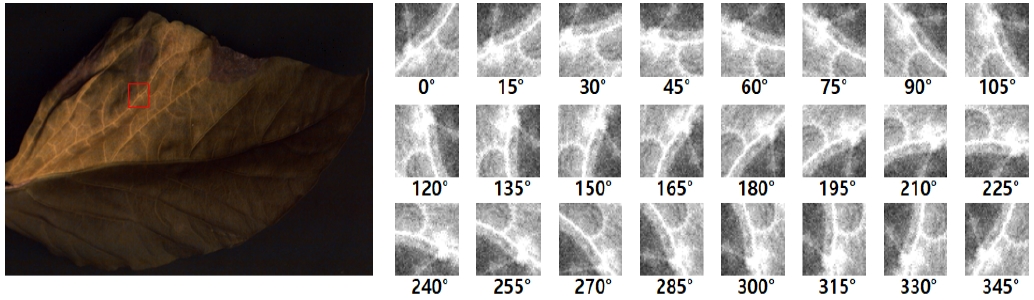
따라서 병해충 피해를 입은 잎은 그림 4처럼 원본 이미지에서 병해충이 있는 지역만을 15 ° 씩 회전하여 추출하도록 하여 패치의 수를 증가시킨다. 회전하여 잘라낸 패치는 질병과 해충에 피해를 입은 잎의 모양이 회전에 덜 의존하기 때문에 새로운 패치로 취급될 수 있다.
표 2는 데이터 전처리 후 DNN에 사용된 패치의 수를 나타낸다. 초분광 이미지의 모든 보정 작업은 Environment for Visualizing Images 소프트웨어 프로그램(ENVI 4.7, Exelis Visual Information Solutions Inc., USA)에 의해 처리 및 분석되었다.
3.4 분류기 학습 단계
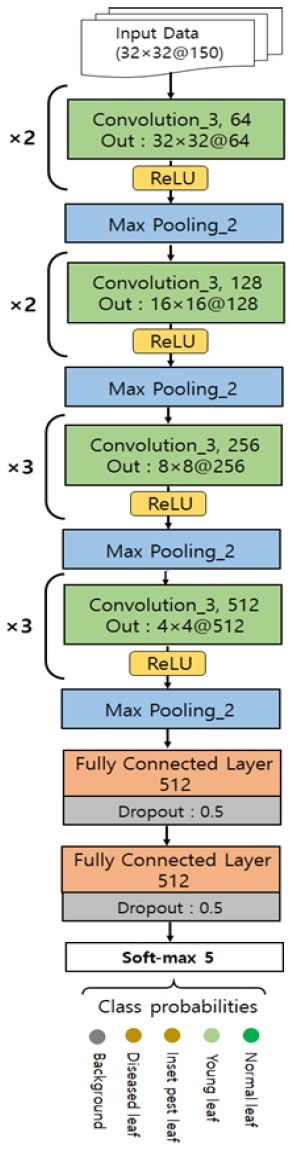
모델에 사용된 DNN의 구조는 VGG-16의 변형으로 그림 5와 같이 4개의 합성곱(Convolution) 층, 4개의 맥스 풀링(Max pooling) 층, 그리고 2개의 완전 연결(Fully connected) 층으로 이루어져 있다. 일반적으로 합성곱 층의 역할은 저수준의 특성을 자동으로 추출하는 것이며 계층이 깊어질수록 점이나 엣지 등의 기능에 영향을 미치는 범위가 넓어진다. 따라서 합성곱 층의 가장 깊은 계층은 광범위한 특징을 찾아낸다. 합성곱 층을 따르는 완전 연결 층은 합성 곱 층에서 탐색 한 기능을 활용하는 분류기를 구성한다. 또한 활성 함수로 은닉 층에서 ReLU와 출력 층에서 Soft-max를 사용한다. ReLU와 Soft-max 함수로 특징 추출 단계에서 학습한 파라미터를 이용하여 영상을 분류한다.
총 150개의 채널로 구성된 입력 구조가 그림 5에 함께 나타나 있다. 패치의 크기는 32×32이다. 모든 합성곱 필터의 크기는 3×3이며, 입력 및 출력의 데이터의 W×H는 제로 패딩으로 인해 동일하다. 그러나 채널의 수는 각 계층의 합성곱의 커널 채널수만큼 증가한다. 합성곱 층을 정해진 횟수만큼 반복한 후 풀링 계층으로 이어진다. 풀링 계층은 모두 최대 풀링과 스텝이 2이므로 합성곱 층을 거친 데이터의 W×H는 절반으로 줄어든다. 결국 32×32의 입력 패치가 네 번째 합성곱 층과 풀링 층을 거치면 출력에서 1024가지 유형의 2×2 하위 특성 맵으로 줄어든다.
하위 특성 맵은 완전 연결 층에 연결되며 각 계층은 512개의 출력이 있다. 그런 다음 Soft-max 연산이 실행되어 클래스의 수만큼 5개의 클래스 확률을 생성한다.
Ⅳ. 기법 적용 및 결과
4.1 실험 환경 및 데이터
작물 생육상태 분류 기법을 적용하기 위한 딥러닝 학습 환경은 다음과 같다. CPU는 Intel Xeon E5-2640 v4를 사용하였고, 학습을 담당하는 그래픽카드는 Nvidia Geforce GTX 1080 Ti를 사용하였다. 메모리는 64GB로 구성하였으며, 윈도우 10 환경에서 가장 범용적으로 빠른 구현이 가능한 딥러닝 프레임워크인 Tensorflow-GPU를 사용하였다.
본 논문에서는 3장에서 제안한 방법으로 그림 6과 같이 네 종류의 잎 상태 초분광 영상을 취득하였다. 각 영상은 1280W × 800H × 150C와 585MB 사이즈를 갖는다.
4.2 샘플 패치 분석 결과
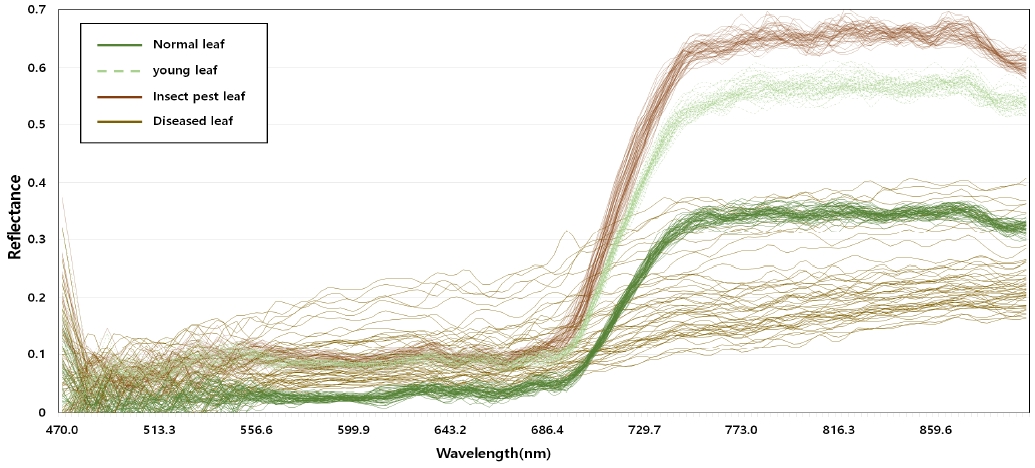
그림 7은 분류기 학습에 사용되는 잎 상태별 패치의 스펙트럼 반사율 그래프이다. 그림에서 정상 잎, 어린 잎, 해충에 피해를 입은 잎은 전 구간에서 비슷한 추이를 보이지만, 근적외선 범위(NIR 700~1000nm)에서 큰 폭의 차이를 보인다. 이러한 이유는 NIR 범위의 산란된 빛의 양이 잎의 세포 구조와 밀접하게 연관되어 있기 때문이다. 그리고 병에 걸린 잎은 다른 잎과는 다른 분포를 알 수 있다. 이러한 결과는 기존의 RGB 영상의 가시광선 범위 (VIS, 400-700nm) 만으로는 식물의 생육상태를 정확히 분류하기가 어렵다는 것을 알 수 있다.
4.3 분류기 학습 결과
그림 5의 DNN에 대한 훈련 데이터 세트는 표 2에 표시된 것처럼 25,000개의 패치로 구성하였다. 완전 연결 층의 드롭 아웃 비율은 0.5이며, 미니 배치 크기는 100이다.
표 3은 파프리카 잎의 생육 상태에 따른 이미지를 패치 크기로 잘라내어 세 가지 모델에 학습한 결과를 보여준다. 각 모델은 비선형 활성화 함수로 ReLU와 PReLU(α = 0.01)가 사용되었고, VGGNet 변형이 90.9%로 가장 높은 정확도를 보였다. 그리고 PReLU는 Dying ReLU 문제를 방지하기 위해 사용되지만 항상 ReLU 보다 나은 성능을 나타내지 않음을 확인할 수 있다.
알고리즘 성능은 표 4의 혼동 행렬(Confusion matrix)을 기준으로 정밀도(Precision), 재현율(Recall), F-점수(F-Measure)와 정확도(Accuracy)를 평가하였다. TP는 잎의 상태를 테스트 결과 정확히 맞춘 경우이며, TN은 잎의 상태가 잘못된 것을 올바르게 잘못되었다고 예측하는 경우이다. 또한 FP는 실제 잎의 상태가 잘못되었는데 올바르게 추론한 경우이며, FN은 실제 잎의 상태에 대해서 맞는 것을 올바르지 않게 틀리다고 예측한 경우이다.
정확도는 예측한 잎의 상태가 얼마나 정확한가의 비율이다. 정밀도는 모델이 옳다고 판단한 잎의 상태에서 실제 잎의 상태가 맞은 경우의 비율이고, 재현율은 실제 잎의 상태가 올바른 것 중에서 모델이 올바르게 판단한 경우의 비율이다. 이 세 지표는 하나만 높다고 하여 모델의 성능이 우수하다고 할 수 없다. 특히, 정밀도와 재현율은 트레이드오프 관계로 이를 통합한 결과인 F-점수를 평가하였다.
| (2) |
| (3) |
| (4) |
| (5) |
표 5는 테스트 패치의 분류 결과를 나타낸 것으로 알고리즘을 평가하기 위해 혼동 행렬로 나타내었다. 이를 기준으로 각 지표에 따라 평가한 결과는 표 6에 나타나있다. 평가 지표를 보면 정확도는 병에 걸린 잎, 정상 잎, 어린 잎, 그리고 해충에 피해를 입은 잎으로 분류가 잘되는 것을 확인할 수 있다.
그리고 어린잎과 해충에 피해를 입은 잎은 다른 상태에 비해 상대적으로 정확도, 정밀도, 재현율이 낮게 평가되었다. 이 결과는 그림 7의 스펙트럼 분포에서도 확인할 수 있듯이 두 잎의 분포 추이와 반사율 값이 매유 유사하기 때문인 것으로 볼 수 있다. 또한 해충에 피해를 입은 잎을 잘못 분류할 경우 해충을 조기에 판단하지 못하여 질병으로 인한 작물의 피해가 커질 수 있기 때문에 재현율을 높이기 위한 방법을 사용해야 할 것이다.
Ⅴ. 결론 및 향후 연구
초분광 영상은 비파괴 분석의 특성과 폭 넓은 스펙트럼 밴드 구성으로 많은 정보를 제공하기 때문에 농업 원격 탐사 영역에서 점점 더 많이 사용된다. 이와 함께 최근 딥러닝이 개발되면서 자동으로 학습하고 효율적인 분류기를 구성할 수 있게 되었다. 모델의 특성상 정확한 분류 작업을 위해선 많은 데이터가 필요하다. 하지만 초분광 영상은 촬영 시간도 길뿐더러, 카메라 가격도 매우 높아 많은 데이터 셋을 구성하기 어렵다.
본 논문에서는 초분광 영상과 DNN을 활용하여 작물의 생육상태 분류기법을 제안하고, 이를 파프리카 잎에 적용하여 초기 실험 결과를 제시 하였다. 또한 효과적인 분류기를 구성하기 위해서 초분광 영상에서 여러 패치들을 추출하고 다양한 전처리 기법을 사용하여 모델에 필요한 데이터의 양을 충분히 확보하였다. 그리고 합성곱 층과 완전 연결 층으로 구성되는 VGG-16의 변형인 DNN모델을 제안하였다. 이때 합성곱 층은 비선형 데이터의 특성을 자동으로 추출하고 완전 연결 층은 이를 훈련으로 정확한 분류를 위해 사용하였다.
이전 연구와 다르게 초분광 영상의 특정 스펙트럼 대역이 아닌 VIS, NIR 범위의 측정 가능한 전체 밴드 대역을 이용하였고, 병해충 탐지에 적합하지 않은 VI 지표가 아닌 DNN 모델을 이용하여 분류 기법을 제안하였다.
실험은 제안하는 패치 추출 및 데이터 생성 방법이 DNN에 부담을 주지 않고 파프리카 잎의 생육상태를 분류하는데 필요한 정보를 잘 보존하고 있다는 것을 보여준다. 또한 DNN은 초분광 영상을 사용하여 많은 스펙트럼 밴드의 조건에서 비교적 잘 분류함을 확인할 수 있었다.
제안된 기법은 패치를 분류하는 것에 그치지 않고 각 패치들의 레이블을 예측하여 클래스를 분류하는 시맨틱 세그멘테이션(Semantic segmentation)을 적용할 수 있다. 또한 식물의 줄기와 과실에도 적용하여 작물의 생육상태 분류 모델이 더욱 정확하고 빠르게 작물의 상태를 진단할 수 있다. 나아가 초분광 영상을 활용한 고성능 DNN 모델의 분류기에 장착하여 맞춤형 스펙트럼 모니터링 시스템을 구성하는데 사용할 수 있다.
Acknowledgments
이 논문은 2019년 정부(과학기술정보통신부)의 재원으로 국가과학기술연구회 창의형 융합연구사업(No. REDE-2018-JB-001)의 지원을 받아 수행된 연구임. (REDE-2018-JB-001)
References
-
F. Martinelli, R. Scalenghe, S. Davino, S. Panno, G. Scuderi, P. Ruisi, P. Villa, D. Stroppiana, M. Boschetti, and L. R. Goulart, "Advanced methods of plant disease detection. a review", Agronomy for Sustainable Development, Vol. 35, No. 1, pp. 1-25, Jan. 2015.
[https://doi.org/10.1007/s13593-014-0246-1]

-
Y. Fang and R. P. Ramasamy, "Current and prospective methods for plant disease detection", Biosensors, Vol. 5, No. 3, pp. 537-561, Aug. 2015.
[https://doi.org/10.3390/bios5030537]

-
S. H. Kim, J. G. Kang, and C. S. Ryu, "Estimation of Moisture Content in Cucumber and Watermelon Seedlings Using Hyperspectral Imagery", Protected Horticulture and Plant Factory, Vol. 27, No. 1, pp. 34-39, Jan. 2018.
[https://doi.org/10.12791/KSBEC.2018.27.1.34]

-
L. Chaerle and D. V. D. Straeten, "Imaging techniques and the early detection of plant stress", Trends in plant science, Vol. 5, No. 11, pp. 495-501, Nov. 2000.
[https://doi.org/10.1016/S1360-1385(00)01781-7]

-
P. Moghadam, D. Ward, E. Goan, S. Jayawardena, P. Sikka, and E. Hernandez, "Plant Disease Detection Using Hyperspectral Imaging", 2017 International Conference on Digital Image Computing: Techniques and Applications (DICTA), pp. 1-8, Dec. 2017.
[https://doi.org/10.1109/DICTA.2017.8227476]

-
L. Liang, L. Di, L. Zhang, M. Deng, and Z. Qin, Zhao, "Estimation of crop LAI using hyperspectral vegetation indices and a hybrid inversion method", Remote Sensing of Environment, Vol. 165, pp. 123-134, Aug. 2015.
[https://doi.org/10.1016/j.rse.2015.04.032]

-
A. Plaza, Benediktsson, J. A., J. W. Boardman, J. Brazile, L. Bruzzone, and M. Marconcini, "Recent advances in techniques for hyperspectral image processing", Remote sensing of environment, Vol. 113, Supplement 1, pp. S110-S122, Sep. 2009.
[https://doi.org/10.1016/j.rse.2007.07.028]

- Y. D. Joo, "Drone Image Classification based on Convolutional Neural Networks", The Journal of IIBC, Vol. 17, No. 5, pp. 97-102, Oct. 2017.
- J. W. Kim and P. K. Rhee, "Image Recognition based on Adaptive Deep Learning", The Journal of IIBC, Vol. 18, No. 1, pp. 113-117, Feb. 2018.
-
S. D. Cha, Y. J. Jeon, G. R. Ahn, J. I. Han, K. H. Han, and S. H. Kim, "Characterization of Fusarium oxysporum isolated from Paprika in Korea", Mycobiology, Vol. 35, No. 2, pp. 91-96, Jun. 2007.
[https://doi.org/10.4489/MYCO.2007.35.2.091]

-
Y. J. Jeon, H. W. Kwon, J. S. Nam, and S. H. Kim, "Characterization of Sclerotinia sclerotiorum isolated from Paprika", Mycobiology, Vol. 34, No. 3, pp. 154-157, Sep. 2006.
[https://doi.org/10.4489/MYCO.2006.34.3.154]

-
C. S. Yoon, E. H. Ju, Y. R. Yeoung, and B. S. Kim, "Survey of fungicide resistance for chemical control of Botrytis cinerea on paprika", The Plant Pathology Journal, Vol. 24, No. 4, pp. 447-452, Dec. 2008.
[https://doi.org/10.5423/PPJ.2008.24.4.447]

-
A. K. Mahlein, T. Rumpf, P. Welke, H. W. Dehne, L. Plumer, U. Steiner, and E. C. Oerke, "Development of spectral indices for detecting and identifying plant diseases", Remote Sensing of Environment, Vol. 128, No. 21, pp. 21-30, Jan. 2013.
[https://doi.org/10.1016/j.rse.2012.09.019]

-
L. I. U. Ke, Q. B. ZHOU, WU, W. B. Tian, X. I. A., and H. J. TANG, "Estimating the crop leaf area index using hyperspectral remote sensing", Journal of integrative agriculture, Vol. 15, No. 2, pp. 475-491, Feb. 2016.
[https://doi.org/10.1016/S2095-3119(15)61073-5]

-
C. Chion, J. A. Landry, and L. D. Costa, "A genetic-programming-based method for hyperspectral data information extraction: Agricultural applications", IEEE transactions on geoscience and remote sensing, Vol. 46, No. 8, pp. 2446-2457, Jul. 2008.
[https://doi.org/10.1109/TGRS.2008.922061]

-
G. L. Hammer, M. J. Kropff, T. R. Sinclair, and J. R. Porter, "Future contribution of crop modeling: From heuristics and supporting decision making to understand genetic regulation and aiding crop improvement", European Journal of Agronomy, Vol. 18, No. 1-2, pp. 15-31, Dec. 2002.
[https://doi.org/10.1016/S1161-0301(02)00093-X]

- S. Khairunniza-Bejo, S. Mustaffha, and W. I. W. Ismail, "Application of artificial neural network in predicting crop yield: A review", Journal of Food Science and Engineering, Vol. 4, No. 1, pp. 1-9, Jan. 2014.
-
P. Bose, N. K. Kasabov, L. Bruzzone, and R. N. Hartono, "Spiking neural networks for crop yield estimation based on spatiotemporal analysis of image time series", IEEE Transactions on Geoscience and Remote Sensing, Vol. 54, No. 11, pp. 6563-6573, Nov. 2016.
[https://doi.org/10.1109/TGRS.2016.2586602]

-
J. A. Xia, Y. Yang, H. Cao, W. Zhang, L. Xu, Q. Wang, and B. Huang, "Hyperspectral identification and classification of oilseed rape waterlogging stress levels using parallel computing", IEEE Access, Vol. 6, pp. 57663-57675, Oct. 2018.
[https://doi.org/10.1109/ACCESS.2018.2873689]

-
T. Rumpf, A. K. Mahlein, U. Steiner, E. C. Oerke, H. W. Dehne, and L. Plimer, "Early detection and classification of plant diseases with support vector machines based on hyperspectral reflectance", Computers and Electronics in Agriculture, Vol. 74, No. 1, pp. 91-99, Oct. 2010.
[https://doi.org/10.1016/j.compag.2010.06.009]

- H. Zhu, H. Cen, C. Zhang, and Y. He, "Early detection and classification of tobacco leaves inoculated with tobacco mosaic virus based on hyperspectral imaging technique", 2016 ASABE Annual International Meeting. American Society of Agricultural and Biological Engineers, pp. 1, 2016.
-
D. Moshou, C. Bravo, J. West, S. Wahlen, A. McCartney, and H. Ramon, "Automatic detection of yellow rust in wheat using reflectance measurements and neural networks", Computers and electronics in agriculture, Vol. 44, No. 3, pp. 173–188, Sep. 2004.
[https://doi.org/10.1016/j.compag.2004.04.003]

-
K. R. Gavhale and U. Gawande, "An overview of the research on plant leaves disease detection using image processing techniques", IOSR Journal of Computer Engineering (IOSR-JCE), Vol. 16, No. 1, pp. 10-16, Jan. 2014.
[https://doi.org/10.9790/0661-16151016]

-
S. Sladojevic, M. Arsenovic, A. Anderla, D. Culibrk, and D. Stefanovic, "Deep neural networks based recognition of plant diseases by leaf image classification", Computational intelligence and neuroscience, Vol. 2016, Article ID 3289801, 11pages, May 2016.
[https://doi.org/10.1155/2016/3289801]

-
A. Fuentes, S. Yoon, S. Kim, and D. Park, "A robust deep-learning-based detector for real-time tomato plant diseases and pests recognition", Sensors, Vol. 17, No. 9, pii: E2022. Sep. 2017.
[https://doi.org/10.3390/s17092022]

-
H. Yang, J. Lee, H. Lee, and H. Kim, "Automatic Detection of Paprika Diseases/Pests Outbroken during the Hydroponic Cultivation in Greenhouse using Artificial Intelligence", Journal of Institute of Control, Robotics and Systems, Vol. 24, No. 11, pp. 1020-1024, Nov. 2018.
[https://doi.org/10.5302/J.ICROS.2018.18.0165]

-
K. Park, Y. K. Hong, G. H. Kim, and J. Lee, "Classification of apple leaf conditions in hyper-spectral images for diagnosis of Marssonina blotch using mRMR and deep neural network", Computers and Electronics in Agriculture, Vol. 148, pp. 179-187. May 2018.
[https://doi.org/10.1016/j.compag.2018.02.025]

- K. Simonyan and A. Zisserman, "Very deep convolutional networks for large-scale image recognition", arXiv preprint arXiv:1409.1556, 2014.

2016년 2월 : 전북대학교 정보소재공학과(공학사)
2016년 5월 ~ 현재 : 전자부품연구원 연구원
2017년 3월 ~ 현재 : 전북대학교 소프트웨어공학과(공학석사과정)
관심분야 : IT Convergence, AI Convergence, 스마트 팜, AI

2000년 : 전북대학교 컴퓨터과학과(이학사)
2002년 : 전북대학교 대학원 전산통계학과(이학석사)
2006년 : 전북대학교 대학원 컴퓨터통계정보학과(이학박사)
2011년 3월 ~ 현재 : 전북대학교 소프트웨어공학과 강의전담교수
관심분야 : 소프트웨어공학, 소프트웨어 아키텍처, 소프트웨어 테스팅, HCI, 정형 명세 기법, 사물인터넷, 빅데이터, 기계학습, 정밀농업 등

2004년 2월 : 전주대학교 컴퓨터공학과/정보통신공학과(공학사)
2006년 2월 : 전북대학교 의용생체공학과(공학석사)
2012년 8월 : 전북대학교 컴퓨터공학과(공학박사)
2012년 5월 ~ 현재 : 전자부품연구원 IT응용연구센터 선임연구원
관심분야 : 영상인식, 인공지능, 빅데이터 분석, 스마트 팜 등

1982년 2월 : 전북대학교 전산통계학과(이학사)
1985년 8월 : 전남대학교 대학원 계산통계학과(이학석사)
1994년 8월 : 전북대학교 대학원 전산통계학과(이학박사)
2012년 1월 ~ 2013년 7월 : University of California, Irvine(UCI) 국외연구교수
1997년 ~ 현재 : 전북대학교 소프트웨어공학과 교수
관심분야 : 소프트웨어 품질/메트릭스/테스팅, 임베디드 소프트웨어/테스팅, 빅데이터 분석, 머신러닝, 정밀농업 등