CNN-RNN 특징 추출을 활용한 LncRNA 서열 데이터 기반 질병 관련 LncRNA 예측 모델 개발
초록
LncRNA(Long non-coding RNA)는 다양한 생물학적 과정에서 중요한 역할을 하며, 비정상적인 발현은 질병 발생의 주요 원인 중 하나이다. 따라서 lncRNA와 질병 간의 연관성을 밝히는 것은 질병 메커니즘을 이해하는 데 중요하다. 전통적인 생물학적 실험은 시간과 비용이 많이 들기 때문에, 딥러닝 기반 데이터 분석 기법이 이를 보완할 수 있다. 여러 방법들이 제안되었지만, 일반화 능력에 한계가 있다. 이러한 한계를 극복하기 위해, 본 논문에서는 CNN-RNN 구조의 특징 추출 모델을 사용하여 lncRNA 서열 데이터로부터 중요 특징 벡터를 추출하였다. 이 벡터를 통합된 LDA(LncRNA Disease Association) 데이터에 사용하여 lncRNA와 질병 간의 관계를 예측하였다. 실험 결과, CNN-RNN 구조를 통해 유용한 특징 벡터를 추출하여 lncRNA와 질병의 연관성을 효과적으로 예측할 수 있었다.
Abstract
Long non-coding RNA(LncRNA) plays a crucial role in various biological processes, and its abnormal expression is a major cause of disease development. Therefore, elucidating the relationship between lncRNA and diseases is essential for understanding disease mechanisms. Traditional biological experiments are time-consuming and costly, so deep learning-based data analysis techniques can complement these methods. Although several approaches have been proposed, they all have limitations in generalization ability. To overcome these limitations, this paper employs a CNN-RNN feature extraction model to derive significant feature vectors from lncRNA sequence data. These vectors are then used to predict the relationship between lncRNA and diseases using integrated LncRNA Disease Association(LDA) data. Experimental results demonstrate that the CNN-RNN structure effectively extracts useful feature vectors, enabling accurate prediction of lncRNA-disease associations.
Keywords:
long non-coding RNA, lncRNA, disease association prediction, CNN-RNN framework, sequence dataⅠ. 서 론
RNA는 단백질을 인코딩할 수 있는 messenger RNA와 인코딩할 수 없는 noncoding RNA(ncRNA)로 나누어 진다[1][2]. 그 중에서도 lncRNA(long non-coding RNA)는 200개 이상의 뉴클레오타이드(Nucleotide)로 이루어진 ncRNA로서 전사, 번역, 후성유전 조절, 스플라이싱(Splicing), 분화, 면역 반응 및 세포 주기 조절 등의 생명활동에서 중요한 역할을 한다[3][4]. 최근 연구들은 lncRNA의 비정상적인 발현이 인간의 다양한 질병 발생에 중요한 원인이 될 수 있음을 보여주고 있다[2]. 예를 들어, lncRNA는 암, 신경질환, 심혈관 질환 등 다양한 질병과 연관되어 있으며, 이러한 연관성을 밝히는 것은 질병의 예방 및 치료에 큰 도움이 된다. 그러나, lncRNA와 질병의 관계를 알아내는 전통적인 생물학적 실험 방법은 많은 시간과 비용을 소모하기 때문에, 새로운 접근 방법이 필요하다.
딥러닝 기반 데이터 분석 기법은 이러한 문제를 해결할 수 있는 유망한 도구로 부상하고 있다. 딥러닝은 대량의 생물학적 데이터를 효과적으로 처리하고, 잠재적인 lncRNA와 질병의 관계를 빠르고 저렴한 비용으로 식별할 수 있게 해준다. 이러한 방법은 전통적인 생물학적 방법에 사용할 후보군을 효과적으로 선별할 수 있으며, 연구의 효율성을 크게 향상시킬 수 있다.
본 연구에서는 lncRNA의 서열 데이터를 기반으로 CNN-RNN 구조의 특징 추출 모델을 사용하여 중요 특징 벡터를 추출하였다. 이후 이 특징 벡터를 사용하여 통합된 LDA 데이터로부터 lncRNA와 질병의 관계를 예측하였다.
Ⅱ. 관련 연구
최근 딥러닝을 포함한 인공지능 모델들이 다양한 분야에서 막강한 성능을 나타내고 있음에 따라, 질병 관련 lncRNA 추출 연구와 더불어 다양한 생물정보학 연구 분야에서도 인공지능 모델들이 핵심 기술이 되고 있다[5]-[12].
LncRNA와 질병의 연관성을 알아내기 위한 계산적 접근법 중 하나인 Ping’s method는 lncRNA-질병 연관성 데이터를 기반으로 이분 네트워크(Bipartite network)를 구축하여, lncRNA와 질병 간의 잠재적 연관성을 예측한다. 이 모델은 확인된 연관성만을 활용하여 네트워크를 형성하고, 공통 이웃 노드(Node)를 가진 노드들 간의 유사성을 계산하여 예측을 수행한다[13]. 그러나 이러한 방법은 알려진 관련 lncRNA가 없는 새로운 질병에 적용하기 어렵다.
이러한 문제를 해결하기 위해 다양한 데이터를 통합하여 lncRNA와 질병의 연관성을 예측하는 연구도 있다. Zhang et al이 제안한 LncRDNetFlow 모델은 lncRNA 유사성 네트워크, 단백질-단백질 상호작용 네트워크, 질병 유사성 네트워크 등을 포함한 여러 이질적인 데이터 네트워크를 통합한다. 이 모델은 흐름 전파(Flow propagation) 알고리즘을 사용하여 네트워크의 위상 정보(Topological information)를 고려한 글로벌 거리 측정(Global distance measurement)을 통해 잠재적인 lncRNA-질병 연관성을 예측한다[14]. LDAP는 lncRNA의 서열 정보, 단백질-단백질 상호작용, 질병 유사성 데이터 등을 이용하여 유사성을 평가하고 이를 통합하여 Bagging SVM을 활용한 예측을 수행한다[15]. MFLDA는 다양한 생물학적 데이터를 활용하여 lncRNA-질병 연관성을 예측하는 행렬 분해 기반의 데이터 퓨전(Data fusion) 방법을 사용하며, 이를 통해 잠재적인 연관성을 식별한다[16]. 그러나 이러한 방법들은 연관성의 깊고 복잡한 표현을 학습할 수 없는 얕은 학습 방법이다. 따라서 딥러닝 기술을 사용한 모델이 더 좋은 성능을 보이고 있다.
딥러닝 기술을 활용한 방법으로는 CNNLDA가 있다. 이 모델은 어텐션 메커니즘(Attention mechanism)을 포함한 이중 합성곱 신경망을 통해 구현되며, lncRNA, 질병, 그리고 miRNA(micro RNA) 간의 복잡한 상호작용과 연관성 정보를 통합적으로 분석한다. 어텐션 메커니즘을 활용하여 중요한 생물학적 특징을 강조하고, 이중 합성곱 신경망 구조를 사용하여 lncRNA-질병 연관성의 전반적인 패턴과 주목할 만한 세부 특징을 학습한다. 이를 통해 lncRNA와 질병 간의 연관성을 예측한다[17]. 하지만 이 방법은 lncRNA, 질병, miRNA 사이의 유사도만을 사용하여 학습을 진행하며, 각 객체의 고유한 특징 벡터를 포함하지 않아 일반화에 한계가 있다.
Ⅲ. 데이터셋
본 연구를 진행하기 위해 필요한 데이터셋(data set)은 세 가지 종류로 구성된다. 우선 lncRNA와 질병의 연관성을 파악할 수 있는 데이터셋이 필요하다. 다음으로, lncRNA의 특징을 파악할 수 있는 lncRNA의 서열 데이터를 사용한다. 이 서열 데이터는 각 lncRNA의 생물학적 특성을 이해하는 데 중요한 역할을 한다. 마지막으로, 질병의 특징을 표현할 수 있는 질병들의 유사성 데이터를 활용한다.
LncRNA와 질병의 연관성을 파악하기 위해, 세 개의 주요 데이터셋을 통합하여 사용하였다. 첫 번째 데이터셋은 LncRNADisease v3.0으로, PubMed 데이터베이스에서 검토된 11,840개의 논문을 기반으로 구축되었다. 이 데이터셋은 6,066개의 lncRNA와 566개의 질병에 대해 실험적으로 뒷받침되는 13,191개의 lncRNA와 질병 연관성 데이터를 포함하고 있다[18]. 두 번째 데이터셋은 Lnc2Cancer v3.0으로, 15,000개 이상의 논문을 검토하여 구축되었으며, 2,659개의 lncRNA와 216개의 암 하위 유형에 대한 9,254개의 lncRNA와 암 연관성 데이터를 포함하고 있다[19]. 세 번째 데이터셋은 RNADisease v4.0으로, 이전 버전인 MNDR v3.0에서 크게 확장된 버전으로, 58,401편의 문헌에서 추출된 343,273건의 실험적으로 검증된 데이터를 포함하고 있다. 이 데이터셋에는 117종의 생물, 18종류의 RNA, 그리고 4,090개의 질병이 포함되어 있으며, 특히 인간(Homo sapiens)을 위한 76,871건의 lncRNA-질병 연관성 데이터가 포함되어 있다[20]. 이렇게 통합된 데이터셋은 주로 lncRNA와 질병의 positive 연관성에 대한 정보를 제공하며, 소수의 negative 연관성 데이터도 포함하고 있다.
LncRNA의 특징을 표현하기 위해 RNAcentral 데이터베이스에서 lncRNA의 서열(Sequence) 데이터를 수집하였다. RNAcentral은 44개의 RNA 데이터베이스를 통합하여 1800만 개 이상의 ncRNA 서열을 제공한다[21]. 데이터 수집 방법으로는 통합된 연관성 데이터에서 lncRNA symbol과 rna_central_id를 연결하고, RNAcentral API를 이용하여 해당 rna_central_id에 대한 서열 데이터를 요청하여 수집하였다.
질병의 특징을 표현하는 데에는 질병 간의 유사성 데이터가 사용된다. 이 데이터는 SemFunSim 연구에서 제공되며, SemFunSim은 질병 간의 의미론적(Semantic) 연관성과 유전자의 기능적(Functional) 연관성을 통합하여 질병 간의 유사성을 계산하는 방법을 사용한다[22].
Ⅳ. 데이터 전처리 및 레이블링
4.1 lncRNA 특징 데이터
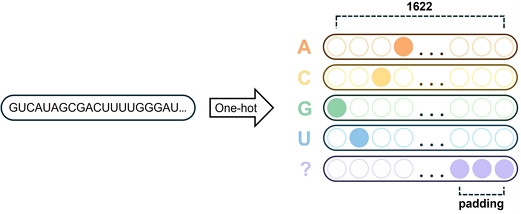
lncRNA의 특징을 분석하기 위해 서열 데이터를 사용한다. lncRNA 별로 서열의 길이가 다르기 때문에 가장 긴 서열을 기준으로 패딩(Padding) 처리를 하였다. 서열 데이터는 문자열로 이루어져 있으므로 벡터로 변환할 필요가 있다. 이를 위해 그림 1과 같이 one-hot 인코딩을 적용하여, 5개의 서열 요소를 갖는 1622차원의 벡터로 나타내었다.
4.2 질병 특징 데이터
J.-H. Park et al.[23]의 연구에 따르면, 질병-유전자 상관관계를 예측하기 위해 유전자 온톨로지(GO)와 질병 온톨로지(DO)를 사용하여 질병-유전자 간의 연관성을 예측하였을 때, 질병 간의 유사성을 더 정밀하게 반영할 수 있어 높은 예측 정확도를 달성할 수 있었다고 한다. 따라서 질병의 특징으로는 이와 관련된 질병 간 유사성 데이터를 활용하였다.
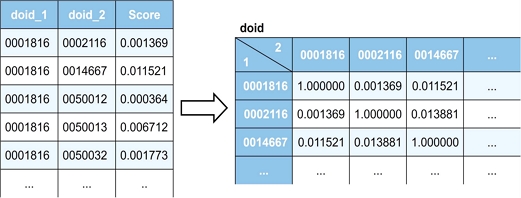
사용된 유사성 데이터는 SemFunSim 연구 결과로부터 얻은 것이다. SemFunSim은 질병 온톨로지(Disease ontology)를 이용해 두 질병 간의 의미론적 유사성을 계산하는 SemSim과, 질병 관련 유전자 집합 간의 유사성을 인간 유전자 기능 상호작용 네트워크인 HumanNet을 기반으로 계산하는 FunSim을 통합하여 최종적인 질병 유사성 점수를 제공한다[22]. 이 연구에서 제공하는 질병 간 유사성 점수 데이터를 그림 2와 같이 매트릭스(Matrix)형태로 변환하여, 3525개의 질병에 대해 3525차원의 벡터로 표현하였다.
4.3 lncRNA 와 질병 연관성 데이터 통합
이 연구에서는 세 개의 LDA 데이터셋을 통합하였다. 각각의 데이터셋이 다른 형식을 가지고 있기 때문에, 필요한 열들을 추출하고 질병 이름을 통일하는 작업을 진행하였다. 이후 중복된 데이터를 제거하여 데이터의 정확성을 높였고, lncRNA의 symbol에 해당하는 rna_central_id와 질병에 할당된 doid를 연결함으로써 lncRNA와 질병의 특징 데이터를 연계하였다. 마지막으로 라벨 데이터를 추가하여, 최종적으로 13,549개의 데이터를 완성하였다. 이 데이터셋 중 13,165개는 양성(Positive) 연관성을, 384개는 음성(Negative) 연관성을 나타내며, 이 정보는 lncRNA와 질병 간의 관계를 분석하는 데 핵심적으로 활용된다.
Ⅴ. 모 델
본 연구에서 사용된 모델은 서열 데이터와 질병 특징 벡터로부터 중요한 특징을 추출하기 위해 CNN-RNN 모델을 활용하고 있다. 이러한 모델 선택은 데이터의 특성을 고려한 것으로, lncRNA 서열 데이터와 질병 특징 벡터의 복잡한 관계를 효과적으로 학습하고 예측 성능을 극대화하기 위한 것이다. 추출된 특징들은 통합되어 완전 연결 계층(Fully connected layer)을 통해, lncRNA와 질병 간의 연관성을 분류하는 데 사용된다.
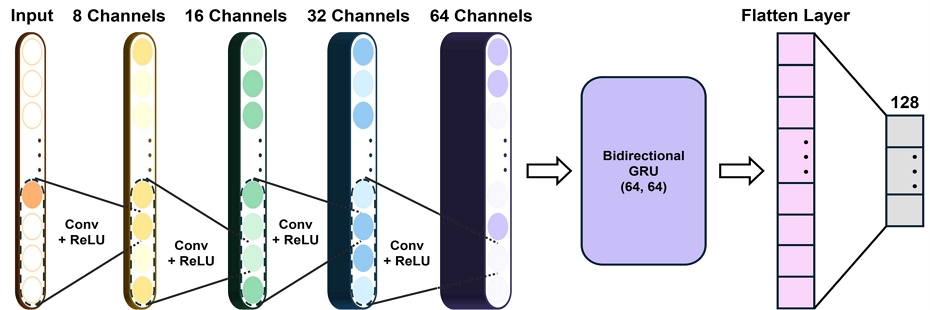
CNN-RNN 모델의 구조는 그림3과 같다. CNN-RNN 모델의 구조에서 CNN(Convolutional Neural Network)의 역할은 데이터 내에서 지역적 패턴을 포착하는 것이다. 생물학적 서열 데이터에서 이 층은 중요한 모티프(Motif)와 구조적 특성을 자동으로 학습하여 추출할 수 있다. 질병 유사성 벡터에 대해서는 CNN 층이 여러 질병 간의 상호 연관된 패턴을 식별하여 유용한 정보로 변환한다. 이러한 CNN 층은 4개의 1차원 합성곱(Convolution)층으로 구성되어 있으며, 각 층은 kernel size 4, stride 1로 설정하였다. 각 1차원 합성곱층 이후에는 ReLU 활성화 함수를 도입하여 모델의 표현력을 향상시키고, 필터(Filter) 수를 증가시켜 고차원의 특징을 추출하고자 하였다. RNN(Recurrent Neural Network) 부분에서는 GRU(Gated Recurrent Unit)를 사용하였다.
GRU는 LSTM(Long Short-Term Memory)과 비교하여 구조가 간결하면서도 장기 의존성 문제를 효과적으로 해결하며, 계산 효율성이 높다는 장점을 가진다. GRU는 서열 데이터의 연속적인 관계를 학습하는 데 적합하며, 질병 유사성 벡터에서 다양한 특성을 통합하여 분석함으로써 정확한 예측에 기여한다. GRU는 64차원의 은닉 상태를 갖는 양방향 구조로 데이터를 처리하며, GRU의 출력값은 평탄화(Flatten) 층을 거쳐 선형(Linear) 층을 통해 128차원으로 축소된다. 이후, 서열 데이터와 질병 유사성 벡터에서 추출된 128차원의 특징들을 연결(Concatenate)하여 256차원의 통합 데이터를 생성한다.
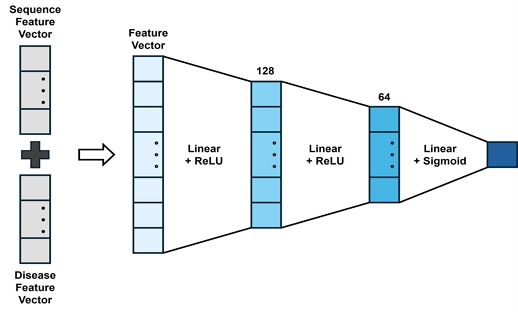
본 연구에서 사용된 분류기는 그림 4와 같이 세 계층의 완전 연결 계층으로 구성되어 있다. 첫 번째 계층에서 256차원의 입력 벡터를 받아 이를 먼저 128차원으로, 이어서 두 번째 계층에서 64차원으로 축소한다. 두 계층에서는 ReLU 활성화 함수를 적용하여 비선형성을 추가함으로써, 모델의 학습 능력을 강화한다. 마지막으로, 세 번째 계층에서는 64차원의 입력 벡터를 받아 단일 값으로 출력하며, 이는 lncRNA와 질병의 연관성을 나타내는 예측값으로 사용된다.
모델의 학습 과정에서는 BCEWithLogitsLoss 손실 함수를 사용하였다. 이 손실 함수는 이진 분류 문제에 적합하며, 모델의 예측이 정확도를 높이는 데 중요한 역할을 한다. 최적화 기법으로는 Adam 알고리즘을 선택했다. Adam은 그 효율성과 자동 조정 기능 때문에 널리 사용된다. 이 모델에서 Adam의 매개변수로는 learning_rate를 0.001로 설정하여, 모델이 학습 과정에서 안정적으로 수렴할 수 있도록 하였고, weight_decay를 0.00001로 설정하여, 과적합을 방지하고, 일반화 성능을 향상하였다.
Ⅵ. 실 험
본 연구에서는 lncRNA와 질병의 연관성 예측 모델을 최적화하기 위해 다섯 가지 실험을 진행하였으며, CNN-RNN 모델이 연관성 예측에 중요한 역할을 하는지를 확인하기 위한 실험도 수행하였다.
모든 실험은 데이터셋을 학습 데이터 60%, 검증 데이터 20%, 테스트 데이터 20%로 분할하여 수행하였으며, 모델의 성능은 테스트 데이터에 대한 AUC 값으로 평가하였다. 실험의 내용은 다음과 같다. 1) 다양한 모델 구성을 테스트함으로써 최적의 합성곱 층수를 결정하였고, 이를 통해 모델이 데이터에서 중요한 특성을 효과적으로 학습할 수 있는지를 검토하였다. 2) GRU의 양방향과 단방향 구성을 비교하여 데이터의 전체적인 문맥 파악 능력이 예측 정확도에 미치는 영향을 평가하였다. 3) 합성곱 층 이후 맥스풀링(maxpooling)을 적용하면 모델이 데이터의 중요 부분에 집중하고 계산 효율성을 높이는지 확인하였다. 4) 배치 크기 조정을 통해 모델의 학습 안정성과 성능에 미치는 영향을 관찰하였다. 5) 양성 샘플에 대한 가중치 조정을 통해 데이터의 불균형 문제를 해결할 수 있는지 실험하였다. 6) CNN-RNN 모델의 포함 여부에 따른 성능을 비교하여 CNN-RNN 모델의 효과를 확인하였다.
6.1 합성곱 층 조합 실험
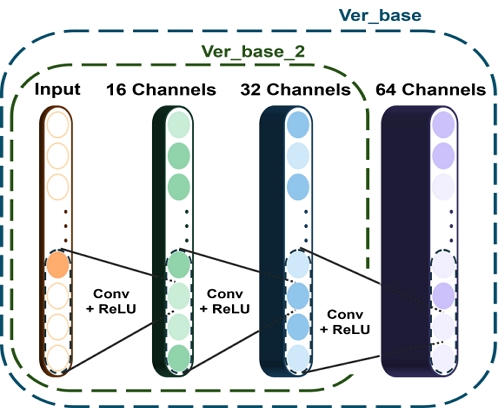
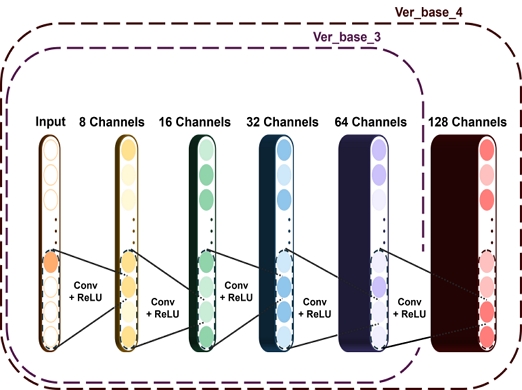
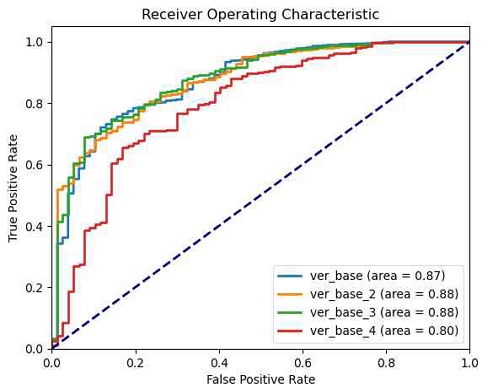
첫 번째 실험은 모델의 최적화를 목적으로 그림 5와 그림 6과 같은 다양한 합성곱 층과 필터 수의 조합을 실험하였다. 이를 통해 모델이 데이터에서 중요한 특성을 가장 효과적으로 학습할 수 있는 구성을 찾고자 하였고, 표 1의 test AUC 값을 비교한 그림 7에서 확인할 수 있다.
기본 베이스 모델(ver_base)은 [16, 32, 64] 필터 수를 갖는 세 개의 합성곱 층을 사용하여 AUC 0.873의 결과를 달성하였다. 다음으로, ver_base_2 모델에서는 합성곱 층을 두 개로 감소시키고, 필터 수를 [16, 32]로 설정하였음에도 불구하고 AUC가 0.8752로 약간 향상되어 안정적인 성능을 나타냈다.
이 결과는 모델이 적은 수의 필터로도 중요 특성을 충분히 추출할 수 있음을 시사한다. 더 나아가, ver_base_3 모델에서는 합성곱 층을 네 개로 늘리고 필터 수를 [8, 16, 32, 64]로 설정하여 성능을 개선하였으며, 이 구성은 AUC 값 0.8789로 가장 높은 성능을 기록하였다. 그러나, 합성곱 층과 필터를 추가로 늘린 ver_base_4 모델([8, 16, 32, 64, 128])에서는 AUC가 0.7971로 급격히 감소하였다. 이는 필터 수의 증가가 모델에 과적합(Overfitting)을 유발하며, 이로인해 모델이 데이터의 불필요한 데이터까지 학습하여 일반화 능력이 저하되었음을 나타낸다.
이 연구에서는 합성곱 층의 수를 네 개로 설정하고, 필터의 최적 구성을 [8, 16, 32, 64]로 결정함으로써 가장 효과적인 모델 구성을 확립하였다.
6.2 GRU 층의 양방향성 실험
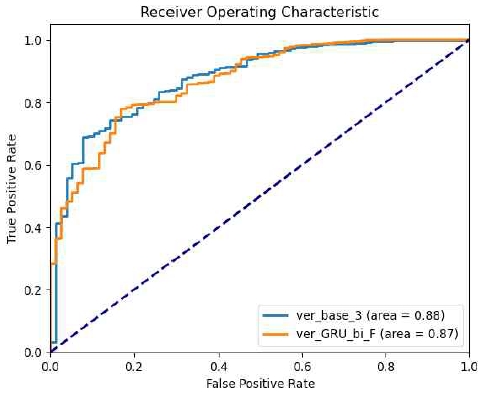
두 번째 실험은 GRU 층의 양방향성이 모델의 예측 정확도에 어떠한 영향을 미치는지 평가하는 것을 목적으로 하였고, ver_base_3에서 양방향 GRU를 단방향 GRU로 변경하여 비교하였다. 그 결과는 표 2의 test AUC 값을 비교한 그림 8에서 확인할 수 있다.
이 실험에서 양방향 GRU를 사용한 ver_base_3 모델은 AUC 값 0.8789을 기록하였으며, 이는 단방향 GRU를 사용한 ver_GRU_bi_F 모델의 AUC 값 0.8695보다 우수한 성능을 보였다. 이 결과는 데이터의 전체적인 문맥을 파악하는 능력이 모델의 예측 정확도에 중요한 역할을 한다는 것을 알 수 있다. 이는 특히 생물학적 서열 데이터에서 중요한 의미를 갖는다.
LncRNA의 서열 내에서 앞뒤의 정보가 서로에게 중요한 영향을 미칠 수 있는 경우, 양방향 GRU는 이러한 상호작용을 효과적으로 포착하여, 관련된 생물학적 기능이나 질병과의 연관성을 더욱 정확하게 예측할 수 있게 한다. 따라서, 양방향 GRU가 서열 데이터를 처리하는 데 있어서 단방향 GRU보다 우수한 선택임을 시사한다.
6.3 맥스풀링의 영향 실험
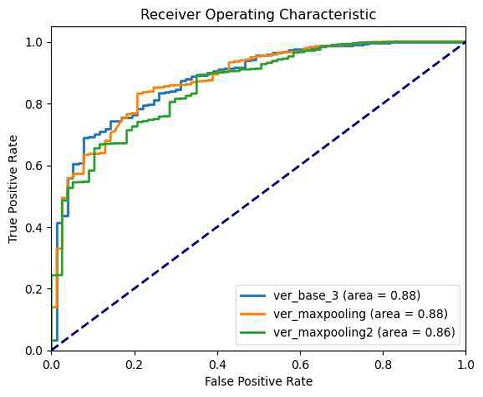
세 번째 실험에서는 맥스풀링(Maxpooling)이 모델 성능에 미치는 영향을 조사하였다. 기준 모델인 ver_base_3, 그리고 기준 모델에 맥스풀링 커널 크기를 다르게 적용한 두 모델, ver_maxpooling 및 ver_maxpooling2의 성능을 비교 분석하였고, 그 결과는 표 3의 test AUC 값을 비교한 그림 9에서 확인할 수 있다.
맥스풀링 kernel size는 2를 적용한 ver_maxpooling 모델은 AUC 값이 0.8772로, 기준 모델의 AUC 값 0.8789와 유사한 수준을 보였다. 반면, 맥스풀링 kernel size를 4로 적용한 ver_maxpooling2 모델은 AUC 값이 0.8572로, ver_maxpooling 모델보다 낮은 성능을 나타냈다. 이 결과는 맥스풀링의 적용이 모델의 전반적인 예측 성능 향상에 결정적인 영향을 미치지 않음을 시사한다. 하지만 성능의 큰 차이가 없는 경우, 맥스풀링 적용이 모델의 계산 효율성을 높일 수 있음을 의미한다.
6.4 배치 크기의 영향 실험
네 번째 실험은 기준 모델 ver_base_3를 사용하여 다양한 배치 크기(32, 64, 128, 256)의 영향을 평가하였고, 그 결과는 표 4의 test AUC 값을 비교한 그림 10에서 확인할 수 있다.
배치 크기가 64일 때 모델은 가장 높은 AUC 값을 0.8789로 기록하였으며, 이는 다른 배치 크기에 비해 우수한 성능을 나타냈다. 배치 크기 256에서 AUC 값은 0.8729로 나타났고, 128에서는 0.871, 가장 작은 배치 크기인 32에서는 0.824로 가장 낮은 성능을 보였다. 따라서, 본 연구의 데이터에서 배치 크기를 64로 설정할 때 모델이 LncRNA와 질병의 연관성 예측에 있어 가장 높은 정확도를 달성할 수 있음을 제안한다.
6.5 가중치 영향 실험
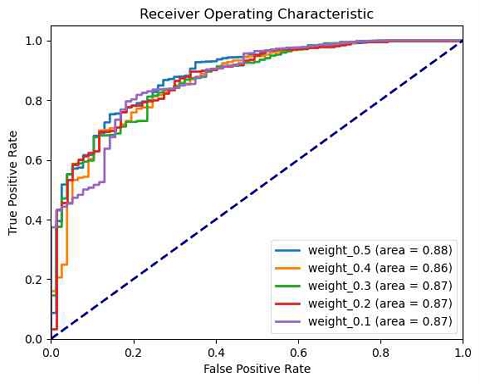
본 연구에서는 데이터의 불균형 문제를 해결하기 위해 양성 샘플에 대한 가중치(pos_weight) 조절을 시도하였다. 기준 모델인 ver_base_3을 사용하고, 양성 샘플의 가중치를 0.1부터 0.9까지 다양하게 적용하여 그 영향을 평가하였고, 그 결과는 표 5의 test AUC 값을 비교한 그림 11과 그림 12에서 확인할 수 있다.
가중치가 0.5일 때 AUC 값이 0.8832로 가장 높게 나타났다. 그러나 전반적으로 가중치 조정에 따른 AUC 값들이 큰 차이를 보이지 않았다.
6.6 CNN-RNN 모델의 연관성 예측 성능 검증
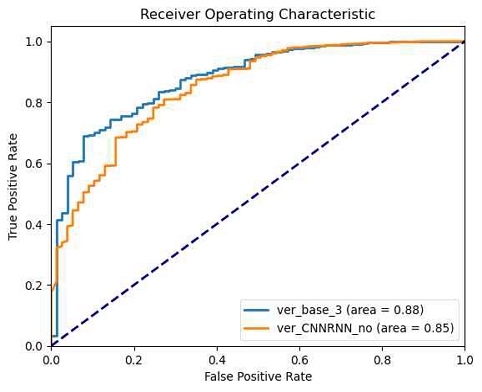
여섯 번째 실험으로 CNN-RNN 모델의 포함 여부에 따른 lncRNA-질병 연관성 예측 모델의 성능을 비교하였고, 그 결과는 표 6의 test AUC 값을 비교한 그림 13에서 확인할 수 있다.
CNN-RNN 모델이 포함된 ver_base_3의 AUC 값은 0.8789이고, CNN-RNN 모델 대신 완전 연결 계층이 적용된 ver_CNNRNN_no의 AUC값은 0.8487로 CNN-RNN 모델이 포함된 ver_base_3에 비해 낮은 AUC 값을 보였다. 이 결과, CNN-RNN 모델이 lncRNA-질병 연관성 예측에서 중요한 역할을 하여, 완전 연결 계층에 비해 서열 데이터와 질병 유사성 데이터에서 중요한 특징을 효과적으로 추출하여 예측 성능을 향상시킨다는 것을 확인할 수 있었다. 이는 lncRNA와 질병 간의 연관성을 효율적이고 정확하게 예측하는 데 있어 CNN-RNN 모델이 유용함을 뒷받침한다.
6.7 다른 방법과 비교실험
본 연구에서는 제안한 ver_base_3 모델의 성능을 평가하기 위해 기존의 여러 모델들과 비교 실험을 수행하였다. 비교 대상 모델들은 CNNLDA, SIMCLDA, Ping's method, MFLDA, LDAP 모델들로, AUC 값은 CNNLDA 논문의 비교 실험 부분에서 얻었다. 실험 방법은 CNNLDA 논문의 방식과 동일하게 평가를 수행하였다. 따라서 동일한 데이터셋의 질병 402개에 대해 실험을 진행하였으며, 음성 표본은 질병과 lncRNA의 무작위 조합 방식으로 생성하였다. 평가 방법으로는 5겹 교차 검증(5-fold validation)을 사용하여 평균 AUC 값을 구하였다.
실험 결과, 제안된 ver_base_3 모델은 AUC 0.918의 성능을 기록하였다. 이는 다른 비교 모델들인 SIMCLDA, Ping’s method, MFLDA와 LDAP와 비교했을 때 높은 예측 성능을 보여주었다. CNNLDA 모델이 AUC 0.952로 가장 높은 성능을 보였으나, 제안된 ver_base_3 모델은 lncRNA의 서열 데이터와 질병 유사도 데이터를 통합하는 과정에서 모든 데이터를 포함하지 못했다. 그 결과, 402개의 질병 중 192개의 질병에 대한 표본만을 학습할 수 있었다. 이러한 제한에도 불구하고, 제안된 ver_base_3 모델의 예측 정확도는 여전히 높은 수준을 유지하며, 경쟁력 있는 성능을 보여줬다.
Ⅶ. 결론 및 향후 과제
본 연구에서는 CNN-RNN 구조를 활용하여 lncRNA의 서열 데이터와 질병 유사성 데이터로부터 중요한 특징을 추출하였다. 이후, 다양한 LDA (LncRNA Disease Association) 데이터를 통합하여 lncRNA와 질병 간의 연관성을 예측하고, 다양한 실험 결과를 통해 본 연구에서 제안하는 모델의 우수성을 증명하였다.
제안하는 방법론에서는 유전 정보가 함축된 서열 데이터로부터 CNN-RNN 기반 특징 추출 모델을 적용하여 유의미한 특징 벡터를 추출함으로써 lncRNA와 질병 간의 연관성을 효과적으로 예측할 수 있었다. 특히, 본 연구에서 제안하는 모델의 우수성은 다음과 같은 원인에 기반하고 있다. 1) CNN을 통해 서열 데이터의 유전 정보 간의 지역적 패턴을 포착하고 2) 추출된 특징 벡터에 RNN 모델을 활용하여 서열 데이터의 순차적인 유전 정보를 더욱 효과적으로 정제하였다. 결과적으로 CNN과 RNN 모델을 결합하여 적용한 서열 데이터 기반 특징 벡터 추출이 lncRNA와 질병 간의 복잡한 연관성을 밝히는 데 있어서 중요한 역할을 한다는 것을 시사하며, 이는 생물정보학에서 딥러닝 기반 방법론의 적용이 다양한 생물학적 문제를 해결하는 핵심 기술로 자리매김할 수 있음을 보여준다.
본 연구에서 제안한 모델에는 한계점이 존재한다. lncRNA와 질병의 연관성 데이터에서 양성 표본과 음성 표본 간의 심한 불균형이 관찰되었다. 이러한 불균형은 모델 학습 과정에서 특정 클래스에 대한 예측 성향을 강화하여 실제 예측 성능의 저하를 초래하는 주요 요인으로 작용하였다. 따라서, 미래의 연구에서는 데이터의 불균형 문제를 효과적으로 해결할 수 있는 새로운 방안을 모색하는 것이 필요하다.
J. Kim et al.[24]연구에 따르면, 거리 기반 데이터 레이블링을 통해 음성 데이터를 생성하여 이를 해소하고자 했다. 그러나 계산에 사용된 각각의 거리 측정 방법의 한계로 인해 모델이 특정 특징에 과도하게 의존하게 만들어 일반화 능력을 저하시킬 수 있다. 이를 해결하기 위한 하나의 제안으로, LDA에 존재하는 소수의 음성 데이터를 기반으로 양성 데이터를 제외한 무작위 LncRNA와 질병의 조합에 그래프 전파(Graph propagation) 방법을 활용하여 추가적인 음성 데이터를 샘플링(Sampling)하는 방식을 고려할 수 있다.
Acknowledgments
이 성과는 정부(과학기술정보통신부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임 (2022R1G1A1003616)
이 성과는 정부(과학기술정보통신부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임 (RS-2023-00242528)
References
-
J. Beermann, M.-T. Piccoli, J. Viereck, and T. Thum, "Non-coding RNAs in Development and Disease: Background, Mechanisms, and Therapeutic Approaches", Physiological Reviews, Vol. 96, No. 4, pp. 1297-1325, Oct. 2016.
[https://doi.org/10.1152/physrev.00041.2015]

-
X. Zhang, R. Hong, W. Chen, M. Xu, and L. Wang, "The role of long noncoding RNA in major human disease", Bioorganic Chemistry, Vol. 92, pp. 103214, Nov. 2019.
[https://doi.org/10.1016/j.bioorg.2019.103214]

-
K. C. Wang and H. Y. Chang, "Molecular Mechanisms of Long Noncoding RNAs", Molecular Cell, Vol. 43, No. 6, pp. 904-914, Sep. 2011.
[https://doi.org/10.1016/j.molcel.2011.08.018]

-
M. Kazimierczyk, M. K. Kasprowicz, M. E. Kasprzyk, and J. Wrzesinski, "Human Long Noncoding RNA Interactome: Detection, Characterization and Function", IJMS, Vol. 21, No. 3, pp. 1027, Feb. 2020.
[https://doi.org/10.3390/ijms21031027]

-
J. Ha, C. Park, C. Park, and S. Park, "IMIPMF: Inferring miRNA-disease interactions using probabilistic matrix factorization", Journal of Biomedical Informatics, Vol. 102, pp. 103358, Feb. 2020.
[https://doi.org/10.1016/j.jbi.2019.103358]

-
J. Ha, C. Park, C. Park, and S. Park, "Improved prediction of miRNA-disease associations based on matrix completion with network regularization", Cells, Vol. 9, No. 4, pp. 881, Apr. 2020.
[https://doi.org/10.3390/cells9040881]

-
J. Ha, C. Park, and S. Park, "PMAMCA: prediction of microRNA-disease association utilizing a matrix completion approach", BMC Systems Biology, Vol. 13, No. 1, pp. 1-13, Mar. 2019.
[https://doi.org/10.1186/s12918-019-0700-4]

-
J. Ha and C. Park, "MLMD: Metric Learning for Predicting MiRNA-Disease Associations'', IEEE Access, Vol. 9, pp. 78847-78858, May 2021.
[https://doi.org/10.1109/ACCESS.2021.3084148]

-
J. Ha, "MDMF: Predicting miRNA–Disease Association Based on Matrix Factorization with Disease Similarity Constraint", Journal of Personalized Medicine, Vol. 12, No. 6, pp. 885, May 2022.
[https://doi.org/10.3390/jpm12060885]

-
J. Ha and S. Park, "NCMD: Node2vec-based neural collaborative filtering for predicting miRNA-disease association", IEEE/ACM Transactions on Computational Biology and Bioinformatics, Vol. 20, No. 2, pp. 1257-1268, Mar. 2022.
[https://doi.org/10.1109/TCBB.2022.3191972]

-
J. Ha, "SMAP: Similarity-based matrix factorization framework for inferring miRNA-disease association", Knowledge-Based Systems, Vol. 263, pp. 110295, Mar. 2023.
[https://doi.org/10.1016/j.knosys.2023.110295]

-
J. Ha, "LncRNA Expression Profile-Based Matrix Factorization for Predicting lncRNA- Disease Association", IEEE Access, Vol. 12, pp. 70297-70304, May 2024.
[https://doi.org/10.1109/ACCESS.2024.3401005]

-
P. Ping, L. Wang, L. Kuang, S. Ye, M. F. B. Iqbal, and T. Pei, "A Novel Method for LncRNA-Disease Association Prediction Based on an lncRNA-Disease Association Network", IEEE/ACM Trans. Comput. Biol. and Bioinf., Vol. 16, No. 2, pp. 688-693, Mar. 2019.
[https://doi.org/10.1109/TCBB.2018.2827373]

-
J. Zhang, Z. Zhang, Z. Chen, and L. Deng, "Integrating Multiple Heterogeneous Networks for Novel LncRNA-Disease Association Inference", IEEE/ACM Trans. Comput. Biol. and Bioinf., Vol. 16, No. 2, pp. 396-406, Mar. 2019.
[https://doi.org/10.1109/TCBB.2017.2701379]

-
W. Lan, et al., "LDAP: a web server for lncRNA-disease association prediction", Bioinformatics, Vol. 33, No. 3, pp. 458-460, Feb. 2017.
[https://doi.org/10.1093/bioinformatics/btw639]

-
G. Fu, J. Wang, C. Domeniconi, and G. Yu, "Matrix factorization-based data fusion for the prediction of lncRNA–disease associations", Bioinformatics, Vol. 34, No. 9, pp. 1529-1537, May 2018.
[https://doi.org/10.1093/bioinformatics/btx794]

-
P. Xuan, Y. Cao, T. Zhang, R. Kong, and Z. Zhang, "Dual Convolutional Neural Networks With Attention Mechanisms Based Method for Predicting Disease-Related lncRNA Genes", Front. Genet., Vol. 10, pp. 416, May 2019.
[https://doi.org/10.3389/fgene.2019.00416]

-
X. Lin, et al., "LncRNADisease v3.0: an updated database of long non-coding RNA-associated diseases", Nucleic Acids Research, Vol. 52, No. D1, pp. D1365-D1369, Jan. 2024.
[https://doi.org/10.1093/nar/gkad828]

-
Y. Gao, et al., "Lnc2Cancer 3.0: an updated resource for experimentally supported lncRNA/circRNA cancer associations and web tools based on RNA-seq and scRNA-seq data", Nucleic Acids Research, Vol. 49, No. D1, pp. D1251-D1258, Jan. 2021.
[https://doi.org/10.1093/nar/gkaa1006]

-
J. Chen, et al., "RNADisease v4.0: an updated resource of RNA-associated diseases, providing RNA-disease analysis, enrichment and prediction", Nucleic Acids Research, Vol. 51, No. D1, pp. D1397–D1404, Jan. 2023.
[https://doi.org/10.1093/nar/gkac814]

-
RNAcentral Consortium, "RNAcentral 2021: secondary structure integration, improved sequence search and new member databases", Nucleic Acids Research, Vol. 49, No. D1, pp. D212-D220, Jan. 2021.
[https://doi.org/10.1093/nar/gkaa921]

-
L. Cheng, J. Li, P. Ju, J. Peng, and Y. Wang, "SemFunSim: A New Method for Measuring Disease Similarity by Integrating Semantic and Gene Functional Association", PLoS ONE, Vol. 9, No. 6, pp. e99415, Jun. 2014.
[https://doi.org/10.1371/journal.pone.0099415]

-
J.-H. Park and Y.-R. Cho, "Disease-Gene Association Prediction using Heterogeneous Networks based on Ontologies", JKIIT, Vol. 21, No. 2, pp. 145-153, Feb. 2023.
[https://doi.org/10.14801/jkiit.2023.21.2.145]

-
J. Kim, S.-W. Yoon, I.-W. Hwang, and K.-C. Lee, "LncRNA-Disease Association Prediction Model Applying Distance-based Data Labeling", JKIIT, Vol. 50, No. 5, pp. 420-428, May 2023.
[https://doi.org/10.5626/JOK.2023.50.5.420]


2024년 8월 : 부경대학교 냉동공조공학과(공학사)
2024년 8월 : 부경대학교 빅데이터융합전공(공학사)
2024년 9월 ~ 현재 : 부경대학교 데이터공학과 석사과정
관심분야 : 기계학습, 생물정보학, 데이터마이닝, 딥러닝, 추천시스템

2015년 8월 : 연세대학교 컴퓨터과학과(공학석사)
2020년 8월 : 연세대학교 컴퓨터과학과(공학박사)
2020년 ~ 2021년 : 하와이 암센터 포스닥 연구원
2021년 9월 ~ 현재 : 부경대학교 데이터정보과학부 빅데이터융합전공 조교수
관심분야 : 기계학습, 생물정보학, 데이터마이닝, 딥러닝, 추천시스템