GRU 모델을 통한 QQ 기반 산소추출률 예측 정확도 향상 방법
초록
자기공명영상(MRI, Magnetic Resonance Imaging)을 활용하여 뇌 조직의 혈액 내 산소추출률을 계산하여 뇌 질환을 분석할 수 있다. 기존 연구에서는 QQ 모델을 통하여 CCM, CAT 등 클러스터 기법을 사용하여 산소추출률을 계산하였고, DNN 등 딥러닝 모델을 활용하는 연구도 초기 단계이다. 뇌 MRI 데이터는 시계열적인 특성이 있기에, 순환적인 구조를 갖는 GRU 모델이 DNN 모델보다 시계열 데이터를 더 효과적으로 처리할 수 있다. 따라서 본 논문에서는 QQ 모델을 기반으로 GRU 모델을 구성한 후에 뇌 MRI 데이터를 활용하여 산소추출률 예측의 정확도를 향상하는 방법을 제안하였다. 제안하는 GRU 모델의 학습과 평가는 환자 17명의 뇌 MRI 신호 감쇄 및 자화율을 이용하여 진행하였으며, 평균적으로 RMSE 5.39 정확도로 OEF를 예측했다.
Abstract
Using Magnetic Resonance Imaging(MRI), brain diseases can be analyzed by calculating the Oxygen Extraction Fraction(OEF) in the blood of the brain tissue. In existing research, the oxygen extraction rate was calculated using cluster techniques such as CCM and CAT through the QQ model, and research using deep learning models such as DNN is also in its early stages. Because brain MRI data has time-series characteristics, the GRU model with a circular structure can process time-series data more effectively than the DNN model. Therefore, this paper propose a method to improve the accuracy of OEF prediction by constructing a GRU model based on the QQ model. The proposed GRU model was trained and tested using MRI signal attenuation and susceptibility of 17 patients. A OEF prediction accuracy of 5.39 was achieved.
Keywords:
brain MRI, oxygen extraction fraction, QQ model, GRU, deep learningⅠ. 서 론
뇌와 관련된 질병은 그 심각성으로 인해 많은 사람에게 영향을 미치고 있다. 일부 뇌 질환은 치명적이어서 조기 발견과 정확한 진단은 생명을 구하는 결정적인 역할을 할 수 있다. 이를 위해 비침습적 자기공명영상(MRI, Magnetic Resonance Imaging)이 많이 사용되며, 뇌를 촬영한 영상을 분석하여 다양한 뇌 질환에 대한 검사가 가능하다.
MRI를 통해 뇌 영상을 분석하는 방법의 하나는 뇌 조직의 산소사용률을 사용하는 것이다. 이 방법은 뇌 조직에서 산소를 사용하는 정도를 측정하여 뇌 질환의 조기 진단과 치료에 도움을 줄 수 있다. 뇌 조직에서의 산소사용률은 정상적인 뇌 활동과 뇌 질환 간에 차이가 있을 수 있으며, 이를 분석함으로써 뇌졸중, 뇌종양, 알츠하이머병 등의 질환을 조기에 감지할 수 있다. 뇌 조직의 산소사용률은 산소추출률(OEF, Oxygen Extraction Fraction)을 통해 알 수 있고, QUIXOTIC[1], Calibrated fMRI[2], qBOLD[3], QQ[4]가 OEF 예측을 위해 제안되었다.
본 논문에서는 뇌 MRI와 같은 시계열 데이터를 효과적으로 처리할 수 있는 GRU(Gated Recurrent Unit) 모델을 구성한 후에 QQ 기반 산소추출률 예측 정확도를 향상하기 위한 방법을 제안한다. GRU 모델은 1개 입력 계층, 4개 GRU 계층, 다차원의 입력 데이터를 1차원 형태로 변환하기 위한 Flatten 계층, 4개 Dense 계층, 1개 출력 계층으로 구성하였다. 또한 제안한 모델은 최적화 과정을 거쳐 정확도를 향상시켰고, 17명의 환자로부터 수집된 MRI 신호 데이터를 활용하여 학습 및 검증을 수행하였다.
본 논문의 구성은 다음과 같다. 2장에서는 관련 연구를 요약하고, 3장에서는 뇌 MRI에서 GRU 모델을 통한 QQ 기반 산소추출률 예측 정확도 향상 방법에 관해 설명한다. 4장에서는 수행한 실험과 분석 결과를 제시하며, 마지막으로 5장에서는 연구 결과를 종합하여 결론을 도출한다.
Ⅱ. 관련 연구
인간의 뇌는 전체 산소의 20%를 사용하며, 뇌의 산소사용률은 혈류와 산소추출률을 통해 파악할 수 있고 다양한 뇌 질환에서 중요한 바이오마커로 활용될 수 있다. 특히, 진단에 사용되는 MRI 기술의 발전은 비침습적이며 방사선의 노출이 없고, 더 광범위하게 사용될 수 있는 장점으로 산소추출률 측정 방법에 새로운 가능성을 보였다[5].
2.1 QQ 모델 및 OEF 계산
혈액의 디옥시헤모글로빈 효과를 모델링하는 위상 신호 기반 QSM 모델은 정맥 내 디옥시헤모글로빈 영향과 비혈액 조직의 자화율을 구별할 수 있고, 세기 신호 기반 qBOLD 모델은 mGRE 시퀀스 MRI 신호 강도를 활용해 OEF 영향을 모델링할 수 있다. 이 QSM 모델과 qBOLD 모델은 제약이 있어서, 이를 결합한 QQ는 QSM과 qBOLD 모델에서 요구되는 파라미터들을 상호 보완하고, 기존 OEF 계산 모델들이 요구하는 특정 가정을 필요로 하지 않는다.
OEF 예측을 위한 QQ는 QSM과 qBOLD를 결합하여 식 (1)과 같이 정의된다[6].
| (1) |
여기서, Y는 정맥의 산소포화도, v는 정맥 혈액량, R2는 BOLD 효과가 없는 조직의 가로 이완, S0는 에코 타임이 0일 때의 신호 세기, χnb는 비혈류 물질의 자화율이다. 또한, λ는 정규화 가중치, w는 QSM 가중치이다. FQSM 모델과 SqBOLD 모델은 Cho et al에 의해 설명되어 있다[6].
OEF는 정맥 산소포화도 Y를 이용하여 1 - Y/Ya를 통해 계산할 수 있다. Ya는 동맥 산소화 값으로 0.98이며 보통 동맥에서 산소화 실험으로 획득한 값을 사용한다[7].
2.2 클러스터 기반 방법
이론적으로 QQ 모델은 mGRE 시퀀스 MRI 신호의 크기와 위상을 사용하여 계산할 수 있지만, 실제로 정맥 혈액량(v)와 정맥 산소포화도(Y) 사이의 관계, 그리고 qBOLD 역변환의 잡음 민감도 등으로 인하여 계산이 복잡하다[7][8]. 이러한 문제를 해결하기 위해 클러스터 기반으로 OEF를 계산하는 CCM(Cluster-based Classification and Matching) 방법[7]과 CAT(Cluster Analysis of Time evolution) 방법[8]이 연구되었다.
CCM은 클러스터 기반의 분류 방법과 정합 및 보간을 통해 샘플 데이터를 처리하고, 이를 통해 QQ 모델이 갖는 잡음에 대한 민감성 문제를 해결하는 접근 방법을 도입했다. 또한, 최근 제안된 QQ 모델을 도입함으로써 특정 가정에 국한되지 않고 일반적인 상황에서도 정상적인 OEF 계산을 수행할 수 있다[7].
CAT는 mGRE 시퀀스에서 유사한 신호 변화를 갖는 복셀들을 같은 클러스터 그룹으로 지정하고, 같은 모델링 파리미터 값을 갖는 것으로 가정한 후 클러스터들에 대하여 평균화 수행하여 파라미터를 추정하였다. 이러한 과정을 통해 SNR을 크게 향상하고 파라미터 추정을 견고하게 수행하였다[8].
2.3 DNN 기반 딥러닝 모델
최근에 딥러닝 기술의 급격한 발전과 확산으로 OEF 계산에도 딥러닝을 활용한 연구가 진행되고 있다[9]-[11].
Kim and Lee는 QQ 모델 기반의 DNN 모델을 이용하여 OEF를 예측하는 연구를 진행하였고, 이 연구에서는 학습과 검증에 사용하지 않은 환자 11명의 평가 데이터를 사용하여 OEF 예측에 대하여 평균 RMSE를 계산하여 정확도 성능 평가를 수행한 결과 클러스터 기반의 CCM 및 CAT 방법보다 높은 정확도를 보여서 딥러닝 적용의 효율성과 가능성을 보였다[10].
그러나 뇌 MRI 데이터는 시계열적인 특성이 있기에 DNN 모델보다 순환적인 구조를 갖는 GRU 모델이 더 효과적일 것으로 판단되며 본 연구에서는 GRU 기반의 신경망을 통하여 더 정확한 OEF 값을 예측할 수 있는 연구를 수행하였다.
Ⅲ. GRU 모델을 통한 QQ 기반 산소추출률의 예측 정확도 향상 방법
본 절에서는 OEF를 정확하게 계산하기 위해, 뇌 MRI 데이터에 QQ 모델을 적용하고 이를 기반으로 한 GRU를 사용하여 OEF 예측 정확도를 향상하는 새로운 방법을 제안한다.
3.1 GRU
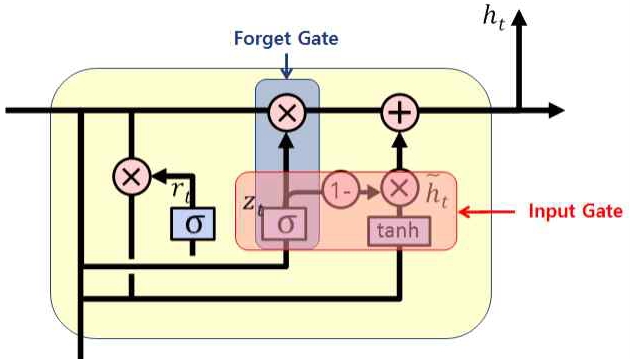
GRU는 순차 데이터를 처리에 사용되는 모델로 장단기 정보를 이용하는 LSTM(Long Short-Term Memory) 네트워크를 개선한 형태이다[12][13]. 그림 1과 같이 GRU에서는 LSTM의 Cell State(C(t))와 Hidden State(h(t))가 하나의 벡터 h(t)로 합쳐지고 LSTM의 input gate와 forget gate를 하나의 update gate로 통합하며, output gate를 reset gate로 대체했다. update gate는 이전 상태의 정보를 얼마나 가져와야 할지를 결정하며 reset gate는 이전 상태를 얼마나 잊어야 하는지를 결정한다. LSTM에서는 forget과 input이 독립적이었으나, GRU에서는 전체 양이 정해져 있어서 forget한 만큼 input하는 방식으로 제어한다. 이는 gate controller인 z(t)에 의해서 조절된다. 이러한 변화로 인해 GRU 모델은 LSTM의 gate 개념을 유지하면서 gate의 개수를 줄이고 매개변수도 줄여 LSTM에 비해 학습 시간을 단축할 수 있고, 낮은 계산 복잡성을 가지고 있다.
GRU 모델은 순환적인 구조를 가지고 있기 때문에 시간에 따라 변하는 시계열적인 특성을 가진 뇌 MRI 데이터를 DNN 모델에 비해 더 효과적으로 처리할 수 있다.
뇌 MRI 데이터에서는 이전 타임 스텝의 정보가 현재 상태 및 미래 예측에 중요한 역할을 하기 때문에 GRU의 장단기 메모리 관리가 장점을 갖는다. 또한 정보의 흐름을 조절하는 update 게이트와 reset 게이트를 통해 모델이 어떤 정보를 유지하고 무시할지를 학습하므로 DNN 모델에 비해 더 많은 파라미터를 학습하면서도 모델이 불필요한 정보를 버리고 중요한 정보에 집중할 수 있도록 도와준다.
3.2 GRU 모델 구조
GRU 모델을 위한 입출력 데이터는 CAT 방법을 개발한 코넬대학교 의과대학의 데이터셋을 활용하였다[8]. 이 데이터셋은 QQ 모델의 입력 변수들인 정맥의 산소포화도(Y), 정맥 혈액량(v), BOLD 효과가 없는 조직의 가로 이완(R2), 에코 타임 0에서의 신호 세기(S0), 비혈류 물질의 자화율(χnb)과 7개의 에코 타임에서의 뇌 MRI 신호값 및 자화율로 구성되어 있다[7].
제안된 GRU 모델에서는 MRI 촬영으로 얻은 7개의 에코 타임에서의 신호값과 1개의 자화율을 시간 개념을 포함하는 타임 스텝이 5인 시퀀스 데이터로 전처리한 후 입력 데이터로 사용하였다. 입력 데이터에 정규화 과정은 수행하지 않았다. 또한, 이 모델은 OEF 계산에 활용하는 QQ에서 요구되는 파라미터인 정맥 혈액량, 정맥 산소포화도, 에코 타임 0에서의 신호 강도, 조직의 가로 이완, 비혈류 물질의 자화율을 출력으로 구성하였다.
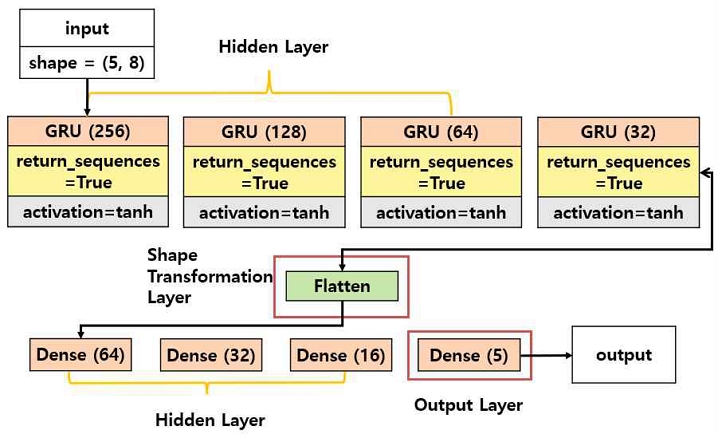
본 논문에서 구성한 GRU 모델의 구조는 그림 2에 도시하였다. 에코 시간에서 측정된 7개의 MRI 신호값과 1개의 자화율을 구성되어 있는 8개의 노드의 입력 계층으로 시작하며 이후 총 4개의 GRU 은닉 계층을 거치며 노드의 수를 256개부터 시작하여 점차 줄어드는 구조로 구성되어 있다. 이후 모든 GRU 층은 시퀀스의 모든 타임스텝에 대한 출력을 반환하고 Dense 계층에서 적용하기 위해 다차원의 입력 데이터를 1차원으로 평탄화하는 기능을 수행하는 Flatten 계층을 가지며 평탄화 과정을 거친 후에 4개의 Dense 계층을 거치며 다양한 층과 뉴런을 통해 복잡한 패턴을 학습한다. 최종적으로 QQ 모델의 입력 파라미터에 해당하는 정맥 혈액량, 정맥 산소포화도, 에코 타임 0에서의 신호 강도, 조직의 가로 이완, 비혈류 물질의 자화율을 예측하기 위한 5개 노드로 구성된 출력값을 예측한다. 또한, 은닉 계층에서 노드의 수를 점차 줄이면서 여러 계층을 쌓는 방식으로 설계되었는데, 이러한 구조는 모델의 복잡성을 감소시키면서 과적합도 방지할 수 있다.
가중치 업데이트를 위한 활성화 함수로는 GRU 계층에서는 하이퍼볼릭 탄젠트를 사용하였고, Dense 계층에서는 선형 활성화 함수를 사용했다.
이러한 구조를 통해 제안한 GRU 모델은 더 효과적으로 OEF를 계산할 수 있으며 예측 정확도를 더 향상시킬 수 있다.
Ⅳ. 실험 결과 및 분석
제안하는 방법은 Intel i9-11900 CPU, nVidia RTX 3090 GPU, 64GB RAM 하드웨어 및 Windows 10 Pro 운영체제에서 Tensorflow의 Keras로 구현하였다.
4.1 학습과 검증 정확도
코넬대학교 의과대학의 데이터셋을 사용하여 GRU 모델의 학습과 검증을 진행했다. 이 데이터셋에는 17명의 환자에게 수집한 뇌 MRI 신호값과 자화율 데이터가 포함되어 있고, 이에 대응한 QQ 모델의 정맥 혈액량, 정맥 산소포화도, 에코 타임 0에서의 신호 강도, 조직의 가로 이완, 비혈류 물질의 자화율에 대한 Ground Truth 값을 포함한다[7][8].
3차원 데이터를 1차원으로 선형화하여 메모리에 적재한 후에 데이터를 학습, 검증 및 평가 용도로 분할하여 사용하였다. 총 17명 환자 데이터 중 6명(P1, P2, P10, P14, P15, P17)의 데이터를 학습과 검증에 활용하였으며, 각각 8:2 비율로 분할하였다. 나머지 11명의 환자 데이터는 평가에 사용하였다.
모델 학습에서 학습률을 1e-4로 설정했고, 손실 함수는 평균 제곱 오차(Root Mean Square Error, RMSE) 및 최적화는 Huber 함수를 사용했다.
학습은 512 배치 크기로 최대 100 에포크 동안 진행하였으며, 3 에포크 동안 검증 데이터의 손실값에 변화가 없을 때 학습을 조기 종료하도록 하였다.
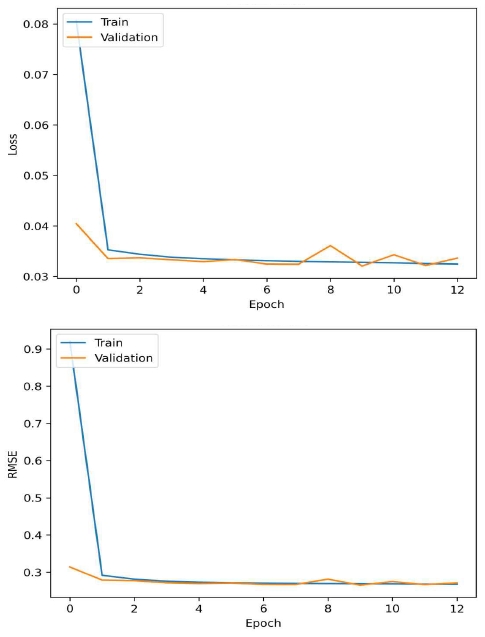
표 1에는 각 에포크 별로 OEF 예측의 RMSE 정확도와 손실률을 정리하였다. 또한, 그림 3에는 GRU 모델을 학습 및 검증 데이터를 사용하여 훈련하는 과정에서 손실 및 RMSE 경향을 도시하였다.
표와 그림에 따르면 RMSE와 손실률이 에포크가 진행됨에 따라 감소하는 경향을 보인다. 13 에포크 시점에서 모델의 학습이 조기 종료되었으며 이때의 손실률은 0.0325로 가장 낮았고, RMSE 또한 0.2676로 가장 높은 정확도를 보였다.
이 결과는 DNN 기반 딥러닝 모델과 비교했을 때 0.0013 더 낮은 손실률과 0.00802 더 낮은 RMSE 값에 해당하여 GRU 모델이 학습을 더 잘 수행한 것을 의미한다.
4.2 평가 정확도
제안하는 GRU 모델의 성능 평가는 학습과 검증에 사용되지 않은 11명의 환자 데이터를 이용해 진행하였다. 이 평가에서는 모델이 예측한 OEF 값의 정확도를 평가하기 위해 Ground Truth와의 평균 RMSE를 계산하였다. GRU 모델의 출력은 QQ 모델의 5개 매개변수, 즉 정맥 산소포화도를 포함한 OEF 예측에 필요한 변수들로 구성되어 있다. 따라서, 모델의 성능을 평가하기 위해서 모델의 출력과 Ground Truth 데이터를 비교하였다.
표 2와 표3에는 각각 DNN 기반 딥러닝 모델[11]과 본 논문에서 제안하는 GRU 기반 딥러닝 모델에 대해 Ground Truth 데이터 간의 RMSE 값을 정리하였다. 이를 통해 기존의 DNN 기반 모델보다 GRU 기반 모델이 에코 타임 0에서의 신호 강도 S0, BOLD 효과가 없는 조직의 가로 이완 R2, 정맥 산소포화도 Y, 비혈류 물질의 자화율 χnb에 대해서는 DNN 모델보다 평균적으로 높은 정확도로 예측했으나, 정맥 혈액량 v에 대해서는 약 0.001정도 낮은 정확도로 예측한 것을 확인할 수 있다. 이는 향후 모델의 구성을 변경하거나 파라미터 조정을 통해 해결할 수 있을 것으로 보인다.
표 4에는 11명의 환자 데이터에 대하여 실제와 예측한 OEF 차이에 대한 DNN 모델의 RMSE와 본 논문에서 제안하는 GRU 모델의 RMSE를 비교하였다. 일부 환자 데이터에 대해서는 DNN 모델에 비해 GRU 모델의 RMSE가 높게 나타났지만, 평균으로 비교해 보았을 때 GRU 모델의 RMSE가 DNN 모델의 RMSE보다 약 0.61 낮게 나타나서 정확도가 향상된 것을 알 수 있다.
그림 4에 Ground Truth 데이터와 DNN 모델을 이용하여 예측한 OEF, GRU 모델을 이용하여 예측한 OEF의 비교 결과를 도시하였고, 예측된 OEF가 Ground Truth와 상당히 일치함을 알 수 있다.
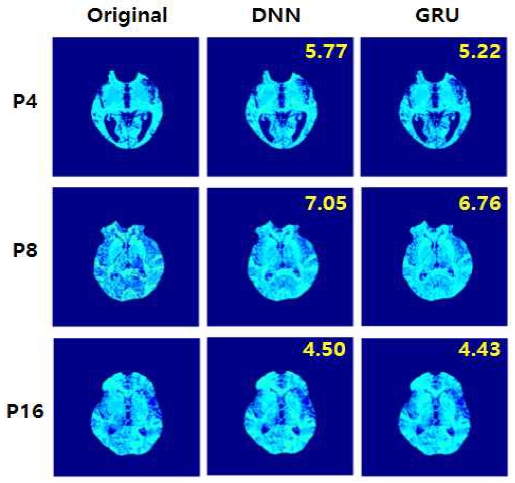
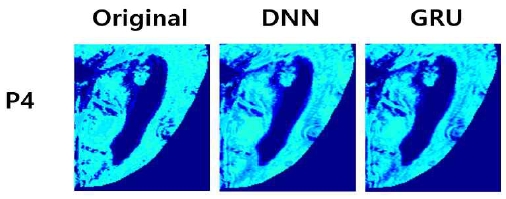
P4 환자의 OEF 예측 결과를 확대하여 그림 5에 나타내었다. 시각적으로 확인해보았을 때도 DNN 모델을 이용한 OEF 예측 결과와 비교하여 GRU 모델을 이용한 OEF 예측 결과가 Ground Truth와 더 유사하며, 선명하게 나타난 것을 확인할 수 있다.
표 5는 기존의 CCM 방법[7], CAT 방법[8], DNN 기반 딥러닝 방법[11] 및 제안한 방법들에 대하여 OEF 예측에 대한 평균 RMSE를 비교하고 있다. 동일한 코넬대학교 의과대학 데이터셋을 사용하여 비교를 수행하였으며, 비록 학습 및 평가에 사용된 환자의 번호에는 차이가 있지만, 제안한 방법이 높은 수준의 OEF 예측 정확도 즉 낮은 RMSE를 달성했다는 것을 확인할 수 있다.
이와 같은 실험 결과를 통하여 뇌 MRI 데이터에 대해 QQ 모델을 이용하고 이를 바탕으로 GRU 기반의 딥러닝 모델을 적용하여 OEF를 효율적으로 계산할 수 있음을 알 수 있다. 또한, 제안한 방법은 기존의 CCM 방법[7], CAT 방법[8], DNN 방법[11]에 비해 더 높은 정확도를 달성할 수 있음을 확인할 수 있다. 이는 제안된 GRU 모델이 뇌 MRI 신호 분석에 있어서 기존 방법들보다 우수한 성능을 제공할 수 있음을 의미한다.
Ⅴ. 결론 및 향후 과제
뇌 질환을 조기에 감지하기 위하여 뇌 MRI를 사용하여 뇌 조직의 OEF를 계산하는 것은 중요하며, 본 논문에서는 QQ 모델을 기반으로 GRU 모델을 구성하여 뇌 MRI를 통해 수집한 8개 신호 데이터를 통해 QQ 모델의 매개변수를 추정하고 OEF 예측 정확도를 향상하는 방법을 제안하였다. 또한, 총 17명 환자의 신호 감쇄 및 자화율을 이용하여 제안한 모델에 대하여 성능 분석을 수행하였다.
본 연구에서 제안한 GRU 모델은 뇌 MRI 데이터의 시계열적 특성을 효과적으로 활용하여 OEF를 예측하여 높은 정확도를 보였다. 이는 기존의 DNN 모델과 비교했을 때 특히 에코 타임 0에서의 신호 강도 S0, BOLD 효과가 없는 조직의 가로 이완 R2, 정맥 산소포화도 Y에서 뚜렷한 개선을 보여주었다.
향후 연구에서는 정맥 혈액량 v와 비혈류 물질의 자화율 χnb에 대한 예측 정확도를 향상시키기 위한 모델의 구조나 파라미터 조정이 필요할 것으로 보인다. 또한, 더 다양한 데이터셋에서 활용이 가능하도록 모델 구조를 일반화하는 것이 필요하다.
Acknowledgments
This work was supported by the National Research Foundation of Korea(NRF) grant funded by the Korea government(MSIT) (No. RS-2023-00242116)
References
-
H. Noh, J. Seo, S. Lee, N. Yi, S. Park, Y. J. Choi, and S. H, "Cause-of-death statistics in 2020 in the Republic of Korea", Journal of the Korean Medical Association, Vol. 66, No. 2, pp. 132-142, Feb. 2023.
[https://doi.org/10.5124/jkma.2023.66.2.132]

-
D. S. Bolar, B. R. Rosen, A. Sorensen, and E. Adalsteinsson, "Quantitative imaging of extraction of oxygen and tissue consumption(QUIXOTIC) using venular-targeted velocity-selective spin labeling", Magnetic Resonance in Medicine, Vol. 66, No. 6, pp. 1550-1562, Jun. 2011.
[https://doi.org/10.1002/mrm.22946]

-
R. G. Wise, A. D. Harris, A. J. Stone, and K. Murphy, "Measurement of OEF and absolute CMRO2: MRI-based methods using interleaved and combined hypercapnia and hyperoxia", NeuroImage, Vol. 83, pp. 135-147, Jun. 2013.
[https://doi.org/10.1016/j.neuroimage.2013.06.008]

-
X. He and D. A. Yablonskiy, "Quantitative BOLD: mapping of human cerebral deoxygenated blood volume and oxygen extraction fraction: default state", Magnetic Resonance in Medicine, Vol. 57, No. 1, pp. 115-126, Dec. 2006.
[https://doi.org/10.1002/mrm.21108]

-
D. Jiang and H. Lu, "Cerebral oxygen extraction fraction MRI: Techniques and applications", Magnetic Resonance in Medicine, Vol. 88, No. 2, pp. 575-600, Aug. 2022.
[https://doi.org/10.1002/mrm.29272]

-
J. Cho, et al., "Cerebral metabolic rate of oxygen(CMRO2) mapping by combining quantitative susceptibility mapping(QSM) and quantitative blood oxygenation level-dependent imaging(qBOLD)", Magnetic Resonance in Medicine, Vol. 80, No. 4, pp. 1595-1604, Mar. 2018.
[https://doi.org/10.1002/mrm.27135]

-
H.-Y. Lee, "OEF Calculation Method through Cluster-based Classification and Matching of QQ Model on Brain MRI Images", Journal of KIIT, Vol. 18, No. 10, pp. 15-23, Oct. 2020.
[https://doi.org/10.14801/jkiit.2020.18.10.15]

-
J. Cho, S. Zhang, Y. Kee, P. Spincemaille, T. D. Nguyen, S. Hubertus, A. Gupta, and Y. Wang, "Cluster analysis of time evolution(CAT) for quantitative susceptibility mapping(QSM) and quantitative blood oxygen level‐dependent magnitude(qBOLD)‐based oxygen extraction fraction(OEF) and cerebral metabolic rate of oxygen(CMRO2) mapping", Magnetic Resonance in Medicine, Vol. 83, pp. 844-857, Sep. 2019.
[https://doi.org/10.1002/mrm.27967]

-
J. Cho, et al., "QQ-NET - using deep learning to solve quantitative susceptibility mapping and quantitative blood oxygen level dependent magnitude (QSM+qBOLD or QQ) based oxygen extraction fraction(OEF) mapping", Magnetic Resonance in Medicine, Vol. 87, No. 3, pp. 1583-1594, Oct. 2021.
[https://doi.org/10.1002/mrm.29057]

-
S. Hubertus, S. Thomas, J. Cho, S. Zhang, Y. Wang, and L. R. Schad, "Using an artificial neural network for fast mapping of the oxygen extraction fraction with combined QSM and quantitative BOLD", Magnetic resonance in medicine, Vol. 82, No. 6, pp. 2199-2211, Jul. 2019.
[https://doi.org/10.1002/mrm.27882]

-
H. S. Kim and H.-Y. Lee, "DNN-based Oxygen Extraction Fraction Prediction Method for QQ Model of Brain MRI", Journal of KIIT, Vol. 21, No. 6, pp. 35-41, Jun. 2023.
[https://doi.org/10.14801/jkiit.2023.21.6.35]

- J. Oh and S. Kum, "A Study on Estimating Geomagnetic Azimuth using LSTM", The Journal of The Institute of Internet, Broadcasting and Communication, Vol. 22, No. 6, pp. 137-141, Dec. 2022.
- S.-M. Lee, Y.-G. Sun, J. Lee, D. Lee, E.-I. Cho, D.-H. Park, Y.-B. Kim, I. Sim, and J.-Y. Kim, "Short-term Power Consumption Forecasting Based on IoT Power Meter with LSTM and GRU Deep Learning", The Journal of The Institute of Internet, Broadcasting and Communication, Vol. 19, No. 5, pp. 79-85, Oct. 2019.

2021년 3월 ~ 현재 : 국립금오공과대학교 컴퓨터소프트웨어공학과 학사과정
관심분야 : 컴퓨터비전, 딥러닝

2021년 3월 ~ 현재 : 국립금오공과대학교 컴퓨터소프트웨어공학과 학사과정
관심분야 : 컴퓨터비전, 딥러닝

1997년 : 성균관대학교 정보공학과(공학사)
1999년 : KAIST 전산학과(공학석사)
2006년 : KAIST 전자전산학과(공학박사)
2008년 ~ 현재 : 국립금오공과대학교 컴퓨터소프트웨어공학과 교수
관심분야 : Digital Forensics, Image Processing, Computer Vision, Internet of Things