멀티모달 데이터를 활용한 멀티 헤드 크로스 어텐션 기반 피부 병변 분류 모델
초록
본 논문에서는 멀티모달 데이터를 활용한 피부 병변 분류 모델을 제안한다. 피부 병변의 정확한 진단을 위해 병변 이미지, 병변 마스크 이미지, 메타데이터를 결합하고 멀티 헤드 크로스 어텐션 메커니즘을 적용하였다. ISIC(International Skin Imaging Collaboration) 데이터셋에서 추출한 26,526개의 데이터와 5개의 피부 병변 클래스를 활용하여 모델 학습과 분류 실험을 수행하였다. 제안된 모델은 단일 모델 대비 정확도에서 2.5%p, F1-Score에서 2.7%p 향상된 성능을 보였다. 또한, ROC(Receiver Operating Characteristic) 커브 분석 결과, 제안된 모델은 각 클래스의 AUC(Area Under the Curve)의 평균값은 0.98 이상으로, 피부 병변 분류에서 효과적임을 확인하였다. 이는 멀티모달 데이터를 결합하여 보다 정확한 피부 병변 분류가 가능함을 시사한다.
Abstract
In this paper, we propose a skin lesion classification model utilizing multimodal data. For accurate diagnosis of skin lesions, we combined lesion images, lesion mask images, and metadata, and applied a multi-head cross-attention mechanism. We conducted model training and classification experiments using 26,526 data points and 5 skin lesion classes compiled from the International Skin Imaging Collaboration dataset. Our proposed model showed an improvement of 2.5 percentage points in accuracy and 2.7 percentage points in F1-Score compared to single models. Additionally, analysis of the Receiver Operating Characteristic curve indicated that the proposed model achieved an average Area Under the Curve value of over 0.98 for each class, confirming its effectiveness in skin lesion classification. This suggests that more accurate skin lesion classification is possible by combining multimodal data.
Keywords:
multimodal data, skin Lesion classification, multi-head cross attention, medical image segmentationⅠ. 서 론
피부 병변은 세포의 비정상적인 과증식이나 악성화로 인해 발생하며, 현대 사회에서 외상, 자외선 노출, 화학물질 접촉이 증가함에 따라 그 발병률 또한 증가하고 있다. 특히 기저세포암(Basal cell carcinoma)은 피부암 중 가장 흔한 유형으로, 전 세계에서 가장 흔한 악성 종양 중 하나이며 발생률이 계속 증가하고 있다[1]. 기저세포암은 남성 성별과 고령이 중요한 독립적인 위험 요소로 작용하며, 초기 발견 시 치료가 용이하지만, 초기 증상이 미미하거나 뚜렷하지 않아 종종 치료 시기를 놓치는 경우가 많다[1].
최근 디지털 이미지 처리와 딥러닝 기술의 발전은 피부암 진단 방법을 개선하고 있다. 특히, CNN(Convolutional Neural Network)은 이미지 특징을 추출하고 분류하는 데 유용하여, 의료 분야에서도 널리 사용되고 있다[2][3]. 이러한 기술을 통해 분류와 분할을 수행하여, 진단의 정확성을 높이는 연구가 진행되고 있다.
그러나 기존의 피부 병변 분류 및 진단에는 여전히 많은 도전 과제가 존재한다. 첫째, 모델 학습을 위해서는 방대한 양의 데이터셋이 필요한데, 기존의 데이터셋은 피부 질환 이미지가 부족하고, 질환별로 클래스 불균형이 심각하다[4]. 둘째, 피부확대경검사 이미지에서는 환자의 피부에 있는 털, 피부 색상, 혈관, 밝기와 대비 차이 등의 외부적 요인들이 존재하여 정확한 분류를 방해할 수 있다[5]. 셋째, 기존의 단일 데이터 기반 분류 모델들은 데이터셋의 편향된 특성에 취약하기 때문에, 다양한 형태의 피부 병변을 정확하게 분류하는 데 한계가 있다[6].
이러한 문제를 해결하기 위해 최근 다양한 데이터셋이 구축되고 있다. 대표적으로 ISIC(International Skin Imaging Collaboration) 데이터셋[7]-[9]은 피부 병변 분류를 위한 데이터셋으로, 다양한 피부 병변 이미지와 클래스로 구성되어 있다. 이 데이터셋은 다양한 크기와 클래스 비율로 제공되며, 피부 병변 분류 및 진단의 정확성을 높이는 것을 목표로 한다.
그러나 ISIC 데이터셋을 활용한 기존 연구들은 주로 병변 이미지만을 사용하여 분류 모델을 학습하였기 때문에, 메타데이터와 같은 추가 정보를 활용하지 못하는 한계점이 있다. 또한, 병변 영역 추출을 위한 분할 정보를 활용하지 않아 병변의 정확한 위치와 형태를 반영하기 어렵다는 문제점이 존재한다.
본 논문에서는 이러한 한계점을 극복하고자 멀티모달 데이터를 활용한 피부 병변 분류 모델을 제안한다. 제안하는 모델은 병변 이미지뿐만 아니라 메타데이터와 병변 마스크 이미지를 함께 사용하여 분류 성능을 높인다. 구체적으로, 이미지 특징 추출 과정에서 CNN을 인코더로 사용하여 병변 이미지와 병변 마스크 이미지로부터 각각 특징을 추출한 후, 멀티 헤드 크로스 어텐션(Multi-Head cross attention) 메커니즘[10]을 이용해 이미지 특징과 메타데이터를 결합한다. 이를 통해 병변의 시각적 특징뿐만 아니라 위치 및 형태 정보, 그리고 환자의 나이, 성별 등의 추가 정보를 모두 활용하여 정확한 분류가 가능하도록 한다.
제안하는 모델의 우수성을 입증하기 위해 ISIC 데이터셋을 활용한 실험을 수행하였다. 실험 결과, 제안된 모델이 병변 이미지만을 사용한 기존 모델들에 비해 우수한 분류 성능을 보임을 확인하였다. 이는 멀티모달 데이터의 활용이 피부 병변 분류 문제에서 효과적임을 시사한다.
본 논문의 구성은 다음과 같다. 2장에서는 피부 병변 분류와 관련된 기존 연구를 소개한다. 3장에서는 제안하는 멀티모달 데이터 기반 피부 병변 분류 모델의 구조와 학습 방법에 대해 자세히 설명한다. 4장에서는 ISIC 데이터셋을 활용한 실험 결과를 분석하고, 5장에서는 결론 및 향후 연구 방향에 대해 논의한다.
Ⅱ. 관련 연구
피부 질환 영상 분류는 CNN[11]을 활용한 이미지 분류 연구의 중요한 응용 분야로 자리 잡고 있다. 그러나, 피부 병변 분류를 위한 CNN 모델에서 단순 모델 학습만으로는 데이터의 불균형 문제로 인해 높은 분류 성능을 얻기 어려웠다. 이러한 문제를 해결하기 위해 다양한 접근 방식이 연구되었다.
J. Zhang et al.[12]은 의료 이미지 분류를 위한 시너지 딥러닝(SDL, Synergic Deep Learning) 모델을 제안하였다. 이 모델은 여러 개의 DCNN(Deep Convolutional Neural Network)을 동시에 사용하여 서로 상호 학습하도록 하여, 이미지 표현을 시너지 네트워크에 결합하여 입력으로 사용한다. 이를 통해 단일 모델보다 우수한 성능을 달성하였으나, 여러 개의 DCNN을 동시에 사용하여 복잡성이 증가해 모델의 훈련과 튜닝이 어려워질 수 있다는 한계점이 있다. 또한, 대규모 데이터셋이 없는 경우 과적합 문제가 발생할 수 있으며, 각 네트워크 간의 상호작용을 최적화하기 위한 추가적인 매개변수와 계산 비용이 요구된다.
M. H. Kwak et al.[13]는 동물 피부 병변 분류를 위해 멀티스케일 어텐션 메커니즘과 심층 앙상블 네트워크를 결합한 조인트 앙상블 방법을 제안하였다. 이 방법은 여러 개의 CNN 모델을 병렬로 학습시킨 후, 각 모델의 출력을 어텐션 메커니즘으로 결합하여 최종 분류를 수행한다. 앙상블 방법은 일반적으로 단일 모델보다 우수한 성능을 보이지만, 개별 모델 간의 상호 의존성과 특성 선택의 복잡성이 증가할 수 있다는 한계점이 있다. 또한, 다양한 스케일의 피부 병변 이미지에 대한 특징을 효과적으로 통합하는 방법에 대한 추가 연구가 필요하다.
S. Benyahia et al.[14]는 다양한 형태의 피부 병변을 분류하기 위해 여러 가지 특징 추출 방법을 조사하였다. 이 연구에서는 17개의 사전 학습된 CNN 아키텍처를 특징 추출기로 사용하고, 24개의 머신러닝 분류 모델을 활용하여 피부 병변을 분류하였다. 그 결과, 앙상블 모델이 단일 모델보다 우수한 성능을 보였으며, 특히 InceptionV3와 DenseNet201 모델을 기반으로 한 앙상블 모델이 가장 높은 성능을 달성하였다. 그러나 다양한 딥러닝과 머신러닝 조합을 찾는 것은 특징 추출과 분류 단계에서 많은 계산 자원이 요구되며, 최적의 모델 조합을 찾기 위한 체계적인 실험 설계와 분석이 필요하다.
G. Cai et al.[15]는 ViT(Vision Transformer) 모델을 기반으로 피부 병변을 분류하기 위한 멀티모달 퓨전 프레임워크를 제안하였다. 이 연구에서는 ViT 모델을 백본으로 사용하여 이미지의 심층 특징을 추출하고, 메타데이터를 라벨로 간주하여 소프트 라벨 인코더(SLE)를 설계하였다. 그리고 디코더 부분에 메타데이터와 병변 이미지를 멀티 헤드 크로스 어텐션을 통해 퓨전 블록을 생성하여 최종 분류를 수행하였다. 그 결과, 메타데이터와 병변 이미지를 함께 활용한 멀티모달 접근 방식이 단일 모달 방식보다 우수한 성능을 보였다. 그러나, ViT 모델은 대규모 데이터셋 학습에 의존하기 때문에 데이터셋이 부족하거나 불완전한 경우 성능이 저하될 수 있으며, 모델 학습에 상당한 계산 비용이 요구된다는 한계점이 있다.
이와 같은 연구들은 CNN 기반의 피부 병변 분류 모델의 성능을 개선하기 위해 앙상블 기법, 멀티스케일 어텐션, 멀티모달 퓨전 등 다양한 접근 방식을 활용하였다. 그러나 대부분의 연구들이 병변 이미지만을 사용하거나, 메타데이터와 병변 마스크와 같은 추가 정보를 충분히 활용하지 못하였다는 한계점이 있다. 또한, 제한된 데이터셋에서 모델의 일반화 능력을 평가하기 어려웠으며, 실제 임상 현장에서의 활용 가능성에 대한 검증이 부족하였다.
본 연구에서는 이러한 한계점을 극복하기 위해 멀티모달 데이터를 활용한 피부 병변 분류 모델을 제안한다. 제안하는 모델은 병변 이미지, 병변 마스크, 메타데이터를 모두 활용하여 분류 성능을 높이고자 한다. 해당 데이터의 결합에는 멀티 헤드 크로스 어텐션 메커니즘을 활용하며, 이종 데이터 간의 상호 연관성을 학습한다. 이는 피부 병변의 시각적 특징뿐만 아니라 위치, 형태, 환자 정보 등을 종합적으로 고려한 분류하여, 서로 다른 데이터 소스 간의 상호작용을 효과적으로 학습한다는 점에서 기존 연구와 차별화된다. 또한, 실험을 통해 제안 모델의 일반화 능력을 평가하고, 모델의 실제 활용 가능성을 논의하고자 한다.
Ⅲ. 멀티모달 데이터 기반 피부 병변 분류 모델
본 연구에서는 멀티모달 데이터를 활용한 피부 병변 분류 모델을 제안한다. 제안하는 모델은 피부 병변 이미지뿐만 아니라 메타데이터와 피부 병변 마스크를 추가로 활용하여, 각 데이터의 특징 벡터를 멀티 헤드 크로스 어텐션 메커니즘을 통해 결합함으로써 피부 병변 이미지 분류 성능을 높이고자 한다.
3.1 데이터셋
본 연구에서는 ISIC 데이터셋을 활용하였다. ISIC 2017 데이터셋은 2,000개의 훈련 이미지와 3개의 클래스로 구성되며, ISIC 2018 데이터셋(HAM10000)은 10,015개의 이미지와 7개의 클래스로 구성된다. ISIC 2019 데이터셋은 25,331개의 이미지와 9개의 클래스로 이루어져 있으며, ISIC 2020 데이터셋은 33,126개의 훈련 이미지와 Melanoma에 대한 악성/양성 분류 및 7개의 세부 클래스로 구성된다[7]-[9].
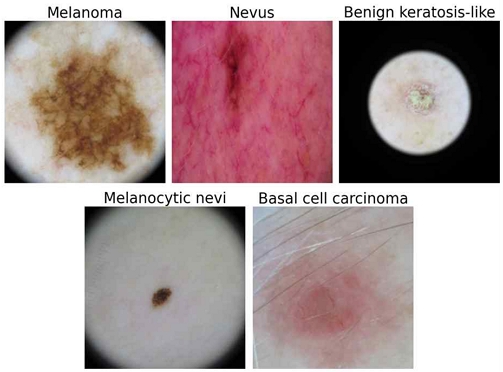
본 연구에서는 ISIC 2017, 2018, 2019, 2020 데이터셋을 통합하여 사용하였으며, 중복 이미지를 제거한 후 5개의 주요 클래스(Melanoma, Nevus, Basal cell carcinoma, Melanocytic nevi, Benign keratosis-like)에 대해 학습을 진행하였다. 피부 병변 분류에서 5개의 클래스를 선택한 이유는 다음과 같다. Melanoma는 전이되면 환자의 생명을 빠르게 위협하는 질환이다[16]. Melanoma의 33%는 Melanocytic nevi에서 직접 유래하며[17], Nevus과 Basal cell carcinoma을 가진 환자는 악성 종양이 발생할 확률이 높다[18][19]. Benign keratosis-like은 흑색종과 유사한 특징을 가지며, 숙련된 의사에게도 임상적으로 구분하기 어려운 경우가 있다[20]. 그림 1은 피부 병변 클래스별 예시 이미지를 보여주며, 각 클래스별 이미지 수는 표 1과 같다.
구축한 데이터셋의 메타데이터는 환자의 나이(Age), 성별(Sex), 병변 위치(Skin lesion location) 정보를 포함한다. 나이는 연속형 변수를 일정한 구간으로 나누어 범주형 변수로 변환하였으며, 병변 위치는 신체 부위에 따라 그룹화하였다. 메타데이터에 결측값이 있는 경우 해당 데이터를 제거하여 최종 데이터셋을 구성하였다. 이러한 메타데이터는 추가적인 진단 정보를 제공함으로써 모델 학습 시 분류 정확도 향상에 기여할 수 있다. 표 2는 메타데이터의 상세 구성을 보여준다.
3.2 제안하는 모델의 구조
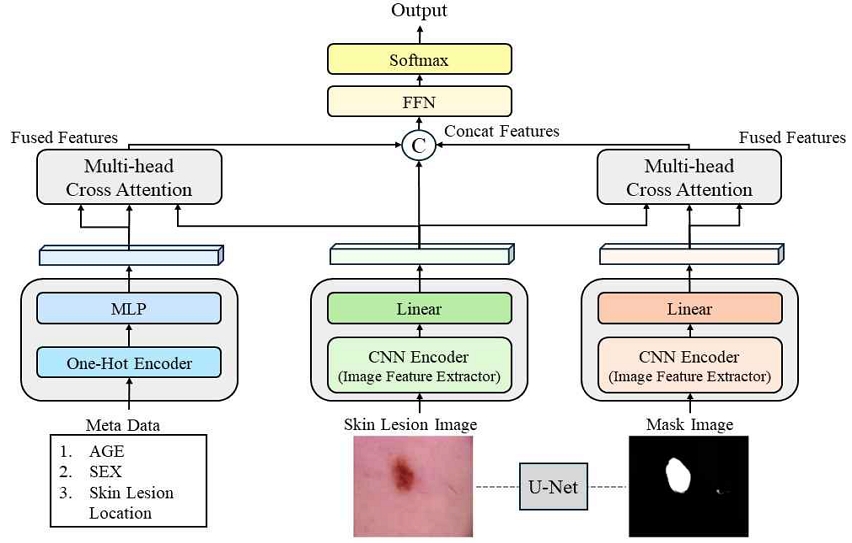
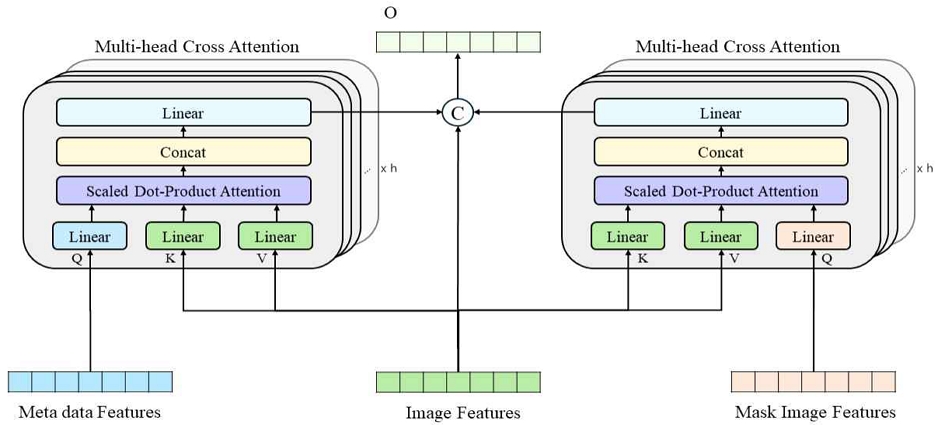
본 논문에서 제안하는 피부 병변 분류 모델은 멀티모달 데이터를 활용하여 피부 병변의 정확한 분류를 목표로 한다. 제안된 모델의 전체 구조는 그림 2와 같으며, 메타데이터, 병변 이미지, 병변 마스크 이미지를 입력으로 받아 피부 병변의 클래스를 출력한다.
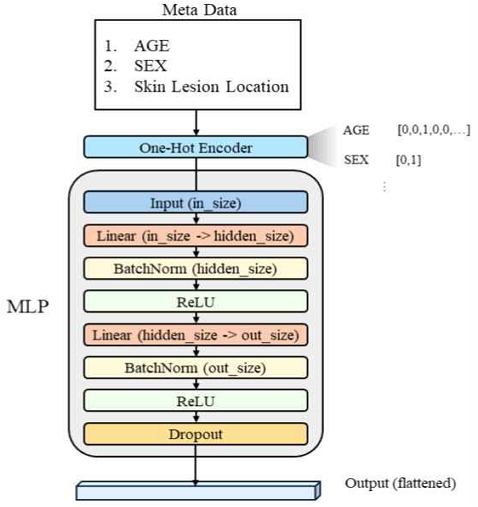
모델의 입력 데이터 중 메타데이터는 범주형 변수로 구성되어 있으므로, 이를 인코딩하기 위해 원-핫 인코더(One-hot encoder)를 사용한다. 원-핫 인코딩은 각 범주를 이진 벡터로 변환하여 서로 독립적인 차원을 가지도록 하는 기법이다. 인코딩된 메타데이터는 다층 퍼셉트론(MLP, Multilayer Perceptron) 네트워크를 통해 특징 벡터로 변환된다. MLP는 입력 차원을 특징 공간으로 매핑하고, 배치 정규화(Batch normalization)와 ReLU 활성화 함수를 적용하여 비선형 변환을 수행한다. 또한, 드롭아웃(Dropout)을 통해 과적합을 방지한다. 이를 통해 메타데이터는 최종적으로 고정된 크기의 특징 벡터로 변환되어 모델에 입력된다. 메타데이터 특징 추출을 위한 MLP 네트워크의 구조는 그림 3과 같다.
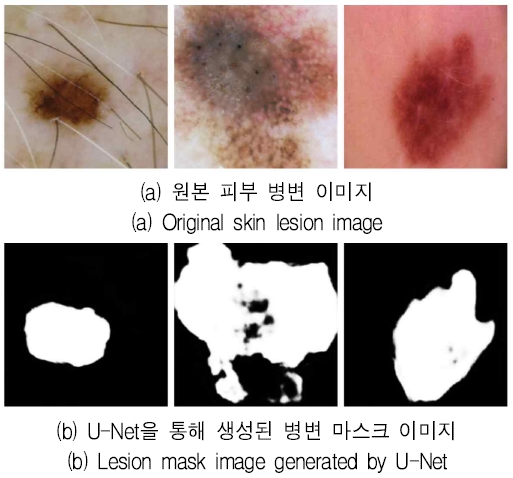
다음으로, 병변 이미지에 대한 병변 마스크 이미지를 생성한다. 병변 마스크는 이미지에서 병변 영역만을 분할하는 이진 마스크로, 병변의 위치와 형태에 대한 명시적인 정보를 제공한다. 본 연구에서는 HAM10000 데이터셋의 세그멘테이션 이미지[21]를 활용하여 U-Net[22] 모델을 학습하였으며, 이를 통해 병변 마스크 생성 모델을 구축하였다. U-Net 모델의 인코더로는 ResNet-34를 사용하였으며, Dice 계수를 손실 함수로 사용하였다. 입력 채널은 3(RGB 이미지), 출력 채널은 1(이진 마스크)로 설정하였다.
그림 4는 피부 병변 이미지(a)와 이에 대해 생성된 병변 마스크 이미지(b)의 예시를 보여준다. 병변 이미지에 대한 병변 마스크가 생성되고 나면, 사전 학습된 CNN 기반 백본 네트워크의 인코더 부분을 특징 추출기로 사용하여 각 이미지로부터 특징 벡터를 추출한다. 본 논문에서는 ImageNet[23] 데이터셋으로 사전 학습된 백본 네트워크를 인코더로 사용하였다. 백본 네트워크를 인코더로 사용하기 위해 마지막 완전 연결층(Fully connected layer)을 제거하여 이전 계층의 출력을 특징 벡터로 추출할 수 있게 하였다. 이를 통해 병변 이미지와 마스크 이미지는 각각 고정된 크기의 특징 벡터로 변환된다.
추출된 병변 이미지 특징 벡터와 병변 마스크 특징 벡터는 서로 다른 차원을 가지므로, 이를 통합하기 위해 크기를 조정한다. 이때, 선형 변환 계층(Linear layer)을 사용하여 두 특징 벡터의 차원을 동일하게 맞춘다. 이렇게 얻어진 병변 이미지 특징과 마스크 특징은 메타데이터 특징과 함께 멀티 헤드 크로스 어텐션 모듈의 입력으로 사용된다.
3.3 멀티 헤드 크로스 어텐션 메커니즘
추출된 병변 이미지 특징, 병변 마스크 특징, 메타데이터 특징은 멀티 헤드 크로스 어텐션 메커니즘을 통해 통합된다. 멀티 헤드 크로스 어텐션은 다양한 유형의 데이터 간의 상호 연관성을 학습할 수 있는 강력한 메커니즘으로, 서로 다른 도메인의 정보를 효과적으로 결합할 수 있다[10]. 이를 통해 병변의 시각적 특징뿐만 아니라 위치, 형태, 환자 정보 등을 종합적으로 고려하여 분류 성능을 높일 수 있다.
멀티 헤드 크로스 어텐션의 동작 과정은 다음과 같다. 먼저 세 가지 입력 특징 벡터 Fmeta(메타데이터), Fimg(병변 이미지), Fmask(병변 마스크)에 대해 각각 쿼리(Query), 키(Key), 값(Value)을 계산한다. 쿼리와 키는 특징 벡터의 유사도를 계산하는 데 사용되며, 값은 어텐션 결과를 생성하는 데 사용된다. 구체적으로, 메타데이터와 병변 마스크 특징 벡터로부터 쿼리 벡터 Qmeta와 Qmask를 생성하고, 병변 이미지 특징 벡터로부터 키 벡터 K와 값 V를 생성한다. 이때, WQmeta, WQmask, WKimg, WVimg는 학습 가능한 가중치 행렬이다.
| (1) |
| (2) |
| (3) |
| (4) |
다음으로, 쿼리와 키의 유사도를 계산하여 어텐션 맵(Attention map)을 생성한다. 어텐션 맵은 쿼리와 키 벡터 간의 내적을 통해 계산되며, 스케일링 팩터 로 나누어 정규화한다.
| (5) |
그 후, 소프트맥스(SoftMax) 함수를 적용하여 정규화한다. 정규화된 점수를 V에 곱하여 최종 어텐션 출력을 얻는다. 이 과정은 모델이 입력 데이터의 다양한 부분 간의 상호작용을 학습하여 더 정확한 특징을 추출할 수 있게 한다.
멀티 헤드 어텐션은 위의 과정을 여러 개의 어텐션 헤드로 병렬 처리한 후, 결과를 연결(Concatenate)하여 최종 출력을 생성한다. 각 어텐션 헤드는 독립적으로 학습되며, 서로 다른 특징 부분 공간에 주목할 수 있다.
| (6) |
| (7) |
멀티 헤드 크로스 어텐션을 통해 메타데이터와 병변 마스크 정보를 병변 이미지 특징에 반영한 결과는 다음과 같이 나타낼 수 있다.
| (8) |
| (9) |
최종적으로, 어텐션 결과와 원본 병변 이미지 특징을 연결하여 통합 특징 벡터를 생성한다.
| (10) |
생성된 통합 특징 벡터 Ffused features은 피부 병변의 시각적 특징, 위치 및 형태 정보, 환자의 메타데이터를 모두 포함하고 있다. Ffused features는 최종 분류를 위해 FFN(Feed-Forward Network)에 입력된다. FFN은 여러 개의 완전 연결층으로 구성되며, 활성화 함수로는 ReLU를 사용한다. FFN의 마지막 계층에서는 소프트맥스 함수를 적용하여 각 피부 병변 클래스에 대한 확률값을 출력한다. 최종적으로 확률값이 가장 높은 클래스를 출력함으로써, 5개의 클래스에 대한 피부 병변 분류 결과를 도출한다.
Ⅳ. 실험 및 성능 평가
4.1 실험 환경
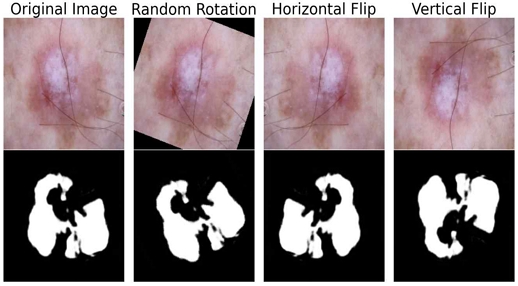
본 논문에서는 ISIC 2017, 2018, 2019, 2020 데이터셋을 통합하여 실험을 진행하였다. 데이터셋에서 중복된 이미지를 제거하고, 메타데이터에 결측값이 있는 경우 해당 데이터를 제외하였다. 구성된 데이터셋은 학습 데이터와 검증 데이터로 7:3 비율로 분할하였다. 클래스 간 샘플 수 불균형 문제를 해결하기 위해 데이터 증강을 통한 클래스 균형 샘플링 방법을 적용하였으며, 랜덤 회전 (Random rotation), 수평 반전 (Horizontal flip), 수직 반전 (Vertical flip) 등을 활용하였다. 이를 통해 모델의 일반화 성능을 향상하고, 피부 병변 분류의 정확도를 높이고자 하였다. 그림 6은 증강된 이미지 예시이다.
모델 학습 시, ImageNet 데이터셋으로 사전 학습된 EfficientNet B3 모델의 가중치를 초기값으로 사용하는 전이 학습(Transfer learning) 기법을 적용하였다. 학습 과정에서 배치 크기(Batch size)는 64로 설정하였으며, 입력 이미지의 크기는 224x224로 조정하였다. 최적화 알고리즘으로는 AdamW를 사용하였고, 초기 학습률(Learning rate)은 0.001로 설정하여 총 50 에포크(Epoch) 동안 학습을 진행하였다. 또한, 학습률 스케줄러로 ReduceLROnPlateau를 사용하여 학습이 진행됨에 따라 학습률을 적응적으로 조정하였다. 모델의 손실 함수로는 크로스 엔트로피(Cross entropy) 함수를 사용하였다. 멀티 헤드 크로스 어텐션의 헤드 수는 8로 설정하였다. 실험에 사용된 하드웨어 및 소프트웨어 환경은 표 3과 같다.
4.2 모델 성능 평가
제안된 모델의 성능을 평가하기 위해 다양한 실험을 진행하였다. 먼저, EfficientNet B3를 포함한 여러 사전 학습 모델을 백본 네트워크로 사용하여 단일 모델의 성능을 비교하였다. 이후, 선정된 백본 네트워크를 기반으로 멀티모달 데이터와 멀티 헤드 크로스 어텐션의 효과를 검증하였다. 모델의 성능은 정확도(Accuracy), 정밀도(Precision), 재현율(Recall), 그리고 정밀도와 재현율의 조화 평균인 F1-Score를 통해 평가하였다. 각 평가 지표는 아래와 같이 정의된다. 이때, TP(True Positive)는 실제 양성 클래스를 양성으로 예측한 수, TN(True Negative)는 실제 음성 클래스를 음성으로 예측한 수, FP(False Positive)는 실제 음성 클래스를 양성으로 예측한 수, FN(False Negative)는 실제 양성 클래스를 음성으로 예측한 수를 나타낸다.
| (11) |
| (12) |
| (13) |
| (14) |
먼저, 다양한 사전 학습 모델을 백본 네트워크로 사용하여 단일 모델의 성능을 비교하였다. 실험에는 VGG16[24], ResNet50[25], EfficientNet B3[26] 모델을 사용하였으며, 각 모델은 ImageNet 데이터셋으로 사전 학습된 가중치로 초기화하였다. 표 4는 각 모델의 성능 평가 결과를 보여준다. 실험 결과, EfficientNet B3 모델이 가장 높은 성능을 보였으며, 정확도 86.9%, F1-Score 86.8%를 달성하였다. 이는 EfficientNet 모델의 높은 표현력과 효율적인 구조에 기인한 것으로 분석된다. 따라서, 이후 실험에서는 EfficientNet B3 모델을 백본 네트워크로 선정하여 사용하였다.
제안 모델의 성능을 평가하기 위해 단일 이미지 데이터, 멀티모달 데이터, 병변 마스크를 사용하여 실험을 진행하였다. 제한 모델의 성능 평가를 위해 EfficientNet B3를 기반으로 특징 벡터를 결합한 모델과 멀티 헤드 크로스 어텐션을 적용한 모델의 성능을 비교했다. "EfficientNet B3 + Combine"은 특징 벡터를 쌓아 결합한 방법이며, "EfficientNet B3 + Multi-head Cross Attention Combine"은 멀티 헤드 크로스 어텐션 메커니즘을 통해 특징 벡터를 생성하여 결합한 방법이다.
해당 방법에서는 메타데이터와 병변 마스크 이미지를 사용한 경우와 사용하지 않은 경우를 비교하여 성능을 평가하였다. "Proposed Model"은 제안된 모델로, EfficientNet B3를 기반으로 메타데이터와 병변 마스크 이미지를 모두 사용하여 멀티 헤드 크로스 어텐션 메커니즘을 활용해 특징 벡터를 생성한 뒤, 결합한 방법이다.
제안된 모델은 89.4%의 정확도, 89.5%의 F1-Score를 달성하며, 다른 방법들과 비교했을 때 높은 성능을 보였다. 제안된 모델은 단일 모델 성능의 정확도에서 2.5%p, F1-Score에서 2.7%p 향상된 결과를 보였다. 또한, "EfficientNet B3 + Combine" 방식을 비교했을 때, 제안된 모델은 정확도에서 1.4%p, F1-Score에서 1.6%p 향상된 성능을 나타내었다. 표 5는 성능 평가 결과에 대한 비교를 보여준다.
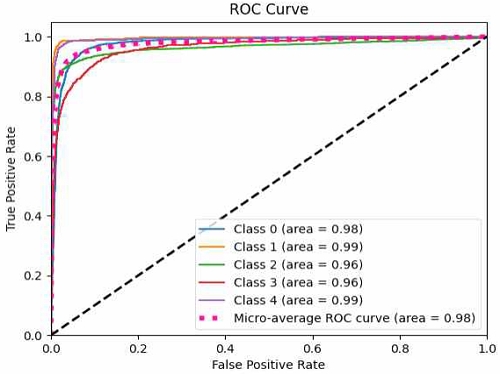
그림 7은 제안하는 모델의 ROC Curve(Receiver Operating Characteristic Curve)를 보여준다. ROC Curve는 분류 모델의 성능을 평가하는 데 사용되는 도구로, True Positive Rate와 False Positive Rate간의 상관관계를 시각화한다. ROC Curve가 좌상단 코너에 가까울수록 모델의 성능이 우수함을 나타낸다. 제안된 모델은 각 클래스의 AUC(Area Under Curve) 값이 평균적으로 0.98 이상으로 높은 수준을 보이고 있어, 제안된 모델이 피부 병변 분류에서 효과적임을 보여준다.
제안된 모델의 우수한 성능은 멀티모달 데이터 융합과 어텐션 메커니즘의 시너지 효과에 기인한 것으로 분석된다. 메타데이터는 환자의 나이, 성별 등 추가적인 정보를 제공하여 분류 성능 향상에 기여하였으며, 병변 마스크는 병변의 위치와 형태에 대한 정보를 제공함으로써 정확한 분류를 가능하게 하였다. 또한, 멀티 헤드 크로스 어텐션을 통해 이종 데이터 간의 상호 연관성을 학습함으로써 다양한 정보를 효과적으로 통합할 수 있었다. 따라서 멀티모달 데이터를 활용하고 어텐션 메커니즘을 통해 융합하는 것이 피부 병변 분류 문제에서 효과적인 접근 방식임을 시사한다.
Ⅴ. 결론 및 향후 과제
본 논문에서는 멀티모달 데이터를 활용한 피부 병변 분류 모델을 제안하였다. 제안된 모델은 병변 이미지, 병변 마스크 이미지, 메타데이터를 함께 사용하여 멀티 헤드 크로스 어텐션(Multi-head cross attention) 메커니즘을 통해 다양한 입력 데이터 간의 상호작용을 학습하고 결합하였다.
이를 통해 병변의 위치와 형태를 정확하게 반영하여 분류의 정확성을 높였다. ISIC 2017, 2018, 2019, 2020 데이터셋을 활용한 실험에서 제안된 모델은 병변 이미지, 병변 마스크 이미지, 메타데이터를 모두 사용한 경우 가장 높은 성능을 나타냈다. 또한, 멀티모달 데이터의 특징 추출 과정에서 멀티 헤드 크로스 어텐션을 사용한 방법과의 비교를 통해 성능 향상을 확인하였다. 이는 멀티모달 데이터를 통합하여 피부 병변 분류에서 성능 향상이 가능함과 멀티 헤드 크로스 어텐션 방법이 데이터 간의 상호작용을 효과적으로 학습 가능하다는 것을 시사한다.
제안된 모델은 병변 이미지와 병변 마스크 이미지를 활용하여 분류 성능을 향상하였으나, 몇 가지 한계점이 존재한다. 첫째, 병변 마스크의 품질에 따른 모델의 성능 측정이 필요하다. 향후 연구에서는 병변 마스크 생성 모델의 고도화를 통해 더욱 정밀한 마스크 이미지를 생성하고, 다양한 분할 모델을 적용할 수 있다. 둘째, 특징 벡터의 결합 과정에 대한 개선이 필요하다. 현재는 멀티헤드 크로스 어텐션을 통해 얻은 특징 벡터를 쌓아 결합하였지만, 특징 벡터의 결합 과정에서 더 정교한 결합 방법을 적용함으로써 성능 향상을 도모할 수 있다.
본 논문에서 제안된 멀티모달 데이터 기반 피부 병변 분류 모델은 다양한 입력 소스를 통합하여 피부 병변 분류의 성능을 향상하는 데 기여하였다. 실험 결과, 단일 모델 대비 높은 성능을 보였으며, 향후 연구를 통해 모델의 성능을 더욱 향상할 수 있는 가능성을 확인하였다. 본 연구는 피부 병변 분류 분야에서 멀티모달 데이터 활용의 중요성을 강조하며, 향후에도 모델 고도화를 통한 멀티모달 분류 방식의 지속적인 발전이 이루어지기를 기대한다.
Acknowledgments
본 논문은 정부(교육부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임(No. 2021R1G1A1006381)
References
-
M. C. Cameron, E. Lee, B. P. Hibler, C. A. Barker, S. Mori, M. Cordova, K. S. Nehal, and A. M. Rossi, "Basal cell carcinoma: Epidemiology; pathophysiology; clinical and histological subtypes; and disease associations", Journal of the American Academy of Dermatology, Vol. 80, No. 2, pp. 303-317, Feb. 2019.
[https://doi.org/10.1016/j.jaad.2018.03.060]

-
T. C. Pham, C. M. Luong, M. Visani, and V. D. Hoang, "Deep CNN and data augmentation for skin lesion classification", Intelligent Information and Database Systems, Vol. 10, pp. 573-582, Feb. 2018.
[https://doi.org/10.1007/978-3-319-75420-8_54]

-
M. A. Albahar, "Skin lesion classification using convolutional neural network with novel regularizer", IEEE Access, Vol. 7, pp. 38306-38313, Mar. 2019.
[https://doi.org/10.1109/ACCESS.2019.2906241]

-
V. D. Nguyen, N. D. Bui, and H. K. Do, "Skin lesion classification on imbalanced data using deep learning with soft attention", Sensors, Vol. 22, No. 19, pp. 7530, Oct. 2022.
[https://doi.org/10.3390/s22197530]

-
M. H. Jafari, N. Karimi, E. Nasr-Esfahani, S. Samavi, S. M. R. Soroushmehr, K. Ward, and K. Najarian, "Skin lesion segmentation in clinical images using deep learning", 2016 23rd International Conference on Pattern Recognition (ICPR), pp. 337-342, Dec. 2016.
[https://doi.org/10.1109/ICPR.2016.7899656]

-
J. Zhang, Y. Xie, Q. Wu, and Y. Xia, "Skin lesion classification in dermoscopy images using synergic deep learning", Medical Image Computing and Computer Assisted Intervention–MICCAI 2018: 21st International Conference, Vol. 11, pp. 12-20, Sep. 2018.
[https://doi.org/10.1007/978-3-030-00934-2_2]

-
P. Tschandl, C. Rosendahl, and H. Kittler, "The HAM10000 dataset, a large collection of multi-source dermatoscopic images of common pigmented skin lesions", Scientific Data, Vol. 5, No. 1, Aug. 2018.
[https://doi.org/10.1038/sdata.2018.161]

- M. Combalia, et al., "BCN20000: Dermoscopic lesions in the wild", arXiv preprint arXiv:1908.02288, , Aug. 2019.
- International Skin Imaging Collaboration, "SIIM-ISIC 2020 Challenge Dataset", International Skin Imaging Collaboration, 2020.
- A. Vaswani, et al., "Attention is all you need", Advances in Neural Information Processing Systems, Vol. 30, pp. 6000-6010, Dec. 2017.
-
A. Krizhevsky, I. Sutskever, and G. E. Hinton, "ImageNet classification with deep convolutional neural networks", Communications of the ACM, Vol. 60, No. 6, pp. 84-90, Jun. 2017.
[https://doi.org/10.1145/3065386]

-
J. Zhang, Y. Xie, Q. Wu, and Y. Xia, "Medical image classification using synergic deep learning", Medical Image Analysis, Vol. 54, pp. 10-19, May 2019.
[https://doi.org/10.1016/j.media.2019.05.001]

- M. H. Kwak, K. T. Kim, and J. Y. Choi, "Multi-scale Attention and Deep Ensemble-Based Animal Skin Lesions Classification", Journal of Korea Multimedia Society, Vol. 25, No. 8, pp. 1212-1223, Aug. 2022.
-
S. Benyahia, B. Meftah, and O. Lézoray, "Multi-features extraction based on deep learning for skin lesion classification", Tissue and Cell, Vol. 74, pp. 101701, Feb. 2022.
[https://doi.org/10.1016/j.tice.2021.101701]

-
G. Cai, Y. Zhu, Y. Wu, X. Jiang, J. Ye, and D. Yang, "A multimodal transformer to fuse images and metadata for skin disease classification", The Visual Computer, Vol. 39, No. 7, pp. 2781-2793, May 2023.
[https://doi.org/10.1007/s00371-022-02492-4]

-
D. Schadendorf, A. C. J. V Akkooi, C. Berking, K. G. Griewank, R. Gutzmer, and A. Hauschild, "Melanoma", SEMINAR, Vol. 392, No. 10151, pp. 971-984, Sep. 2018.
[https://doi.org/10.1016/S0140-6736(18)31559-9]

-
W. Damsky and M. Bosenberg, "Melanocytic nevi and melanoma: unraveling a complex relationship", Oncogene, Vol. 36, pp. 5771-5792, Jun. 2017.
[https://doi.org/10.1038/onc.2017.189]

-
N. E. Thomas and P. Groben, "Invasive superficial spreading melanomas arising from clinically normal skin", Journal of the American Academy of Dermatology, Vol. 51, No. 3, pp. 466-470, Sep. 2004.
[https://doi.org/10.1016/j.jaad.2004.04.027]

-
C. S. M. Wong, R. C. Strange, and J. T. Lear, "Basal cell carcinoma", British Medical Journal, Vol. 327, No. 7418, pp. 794-798, Oct. 2003.
[https://doi.org/10.1136/bmj.327.7418.794]

-
J. Agata, O. Teresa, I. Michela, R. Marco, and D. Valentina, "Seborrheic keratosis-like melanoma: a diagnostic challenge", Melanoma Research, Vol. 31, No. 5, pp. 407-412, Oct. 2021.
[https://doi.org/10.1097/CMR.0000000000000756]

- Kaggle, "HAM10000 Lesion Segmentations", https://www.kaggle.com/datasets/tschandl/ham10000-lesion-segmentations, [accessed: Jun. 13, 2024]
-
O. Ronneberger, P. Fischer, and T. Brox, "U-net: Convolutional networks for biomedical image segmentation", Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, Vol. 18, pp. 234-241, Nov. 2015.
[https://doi.org/10.1007/978-3-319-24574-4_28]

-
J. Deng, W. Dong, R. Socher, L. J. Li, K. Li, and L. Fei-Fei, "Imagenet: A large-scale hierarchical image database", 2009 IEEE Conference on Computer Vision and Pattern Recognition, Miami, FL, USA, pp. 248-255, Jun. 2009.
[https://doi.org/10.1109/CVPR.2009.5206848]

- K. Simonyan and A. Zisserman, "Very deep convolutional networks for large-scale image recognition", arXiv preprint arXiv:1409.1556, , Sep. 2014.
-
K. He, X. Zhang, S. Ren, and J. Sun, "Deep residual learning for image recognition", 2016 IEEE Conference on Computer Vision and Pattern Recognition, Las Vegas, NV, USA, pp. 770-778, Jun. 2016.
[https://doi.org/10.1109/CVPR.2016.90]

- M. Tan and Q. Le, "Efficientnet: Rethinking model scaling for convolutional neural networks", International Conference on Machine Learning, Long Beach, California, USA, Vol. 97, pp. 6105-6114, Jun. 2019.

2020년 3월 ~ 현재 : 경상국립대학교 컴퓨터공학과 학사과정
관심분야 : 인공지능, 데이터분석, 컴퓨터 비전

2019년 3월 ~ 현재 : 경상국립대학교 컴퓨터공학과 학사과정
관심분야 : 인공지능, 데이터분석, 컴퓨터 비전

2021년 3월 ~ 현재 : 경상국립대학교 컴퓨터공학과 학사과정
관심분야 : 데이터분석, 이미지 처리, 인공지능

2021년 3월 ~ 현재 : 경상국립대학교 컴퓨터공학과 학사과정
관심분야 : 데이터분석, 이미지 처리, 인공지능

2010년 2월 : 광주과학기술원 정보기전공학부(공학석사)
2017년 8월 : 광주과학기술원 전기전자컴퓨터공학부(공학박사)
2018년 3월 ~ 현재 : 경상국립대학교 컴퓨터공학과 부교수
관심분야 : 컴퓨터 그래픽스, 증강현실, 인공지능

2006년 12월 : 호주뉴캐슬대학교 컴퓨터공학과(공학사)
2007년 9월 : 호주뉴캐슬대학교 정보공학과(공학석사)
2017년 8월 : 한양대학교 컴퓨터공학과(공학박사)
2021년 9월 ~ 현재 : 경상국립대학교 컴퓨터공학과 조교수
관심분야 : 인공지능, 시멘틱 헬스케어, 데이터마이닝