남성의 체성분 지표 기반 이상지질혈증을 식별하기 위한 기계학습 접근법
초록
이상지질혈증은 비만, 대사증후군 및 심혈관 질환 등의 발생 가능성을 높인다. 따라서 이상지질혈증의 식별을 용이하게 하는 도구의 개발은 예방과 조기 발견을 위해 중요하다. 본 연구에서는 체성분 지표를 기반으로 이상지질혈증을 식별하기 위한 모델을 개발하고 그 성능을 비교하였다. 남성 820명을 대상으로 기계학습 방법을 사용하여 6개의 모델을 생성한 후 AUROC 및 F1 점수 등의 모델 성능 평가 지표를 사용하여 모델의 성능을 평가하였다. 분석 결과, 익스트림 그래디언트 부스팅 방법을 사용한 모델이 높은 성능을 나타냈고 연령, 몸통 위상각, 체지방률, 골격근률이 이상지질형증 식별에 중요한 변수로 선택되었다. 이 결과는 이상지질혈증을 비침습적으로 식별하는 체성분 지표의 잠재력을 입증하였다. 기계학습 알고리즘을 통한 질환의 식별은 임상의가 통찰력을 얻고 의사 결정을 하는데 도움이 될 것이다.
Abstract
Dyslipidemia raises the risk of developing obesity, metabolic syndrome, and cardiovascular disease. Therefore, in order to prevent and detect dyslipidemia early, it is important to develop a model that can identify dyslipidemia. In this study, we developed the models to identify dyslipidemia based on body composition indices in men, and compared the performances of the models. Six models were developed using machine learning approach for 820 men, and assessed using evaluation methods such as areas under the receiver operating characteristic curve(AUROC), F1 score. The model using extreme gradient boosting reported the highest AUROC value (AUROC = 0.825 [95% CI, 0.742-0.893]), and age, phase angle of trunk, percent of body fat, and ratio of skeletal muscle mass to weight were selected as relatively important variables. Thees results demonstrated the potential of body composition indices to identify dyslipidemia noninvasively. Moreover, identifing dyslipidemia based on body composition indices could provide clinicians with valuable insights and help them make well-informed decisions.
Keywords:
dyslipidemia, machine learning, body composition, muscle massⅠ. 서 론
기계학습(Machine learning)은 인공지능의 한 영역으로 대규모 데이터를 기반으로 컴퓨터가 스스로 학습하여 특정 패턴을 가진 모델을 생성한다[1]. 기계학습은 학습하려는 문제의 유형에 따라 지도학습(Supervised learning), 비지도 학습(Unsupervised learning), 강화학습(Reinforcement learning) 등으로 구분되며 많은 기법들이 개발되어 왔다. 기계학습 기법들의 개발과 고성능 컴퓨팅이 가능해짐에 따라 기계학습은 생물정보학[2], 기상학[3], 양식업[4] 등 다양한 분야에서 사용되고 있다. 의료 분야에서도 기계학습은 꾸준히 사용되어 왔는데 질병을 탐지하기 위한 임상 보조도구로 활용될 수 있고 의사가 정확한 진단을 내릴 수 있도록 지원하는 역할을 할 수 있기 때문에 그 필요성이 증가하고 있다[5][6]. 의료 분야의 데이터는 방대한 양의 환자와 각 환자에 대한 방대한 양의 생체 변수 등이 수집된다는 특징이 있다[7][8]. 이러한 크고 복잡한 데이터를 통합하여 분석하기 위해서는 기계학습 접근법 사용이 필요하다[9].
이상지질혈증(Dyslipidemia)은 만성질환 중 하나로 콜레스테롤과 중성지방의 비정상적인 혈청 수치를 특징으로 한다. 이상지질혈증은 고저밀도지단백(High LDL, High Low Density Lipoprotein)-콜레스테롤혈증, 고중성지방혈증, 저고밀도지단백(Low HDL, Low high Density Lipoprotein)-콜레스테롤혈증 중 적어도 하나를 갖는 것으로 정의된다[10]. 이상지질혈증은 고혈압(Hypertension) 및 당뇨병(Diabetes mellitus)과 함께 심혈관 질환(Cardiovascular disease)의 위험을 증가시키는 것으로 알려져 있다[11]. 심혈관 질환은 전 세계적으로 사망의 주요 원인으로 알려져 있으며 WHO의 보고에 따르면 2019년에 전세계 사망자의 32%에 해당하는 약 1,790만명이 심혈관 질환으로 사망하였다[12]. 심혈관 질환의 선행 질환이자 위험인자인 이상지질혈증의 연령표준화 유병률은 한국지질동맥경화학회 보고에 따르면 2020년 45.4%로 나타났고 2016-2020년 동안의 유병률은 48.2%였다[10].
비만은 심혈관 질환, 이상지질혈증, 고혈압, 당뇨병, 암 등 여러 질환과 관련이 있는 것으로 알려져 있다[13]. 비만을 정의하는 체질량지수(BMI, Body Mass Index)에 의해 정상체중에 분류되는 대상자 중 실제로 과도한 지방량을 가지고 있는 경우 심혈관 질환이나 이상지질혈증 등 질환 발병 위험이 높을 수 있다. BMI 값이 동일하더라도 체지방률, 골격근률, 체수분율 등 체성분 지표값이 다를 수 있다. 따라서 비만과 관련된 질병 상태의 변화를 파악하기 위해 BMI만 측정하는 것은 적합하지 않을 수 있다[14][15]. BMI 뿐만아니라 체성분 지표는 이상지질혈증, 고혈압, 당뇨, 심혈관 질환 등과 높은 연관성이 있다[16][17]. 그러나 기계학습 기법을 사용하여 한국 남성을 대상으로 비침습적 방법인 체성분 지표 기반 이상지질혈증을 식별하기 위한 연구는 아직 수행되지 않고 있다.
따라서 본 연구에서는 기계학습 기법을 사용하여 한국 남성을 대상으로 체성분 지표 기반 이상지질혈증을 식별하기 위한 모델을 개발하고 그 성능을 비교하였다. 본 논문의 구성을 다음과 같다. 2장은 기계학습 기반 이상지질혈증 관련 질환 식별에 관한 기존 연구와 최근 연구 동향에 대해 살펴본다. 3장에서는 기계학습 기법을 사용한 체성분 지표 기반 이상지질혈증을 식별 모델 개발 방법에 대하여 기술한다. 4장에서는 체성분 지표 기반 이상지질혈증을 식별 모델의 성능을 평가하고 이상지질혈증 식별에 중요도가 높은 변수를 기술한다. 5장에서는 본 연구의 결론을 기술하고 연구의 제한점과 향후 과제를 논한다.
Ⅱ. 기계학습 기반 이상지질혈증 관련 질환 식별에 관한 기존 연구
이상지질혈증 뿐만아니라 이상지질혈증과 관련된 질환인 고콜레스테롤혈증, 대사증후군 등을 식별하기 위해 생체지표 기반 기계학습 접근법을 사용한 많은 연구가 진행되어 오고 있다.
이상지질혈증 식별에 관한 기존 연구로는 L. Zhang et al.[18]은 딥러닝을 사용하여 1,222개의 고품질 망막 이미지와 50개 이상의 인체 측정 및 생화학적 변수 기반 이상지질혈증 예측 모델에서 성능 값으로 0.703의 AUROC(Areas Under the Receiver Operating characteristic Curve) 값을 얻었다.
M. Correia et al.[19]은 기계학습 접근법 중 로지스틱 회귀분석을 사용하여 혈중 지질 바이오마커 기반 이상지질혈증 분류 모델에서 0.75-0.92의 AUROC 값을 얻었다. G. Gutierrez-Esparza et al.[20]는 랜덤 포레스트, 익스트림 그래디언트 부스팅, 그래디언트 부스팅 방법론을 사용하여 인체측정 변수, 생화학적 변수, 식이 섭취, 가족 건강 이력 및 흡연 습관, 음주, 수면의 질, 신체 활동 변수 기반 이상지질혈증 예측 모델을 개발하였다. 결과 랜덤 포레스트 모델이 80%의 정확도를 나타내며 가장 좋은 성능을 보였다. J. Liu et al.[21]은 이상지질혈증에 대한 분류 예측 모델을 구축하기 위해 딥러닝 방법을 사용했다. 가슴 답답함, 천명음, 진한 보라색 혀 등 총 89개의 전통 중의학 진단 요인 기반 이상지질혈증 분류 모델을 개발하였고 0.8672의 정확도, 0.7138의 정밀도, 0.9268의 AUROC의 성능을 보고하였다. S. Cui et al.[22]은 순환 신경망(RNN, Recurrent Neural Network)과 LSTM(Long Short-Term Memory) 신경망을 사용하여 철강근로자의 이상지질혈증 위험 예측 모델을 개발하였다. 연령, BMI, 근속 기간, 결혼 여부, 교육 수준, 경제 수준, 음주, 흡연, 작업장, 근무 유형, 교대 유형 및 작업 환경의 고온 및 소음에 대한 노출 기반 이상지질혈증 위험 예측 모델에서 LSTM 모델의 성능은 0.895, RNN 모델의 성능은 0.853의 AUROC 값을 나타냈다. A. Takhttavous et al.[23]은 로지스틱 회귀분석과 의사결정 나무 방법을 사용하여 이상지질혈증 발병률 예측 모델을 개발하였다. 인체 측정 지표인 내장 지방 지수, 허리 지수, 체질량 지수, 체지방 지수, 체표면적 등을 기반으로 한 이상지질혈증 예측 모델에서 로지스틱 회귀 모델은 0.63, 의사결정 나무 모델은 0.67의 AUROC 성능을 보고하였다.
고콜레스테롤혈증 분류에 대한 연구에서는 R. Hesse et al.[24]가 로지스틱 회귀, 딥 러닝, 랜덤 포레스트 분류를 결합하여 가족성 고콜레스테롤혈증 분류 모델을 개발하였다. 연령, 성별, 총콜레스테롤, 중성지방, LDL-콜레스테롤, HDL-콜레스테롤, 심혈관 질환 유무 등을 기반으로 한 가족성 고콜레스테롤혈증 분류 모델의 성능은 AUROC 값이 0.711값을 나타냈다. A. Pina et al.[25]은 의사결정 나무, 그래디언트 부스팅, 신경망 방법을 사용하여 가족성 고콜레스테롤혈증의 가상 유전 진단 모델을 생성하였다. 연령, 저밀도지단백-콜레스테롤, 중성지방, HDL-콜레스테롤 등을 기반으로 한 가족성 고콜레스테롤혈증의 가상 유전 진단 모델의 성능은 의사결정 나무 모델이 0.79, 래디언트 부스팅 모델이 0.83, 신경망 모델이 0.83의 AUROC 값을 보고하였다.
대사증후군 분류에 관한 연구에서는 H. Yang et al.[26]이 익스트림 그래디언트 부스팅과 랜덤 포레스트 기법을 사용하여 대사증후군 위험 예측 모델을 개발하였다. 건강검진 후 얻어진 지표인 연령, 중성지방, LDL-콜레스테롤, HDL-콜레스테롤, 이완기 혈압, 수축기 혈압, 허리둘레, 엉덩이둘레 등을 기반으로 대사증후군 위험 예측 모델을 구축한 결과 익스트림 그래디언트 부스팅 모델의 성능은 0.93, 랜덤 포레스트 모델의 성능은 0.916의 AUROC 값을 보고하였다. X. Deng et al.[27]은 체성분 지표를 기반 그래디언트 부스팅 알고리즘을 사용하여 대사성 건강한 정상체중, 대사성 건강한 비만, 대사성 건강에 해로운 정상체중, 및 대사성 건강에 해로운 비만 분류 모델을 개발하였다. 대사성 건강한 정상체중과 대사성 건강에 해로운 정상체중을 분류하는 모델의 성능은 0.842, 대사성 건강한 비만과 대사성 건강에 해로운 비만을 분류하는 모델의 성능은 0.746의 AUROC 값을 나타냈다. M. Akbarzadeh et al.[28]은 로지스틱 회귀분석, 랜덤 포레스트, 의사결정 나무, 서포트 벡터 머신을 사용하여 대사증후군을 예측 모델을 개발하였다. GCKR(Glucokinase regulator) 유전자 다형성 변수, 연령, 성별, 학력, BMI, 신체 활동 등의 인구통계학적 변수를 기반으로 한 대사증후군을 예측 모델에서 랜덤 포레스트 모델의 성능은 0.804, 로지스틱 회귀 모델의 성능은 0.77, 의사결정 나무 모델의 성능은 0.771, 서포트 벡터 머신 모델의 성능은 0.785의 AUROC 값을 나타내어 랜덤 포레스트 모델이 최고의 성능을 보였다. S. Y. Kim et al.[29]는 나이브 베이지안과 로지스틱 회귀분석 기법을 사용하여 대사증후군 식별 모델을 개발하였다. 인체 측정, 혈액 지표, 폐활량 측정 지표를 기반으로 개발된 대사증후군 식별 모델에서 나이브 베이지안 모델의 성능은 남성에서 0.83, 여성에서 0.908, 로지스틱 회귀 모델의 성능은 남성에서 0.863, 여성에서 0.932의 AUROC 값을 나타내어 로지스틱 회귀 모델이 다소 높은 성능을 보고하였다.
Ⅲ. 기계학습 기법을 사용한 체성분 지표 기반 이상지질혈증을 식별 방법
3.1 연구 대상자 선정
본 연구에서는 2022년 4월부터 2022년 12월까지 대한민국의 5개 한방병원(동국대학교 일산한방병원, 동신대학교 한방병원, 가천대학교 부속길한방병원, 부산대학교 한방병원, 세명대학교 부속한방병원)에서 만 20세 이상 성인을 대상으로 모집된 임상 연구 자료를 분석하였다. 각 기관별 600명씩 총 3000명이 임상 연구에 등록하였으나 포함기준을 충족하지 못한 대상자 및 중도 탈락자 총 14명을 제외한 2986명이 임상 연구를 종료하였다. 2986명에서 체성분 분석기 결과값에 오류가 있는 80명을 제외한 2906명(남성 820명, 여성 2086명) 중 남성 820명을 대상으로 분석하였다. 임상 연구는 각 기관의 임상시험 심사위원회의 승인을 받아 헬싱키 선언에 따라 수행되었다(IRB No., 동국대학교 일산한방병원, DUIOH-2022-01-005; 동신대학교 한방병원, NJ – IRB-013; 가천대학교 부속길한방병원, GIRB-22-101; 부산대학교 한방병원, PNUKHIRB-2022-02-001; 세명대학교 부속한방병원, SMJOH-2022-06).
3.2 체성분 지표 측정
체성분 지표 추출을 위해 체성분 분석기(BWA 2.0, InBody Co., Seoul, Republic of Korea)가 사용되었다. 체성분 측정은 잘 훈련받은 임상 연구 코디네이터에 의해 표준작업지짐서에 따라 수행되었다. BWA 2.0은 대상자의 체성분과 체수분을 정확하게 측정할 수 있도록 만들어진 의료용 검사기로 집게 모양의 전극을 통해 몸 상태를 측정한다. 집게를 손, 발목 복사뼈 부위에 집고 누워서 비침습적 방법으로 측정한다. 인체는 크게 체수분, 단백질, 무기질, 체지방의 네 가지 성분으로 구분되는데 체성분 검사는 인체의 구성 성분을 정량적으로 분석하여 기본 정보를 제공한다. 본 연구 모델에서 선택된 체성분 지표는 표 1에 설명되어 있다.
3.3 이상지질혈증군 정의
두 개의 질문인 “의사로부터 이상지질혈증 진단을 받았는가?”와 “현재 이상지질혈증이 지속되는가?”에 둘 다 “예”라고 대답한 대상자들은 이상지질혈증군으로 분류하였고 그 외 대상자들을 정상군으로 분류하였다.
3.4 통계분석
이상지질혈증군과 정상군간의 체성분 지표 비교를 위해 이표본 t 검정(Two-sample t test)를 사용되었다. 체성분 지표 기반 이상지질혈증 식별 모델을 구축하기 엘라스틱 넷(E-net, Elastic Net), 익스트림 그래디언트 부스팅(XGBoost, Extreme Gradient Boosting), 신경망(NN, Neural Network), 랜덤 포레스트(RF, Random Forest), k-최근접 이웃(KNN, k-Nearest Neighbor) 및 서포트 벡터 머신(SVM, Support Vector Machine) 방법이 사용되었다. 체성분 지표는 표준화한 후 모델 생성에 사용되었다.
모델은 전체 데이터의 70%인 훈련데이터 세트를 사용하여 생성되었고 전체 데이터의 30%인 테스트데이터 세트를 사용하여 성능을 평가하였다. 훈련데이터 세트에서 최종 모델 생성을 위하여 5겹 교차 검정을 사용하여 초매개변수를 조정(Tuning)하였다[30][31]. 모델에서 선택된 개별 체성분 지표의 기여도를 결정하기 위해 상대 변수 중요도를 계산하였다. 테스트데이터 세트를 사용하여 모델의 성능을 비교하기 위하여 AUROC, F1 점수, Kappa, 민감도, 특이도 값을 계산하였고 2000번의 부트스랩 복제를 통해 95% 신뢰구간을 계산하였다. F1 점수, Kappa, 민감도, 및 특이도 값을 계산하기 위한 최적 임계값은 Youden 지수에 의해 계산되었다. 익스트림 그래디언트 부스팅 모델의 AUROC 값과 각 모델의 AUROC 값은 z-점수에 의해 유의확률 값을 도출하여 차이를 비교하였다[32][33].
통계 분석은 R 버전 4.2.1(R Foundation for Statistical Computing, Vienna, Austria)을 사용하였다. 통계적 가설은 양측검정에 대하여 유의수준 0.05로 검정되었다.
Ⅳ. 결 과
4.1 이상지질혈증군과 정상군의 비교
이상지질혈증군과 정상군간의 체성분 지표 비교 결과가 표 2에 제시되었다. 총 820명의 남성 중 74명이 이상지질혈증군, 746명이 정상군에 포함되었다. 이상지질혈증군의 평균연령은 57.3세, 정상군의 평균연령은 42.77세로 이상지질혈증군이 유의하게 평균연령이 높았다(p <0.001). 체지방률, 전신 및 부위별 체지방 퍼센트값(오른팔, 왼팔, 몸통, 오른다리, 왼다리), 내장지방레벨, 체지방량지수는 이상지질형증군이 정상군보다 유의하게 높게 나타났다(p <0.001). 50kHz-몸통 위상각, 골격근률, 체수분율은 정상군이 이상지질혈증군 보다 유의하게 높게 나타났다(p <0.001).
4.2 모델의 개발 및 성능평가
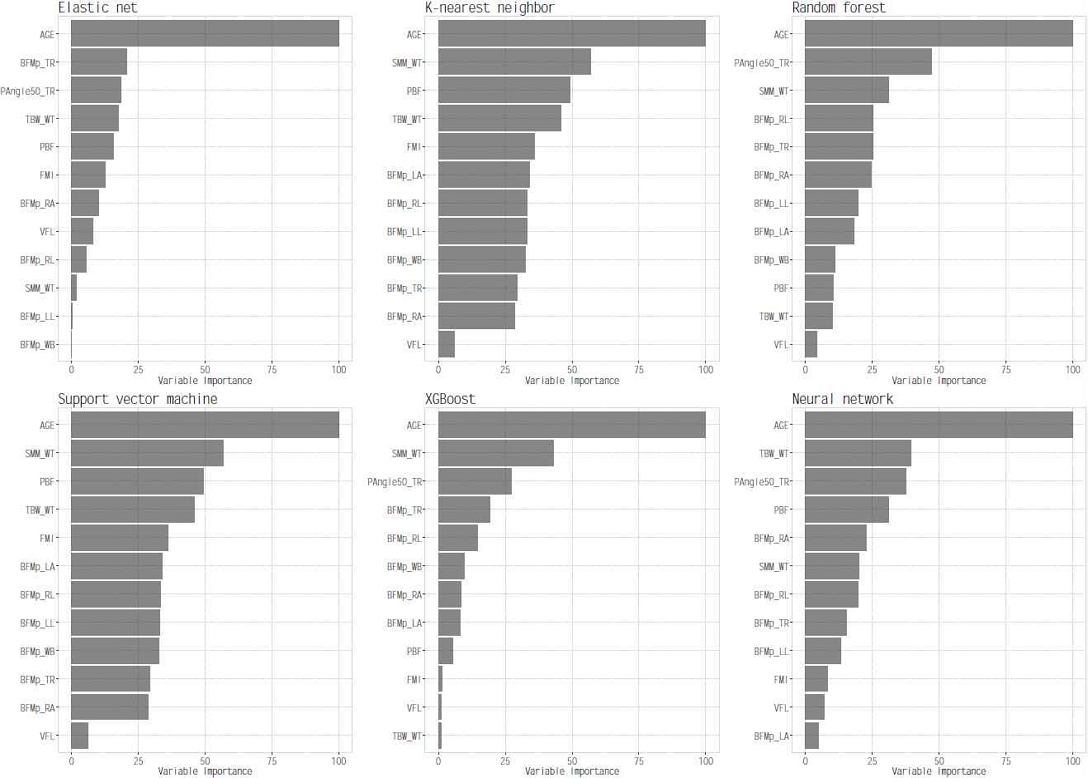
본 연구에서 이상지질혈증 식별 모델은 R의 Caret(Classification and regression training) 패키지의 자동 조정 알고리즘을 사용하여 학습되었다. R의 Caret 패키지는 여러 예측 모델과 데이터 분할, 전처리, 초매개변수 조정, 변수 중요도 추정 등의 기능을 위한 함수들을 포함하고 있어서 비교적 쉽게 기계학습을 사용한 모델을 생성할 수 있다[34]. 이상지질혈증 식별 모델에서 선택된 변수들의 상대적 중요도를 그림 1에서 보여준다. 모든 모델에서 연령이 상대적 중요도가 가장 높게 나타났다.
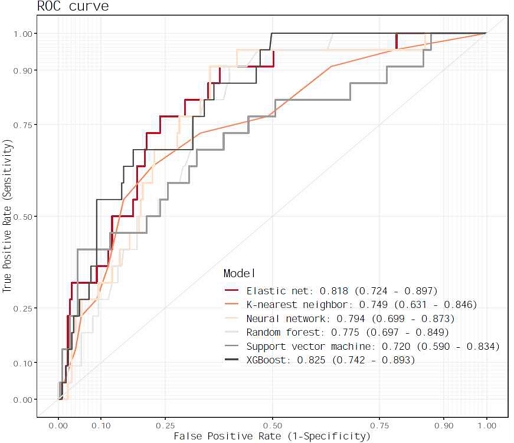
대부분의 모델에서 골격근률, 50kHz-몸통 위상각, 체지방률이 상대적 중요도가 높게 나타났다. 최종 모델의 성능 비교 결과는 표 3과 그림 2에서 보여준다. 익스트림 그래디언트 부스팅 모델의 AUROC 값이 가장 높게 나타났으며 엘라스틱 넷 모델, 신경망 모델이 순서대로 높게 나타났다(익스트림 그래디언트 부스팅, AUROC = 0.825 [95% CI, 0.742-0.893]; 엘라스틱 넷, 0.818 [0.724-0.897]; 신경망 모델, 0.794 [0.699-0.873]). 그러나 가장 높은 AUROC 값을 가지는 익스트림 그래디언트 부스팅 모델의 AUROC 값과 각 모델의 AUROC 값의 비교 결과 유의하게 차이를 보이지 않았다. 서포트 벡터 머신 모델은 F1 점수와 Kappa 값이 가장 높았으나 AUROC 값이 가장 낮게 나타났다(AUROC = 0.72 [0.59-0.834]; Kappa = 0.388 [0.18-0.582]; F1 점수 = 0.439 [0.24-0.615]). AUROC 값과 F1 점수 및 Kappa 값을 종합적으로 봤을 때, 그래디언트 부스팅 모델의 성능이 높았다(AUROC = 0.825 [0.742-0.893]; Kappa = 0.307 [0.159-0.452]; F1 점수 = 0.4 [0.243-0.531]).
V. 결론 및 향후 과제
심혈관 질환은 전 세계적으로 사망의 주요 원인으로 알려져 있으며, 이상지질혈증은 심혈관 질환의 선행 질환이자 위험인자이다. 이상지질혈증은 혈액 채취 통해 콜레스테롤과 중성지방 값을 기준으로 식별해 왔다. 침습적 방법이 아닌 비침습적 방법을 사용하여 이상지질혈증의 식별을 용이하게 하는 도구의 개발은 예방과 조기 발견을 위해 중요하다. 비만은 이상지질혈증과 밀접한 연관성이 있다고 알려져 있다. 비만을 정의하는 BMI 뿐만아니라 체성분 지표는 이상지질혈증과 높은 연관성이 있다.
본 연구에서는 비침습적 측정으로 얻어진 체성분 지표를 이용하여 이상지질혈증 식별 모델을 6개의 기계학습 접근 방식으로 개발하였다. 대부분의 이상지질혈증 식별 모델에서 연령, 골격근률, 50kHz-몸통 위상각, 체지방률이 상대적 중요도가 높은 변수로 선택되었다. 모델의 성능은 AUROC 값과 F1 점수 및 Kappa 값을 종합적으로 봤을 때, 익스트림 그래디언트 부스팅 모델이 높게 나타났다.
본 연구는 몇 가지 제한점이 있다. 첫째, 기계학습을 이용하여 강력한 성능을 가진 모델을 구축하기 위해서는 본 연구에 사용된 표본의 크기는 충분하다고 할 수는 없다. 둘째, 분석에서 사용된 데이터는 5개의 기관에서 수집된 데이터로 동일한 표준작업지짐서에 따라 잘 훈련된 임상 연구 코디네이터가 수행하였다 하더라도 기관별 편향(Bias) 존재할 수 있다. 셋째, 분석에 사용된 데이터는 단면 연구 데이터로 체성분 지표와 이상지질혈증 간에 인과 관계를 결정하기 어렵다. 따라서 이러한 제한점을 극복하기 위해서는 더 큰 표본 크기의 전향적 연구를 실시하여 추가로 검증할 필요가 있다.
그럼에도 불구하고 이 연구는 기계학습 기법을 사용하여 한국 남성을 대상으로 비침습적 방법인 체성분 지표 기반 이상지질혈증을 식별하는 모델을 개발한 첫 연구이다. 이상지질혈증을 식별하는데 체성분 지표의 잠재력을 입증하였다. 기계학습 기법을 사용하여 질환을 식별하는 것은 임상의가 의사 결정을 하는데 도움이 될 것으로 예상된다.
Acknowledgments
본 연구는 한국한의학연구원의 기본사업 과제(KSN1824130, KSN1923111)의 지원을 받아 수행되었음
References
-
A. L. Samuel, "Some studies in machine learning using the game of checkers", IBM Journal of research and development, Vol. 44, No. 1.2, pp. 206-226, Jan. 2000.
[https://doi.org/10.1147/rd.441.0206]

-
L. Kong, et al., "CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine", Nucleic acids research, Vol. 35, No. suppl_2 , pp. W345–W349, Jul. 2007.
[https://doi.org/10.1093/nar/gkm391]

-
S. Cramer, et al., "An extensive evaluation of seven machine learning methods for rainfall prediction in weather derivatives", Expert Systems with Applications, Vol. 85, pp. 169-181, Nov. 2017.
[https://doi.org/10.1016/j.eswa.2017.05.029]

-
X. A. López-Cortés, et al., "Fast detection of pathogens in salmon farming industry", Aquaculture, Vol. 470, pp. 17-24, Mar. 2017.
[https://doi.org/10.1016/j.aquaculture.2016.12.008]

-
J. Kang, et al., "Machine learning approaches for predicting radiation therapy outcomes: a clinician's perspective", International Journal of Radiation Oncology Biology Physics, Vol. 93, No. 5, pp. 1127-1135, Dec. 2015.
[https://doi.org/10.1016/j.ijrobp.2015.07.2286]

-
S. Rauschert, et al., "Machine learning and clinical epigenetics: a review of challenges for diagnosis and classification", Clinical epigenetics, Vol. 12, No. 51, pp. 1-11, Apr. 2020.
[https://doi.org/10.1186/s13148-020-00842-4]

-
A. Belle, et al., "Big data analytics in healthcare", BioMed research international, Jul. 2015.
[https://doi.org/10.1155/2015/370194]

-
F. Wang, L. P. Casalino, and D. Khullar, "Deep learning in medicine—promise, progress, and challenges", JAMA internal medicine, Vol. 179, No. 3, pp. 293-294, Dec. 2018.
[https://doi.org/10.1001/jamainternmed.2018.7117]

-
A. Holzinger and I. Jurisica, "Knowledge discovery and data mining in biomedical informatics: The future is in integrative, interactive machine learning solutions", Interactive knowledge discovery and data mining in biomedical informatics: state-of-the-art and future challenges, Vol. 8401, pp. 1-18, 2014.
[https://doi.org/10.1007/978-3-662-43968-5_1]

-
E.-S. Jin, et al., "Dyslipidemia fact sheet in South Korea, 2022", Diabetes & Metabolism Journal, Vol. 47, No. 5, pp. 632-642, Sep. 2023.
[https://doi.org/10.4093/dmj.2023.0135]

-
A. J. Berberich and R. A. Hegele, "A modern approach to dyslipidemia", Endocrine reviews, Vol. 43, No. 4, pp. 611-653, Aug. 2022.
[https://doi.org/10.1210/endrev/bnab037]

- Organization WHO, Cardiovascular diseases, https://www.who.int/news-room/fact-sheets/detail/cardiovascular-diseases-(cvds), . [accessed: Jun. 11, 2021]
- Organization WHO, Obesity and overweight, https://www.who.int/news-room/fact-sheets/detail/obesity-and-overweight, . [accessed: Mar. 01, 2024]
-
K. J. Smalley, et al., "Reassessment of body mass indices", The American journal of clinical nutrition, Vol. 52, No. 3, pp. 405-408, Sep. 1990.
[https://doi.org/10.1093/ajcn/52.3.405]

-
F. Curtin, et al., "Body mass index compared to dual-energy x-ray absorptiometry: evidence for a spectrum bias", Journal of clinical epidemiology, Vol. 50, No. 7, pp. 837-843, Jul. 1997.
[https://doi.org/10.1016/S0895-4356(97)00063-2]

-
H. Ito, et al., "Excess accumulation of body fat is related to dyslipidemia in normal-weight subjects", International journal of obesity, Vol. 28, No. 2, pp. 242-247, Feb. 2004.
[https://doi.org/10.1038/sj.ijo.0802528]

-
M. Zaid, et al., "Anthropometric and metabolic indices in assessment of type and severity of dyslipidemia", Journal of physiological anthropology, Vol. 36, No. 1, pp. 1-10, Feb. 2017.
[https://doi.org/10.1186/s40101-017-0134-x]

-
L. Zhang, et al., "Prediction of hypertension, hyperglycemia and dyslipidemia from retinal fundus photographs via deep learning: A cross-sectional study of chronic diseases in central China", PloS one, Vol. 15, No. 5, pp. e0233166, May 2020.
[https://doi.org/10.1371/journal.pone.0233166]

-
M. Correia, et al., "Machine learning modelling of blood lipid biomarkers in familial hypercholesterolaemia versus polygenic/environmental dyslipidaemia", Scientific reports, Vol. 11, No. 1, pp. 3801, Feb. 2021.
[https://doi.org/10.1038/s41598-021-83392-w]

-
G. Gutiérrez-Esparza, et al., "A machine learning approach to personalized predictors of dyslipidemia: a cohort study", Frontiers in Public Health, Vol. 11, pp. 1213926, Sep. 2023.
[https://doi.org/10.3389/fpubh.2023.1213926]

-
J. Liu, et al., "Development and validation of predictive model based on deep learning method for classification of dyslipidemia in Chinese medicine", Health information science and systems, Vol. 11, No. 1, pp. 21, Apr. 2023.
[https://doi.org/10.1007/s13755-023-00215-0]

-
S. Cui, et al., "Research on risk prediction of dyslipidemia in steel workers based on recurrent neural network and lstm neural network", IEEE Access, Vol. 8, pp. 34153-34161, Feb. 2020.
[https://doi.org/10.1109/ACCESS.2020.2974887]

-
A. Takhttavous, et al., "Predicting the 10-year incidence of dyslipidemia based on novel anthropometric indices, using data mining", Lipids in Health and Disease, Vol. 23, No. 1, pp. 33, Jan. 2024.
[https://doi.org/10.1186/s12944-024-02006-2]

-
R. Hesse, et al., "Familial hypercholesterolemia identification by machine learning using lipid profile data performs as well as clinical diagnostic criteria", Circulation: Genomic and Precision Medicine, Vol. 15, No. 5, pp. e003324, Oct. 2022.
[https://doi.org/10.1161/CIRCGEN.121.003324]

-
A. Pina, et al., "Virtual genetic diagnosis for familial hypercholesterolemia powered by machine learning", European journal of preventive cardiology, Vol. 27, No. 15, pp. 1639-1646, Oct. 2020.
[https://doi.org/10.1177/2047487319898951]

-
H. Yang, et al., "Machine learning-aided risk prediction for metabolic syndrome based on 3 years study", Scientific Reports, Vol. 12, No. 1, pp. 2248, Feb. 2022.
[https://doi.org/10.1038/s41598-022-06235-2]

-
X. Deng, et al., "Early prediction of body composition parameters on metabolically unhealthy in the Chinese population via advanced machine learning", Frontiers in Endocrinology, Vol. 14, pp. 1228300, Aug. 2023.
[https://doi.org/10.3389/fendo.2023.1228300]

-
M. Akbarzadeh, et al., "Evaluating machine learning-powered classification algorithms which utilize variants in the GCKR gene to predict metabolic syndrome: Tehran Cardio-metabolic Genetics Study", Journal of Translational Medicine, Vol. 20, No. 1, pp. 164, Apr. 2022.
[https://doi.org/10.1186/s12967-022-03349-z]

-
S. Y. Kim, G. H. Nam, and B. M. Heo, "Identification of metabolic syndrome based on anthropometric, blood and spirometric risk factors using machine learning", Applied Sciences, Vol. 10, No. 21, pp. 7741, Nov. 2020.
[https://doi.org/10.3390/app10217741]

-
E. LeDell, M. Petersen, and M. Laan, "Computationally efficient confidence intervals for cross-validated area under the ROC curve estimates", Electronic journal of statistics, Vol. 9, No. 1, pp. 1583-1607, Aug. 2015.
[https://doi.org/10.1214/15-EJS1035]

-
S. Geisser, "The predictive sample reuse method with applications", Journal of the American statistical Association, Vol. 70, No. 350, pp. 320-328, Jan. 1975.
[https://doi.org/10.1080/01621459.1975.10479865]

-
J. A. Hanley and B. J. McNeil, "The meaning and use of the area under a receiver operating characteristic (ROC) curve", Radiology, Vol. 143, No. 1, pp. 29-36, Apr. 1982.
[https://doi.org/10.1148/radiology.143.1.7063747]

-
J. A. Hanley and B. J. McNeil, "A method of comparing the areas under receiver operating characteristic curves derived from the same cases", Radiology, Vol. 148, No. 3, pp. 839-843, Sep. 1983.
[https://doi.org/10.1148/radiology.148.3.6878708]

-
M. Kuhn, "Building predictive models in R using the caret package", Journal of statistical software, Vol. 28, No. 5, pp. 1-26, Nov. 2008
[https://doi.org/10.18637/jss.v028.i05]


1999년 2월 : 이화여자대학교 통계학과(이학사)
2001년 2월 : 이화여자대학교 통계학과(이학석사)
2012년 2월 : 충남대학교 통계학과(이학박사)
2017년 3월 ~ 현재 : 한국한의학연구원 기술연구원
관심분야 : 공개 빅데이터 및 임상 자료 분석, 기계학습

2003년 2월 : 원광대학교 한의학과(한의학사)
2007년 2월 : 원광대학교 한의학과(한의학석사)
2011년 2월 : 원광대학교 한의학과(한의학박사)
2019년 5월 ~ 현재 : 한국한의학연구원 책임연구원
관심분야 : 한의 생체지표 표준화, 과학화, 데이터 표준화