한국 중장년 여성에 대한 기계학습 기반 비침습적 요인들을 이용한 당뇨 및 공복혈당 장애 분류
초록
본 연구는 기계학습 기반으로 비침습적 생체지표들을 이용하여 적극적인 혈당 관리를 필요로 하는 당뇨 및 공복혈당 장애 분류 모델을 생성하고 성능을 비교하는 것을 목적으로 한다. 총 215명의 40세 이상 69세 미만의 여성을 대상으로 6개의 기계학습 알고리즘을 이용하여 모델을 생성하고 중첩 교차검증(Nested cross-validation)을 사용하여 성능을 비교하였다. 그 결과 엘라스틱 넷 로지스틱 회귀분석의 성능이 다소 높게 나타났고 맥박수 표준편차(STD_PR)와 이완기의 면적(Dias_area)이 상대적으로 중요도가 높은 변수로 나타났다. 연구 결과는 당뇨 및 공복혈당 장애 분류를 위한 비침습적 변수의 가능성을 보여주었다. 또한 기계학습 알고리즘을 사용한 분류는 임상의가 의사 결정을 하고 의료 서비스를 제공하는데 도움이 될 것으로 예상된다.
Abstract
This study aimed to build models to classify diabetes and impaired fasting glucose requiring active management of blood sugar based on machine learning approaches using noninvasive variables, and to evaluate the performance of each model. The classification models of diabetes and impaired fasting glucose in a total of 215 women aged 40 to 69 were built through six machine learning approaches. The performance of each model was evaluated using nested cross-validation. The model using elastic net logistic regression reported slightly higher performance. The area of diastolic period and standard deviation of pulse rate were founded to be relatively important variables in diabetes and impaired fasting glucose. These results showed the potential of noninvasive variables for the classification of diabetes and impaired fasting glucose. Also, classification based on machine learning approaches can help clinicians make clinical decisions and provide healthcare services.
Keywords:
machine learning, diabetes, impaired fasting glucose, pulse wave, anthropometric measuresⅠ. 서 론
다양한 종류의 정형 또는 비정형 데이터를 머신러닝이나 딥러닝 기반으로 분석하여 결과를 해석하고 미래를 예측하는 과정은 4차 산업혁명 시대의 핵심기술 중 하나이다. 아서 사뮤엘(Arthur Samuel) 은 기계학습은 컴퓨터가 명시적으로 프로그래밍하지 않고도 학습할 수 있는 기능을 제공하는 컴퓨터 과학 분야라고 정의하였다[1]. 기계학습은 데이터로부터 학습하여 기계에게 데이터를 효율적으로 처리하는 방법을 가르치고 예측할 수 있는 알고리즘을 생성한다. 성능이 좋은 명시적 알고리즘을 개발하는 것이 어렵거나 불가능한 영역에서 기계학습은 뛰어난 성능으로 사용되어 오고 있다[2]. 또한 최근 사용 가능한 대용량 데이터가 많아짐에 따라 기계학습에 대한 수요가 증가하고 있다[3][4].
기계학습은 의료, 보건 산업 및 의생명공학 분야를 비롯한 다양한 산업 분야에서 의사 결정 과정에 중요한 역할을 하고 있다. 최근 의생명공학 분야의 첨단 컴퓨팅과 디지털 이미지 처리 기술이 급속히 발전하고 의생명 데이터의 규모가 커지면서 기계학습 기반의 알고리즘이 요구되고 있다[2]. 생체 신호, 생체 이미지, 인체측정 변수, 사회인구학적 특성 등 다양한 변수들을 기반으로 질환을 분류하거나 예측하고 질환에 대한 치료 효과를 예측하기 위하여 인공지능(Artificial intelligence) 기법인 데이터 마이닝(Data mining), 기계학습(Machine learning), 딥러닝(Deep learning) 등이 사용되고 있다[5]-[9].
당뇨(Diabetes)는 혈당 수치가 높아지거나 인체 내에서 생산되는 인슐린이 몸의 기관에 작용하는 효과의 감소에 따른 당질대사의 장애로 급속한 서구화, 운동 부족, 과다 열량 섭취, 스트레스 등에 의해 급격히 증가하고 있다[10][11]. 당뇨의 세계적 유병률은 1980년 4.7%에서 2014년 8.5%로 거의 두 배 가까이 증가하였고 개입이 이루어지지 않을 경우, 2045년에는 최소 6억 2,900만 명에 이를 것으로 추정한다[12]. 혈당의 증가는 눈, 심장, 신장, 혈관 및 신경에 심각한 손상을 초래하고 빈뇨, 흐릿한 시야, 극심한 피로, 과도한 갈증 등과 같은 증상을 유발한다[13].
당뇨 및 공복혈당 장애(Impaired fasting glucose)는 혈액에서 추출한 공복혈당(Fasting glucose) 또는 당화혈색소(HbA1c)의 수치 등을 기준으로 진단하고 있다. 이 질환은 혈액 지표뿐만 아니라 비침습적으로 측정할 수 있는 변수인 체질량지수, 허리둘레, 키와 허리둘레 비율 등의 인체 측정변수(Anthropometric measures)와 맥파(Pulse wave) 변수 등과 매우 높은 연관성을 지니고 있다[14]-[16]. 그러나 기계학습 기반 40세 이상의 한국 여성을 대상으로 인체 측정변수와 맥파 변수를 사용하여 혈당 관리를 필요로 하는 당뇨 및 공복혈당 장애를 분류하기 위한 연구는 아직 수행되지 않았다.
따라서 본 연구는 여러 기계학습 접근법을 기반으로 혈액검사 결과와 같은 침습적 방법으로 얻어진 정보가 아니라 비침습적 방법으로 얻어진 인체계측 변수와 맥파 변수를 이용하여 당뇨 및 공복혈당 장애 분류 모델을 생성하고 성능을 평가한다.
Ⅱ. 기계학습 기반 질환 예측 및 분류 관련 기존 연구
암이나 질환을 예측하거나 분류하고 암이나 질환에 대한 치료 효과를 예측하기 위하여 기계학습 기반의 알고리즘을 사용한 많은 연구가 진행되어 왔다.
폐암의 경우, A. McWilliams et al.[17]은 다항 로지스틱 회귀분석을 이용하여 환자의 인구통계학적 특성 및 저선량 컴퓨터 단층촬영(Low-dose computed tomography) 이미지를 바탕으로 폐암 확률을 추정하였다. 고령, 여성, 폐암 가족력, 폐기종, 결절 크기, 상엽의 결절 위치, 부분 고형 결절 유형, 하부 결절 수 및 침상이 폐암 예측 변수로 나타났다. C. M. Lynch et al.[18]은 선형 회귀분석, 의사 결정 나무, 그래디언트 부스트, 서포트 벡터 머신 및 맞춤형 앙상블을 사용하여 폐암 환자의 생존 모형을 생성하였다. 폐암 환자의 생존에 영향을 주는 변수는 종양 등급, 종양 크기, 성별, 연령, 암의 병기 및 원발성 수였다.
심혈관 질환의 경우, A. Rahim et al.[19]은 의사결정 나무, k-최근접 이웃, 로지스틱 회귀분석 및 앙상블을 이용하여 심혈관 질환 예측 모형을 개발하였다.
이 모형에서 심혈관 질환 예측에 큰 영향을 주는 상위 5개의 변수는 수축기 혈압, 연령, 총콜레스테롤, 흡연, 이완기 혈압으로 나타났다. A. Dinh et al.[20]은 로지스틱 회귀분석, 서포트 벡터 머신, 랜덤 포레스트, 그래디언트 부스트 및 가중 앙상블을 기반으로 심혈관 감지 모형과 당뇨병 전단계 및 당뇨병 감지 모형을 개발하였다. 심혈관 질환은 연령, 수축기 혈압, 자가 보고 체중, 흉통 발생, 확장기 혈압이 중요한 식별 변수로 나타났고 당뇨는 허리둘레, 연령, 자가 보고 체중, 다리 길이, 나트륨 섭취량이 중요 식별 변수로 보고되었다.
심장 관련 질환에서는 J. P. Li et al.[21]은 로지스틱 회귀분석, k-최근접 이웃, 서포트 벡터 머신, 의사 결정 나무 및 나이브 베이즈 방법을 사용하여 심장병을 진단하는 모형을 개발하였다. 성별, 가슴 통증, 혈청 콜레스테, 심전도 등이 심장병 모형에서 영향력 있는 변수로 나타났다. A. K. Dwivedi[22]은 인공 신경망, 서포트 벡터 머신, 로지스틱 회귀분석, k-최근접 이웃, 의사 결정 나무, 나이브 베이즈 기반 심장병 예측 모형을 생성하였다. 연령, 성별, 가슴 통증 타입, 혈당, 혈청 콜레스테롤 등이 모형의 변수로 사용되었다.
당뇨 관련 질환의 경우, B. Farran et al.[23]은 로지스틱 회귀, k-최근접 이웃, 서포트 벡터 머신을 사용하여 당뇨병(유형 II)의 미래 위험을 예측하는 예후 모형을 구축하였다. 연령, 성별, 체질량 지수, 기존 고혈압 여부, 고혈압 가족력이 모형의 변수로 사용되었다. Q. Zou, et al.[24]은 중국인을 대상으로 당뇨병을 예측하기 위해 의사 결정 나무, 랜덤 포레스트 및 신경망 방법을 사용하였다. 나이, 맥박, 호흡, 좌 수축기 혈압 우 수축기 혈압, 좌 이완기 혈압, 우 이완기 혈압, 신장, 체중, 체격지수, 공복 혈당, 허리둘레, 저밀도 지단백질 및 고밀도 지단백질이 모형의 변수로 사용되었다. A. Dagliati et al.[25]은 로지스틱 회귀분석, 나이브 베이즈, 서포트 벡터 머신 및 랜덤 포레스트 기반으로 제2형 당뇨병(T2DM) 합병증의 예측 모형을 구축하였다. 성별, 연령, 진단 시기, 체질량 지수, 당화혈색소(HbA1c), 고혈압 및 흡연 습관이 중요한 예측 변수로 나타났다. R. Pal et al.[26]은 서포트 벡터 머신, k-최근접 이웃, 의사 결정 나무, 나이브 베이즈 방법을 사용하여 당뇨병성 망막병증을 분류하였다. 미세 동맥류 감지 변수, 황반 중심, 시신경 유두 중심의 유클리드 거리 연령 및 시신경 유두의 직경 등이 입력 변수로 사용되었다.
Ⅲ. 기계학습 기반 당뇨 및 공복혈당 장애 분류 방법
3.1 연구 대상자 선정
본 연구는 대전대학교 천안한방병원에서 2021년 11월부터 2022년 7월까지 40세 이상 69세 미만의 고령자 대상으로 진행된 임상 연구 자료를 분석하였다. 임상 연구는 대전대학교 천안한방병원 임상시험 심사위원회의 승인을 받아 핼싱키 선언의 지침에 따라 수행되었다(IRB No. DJUMC-2021-BM-10). 총 303명의 참가자가 병원을 방문했으며 포함기준을 충족하지 못한 3명을 제외한 300명이 최종 등록하였다. 그중 측정 오류로 결측값을 가지는 대상자와 표본의 크기가 작은 남성 대상자를 제외한 여성 215명을 대상으로 분석하였다.
3.2 당뇨 및 공복혈당 장애군 정의
공복혈당 110mg/dl 이상 또는 당화혈색소 6.5% 이상 또는 의사로부터 당뇨로 진단받은 대상자들은 당뇨 및 공복혈당 장애군으로 분류하였고 그 외 대상자들을 정상군으로 분류하였다.
3.3 비침습적 변수들의 측정
인체계측 변수와 맥파 변수는 표준작업지짐서에 따라 훈련받은 임상 연구 코디네이터에 의해 측정되었다. 체중, 키, 허리둘레(WCIR), 엉덩이둘레(HCIR)는 얇은 옷을 입은 상태에서 측정하였다. 체질량 지수(BMI)는 체중을 키의 제곱으로 나눈 값으로, 키 대비 허리둘레(WHtR)는 허리둘레를 키로 나눈 값으로 계산되었다. 수축기 혈압(SYS)과 이완기 혈압(DIAS)은 측정 전 최소 10분 동안 휴식을 취한 후 측정되었다. 맥파는 안압계 맥파 측정기 DMP-LIFE plus(DAEYOMEDI CO., South Korea)를 이용하여 좌측 요골동맥에서 측정하였고 측정 오류가 발생할 경우, 최대 3회까지 다시 측정되었다. 이 연구에 사용된 인체계측 변수와 맥파 변수에 대한 자세한 정보는 표 1에 설명되어 있다.
모든 통계 분석은 R 버전 4.2.1(R Foundation for Statistical Computing, Vienna, Austria)을 사용하여 수행되었고 모든 통계적 가설검정은 유의수준 0.05와 양측검정을 사용하였다. 당뇨 및 공복혈당 장애 군과 정상군 간에 인체계측 변수와 맥파 변수를 비교하기 위해 이표본 t 검정(two-sample t test)를 사용하였다.
3.4 통계분석
인체계측 변수와 맥파 변수를 이용한 당뇨 및 공복혈당 장애의 분류 모형은 6가지 기계학습 접근 방식을 통해 구축되었다. 사용된 모형은 엘라스틱 넷(Elastic Net, E-net), k-최근접 이웃(k-Nearest Neighbor, K-NN), 랜덤 포레스트(RF, Random Forest), 서포트 벡터 머신(Support vector machine, SVM), 익스트림 그래디언트 부스트(XGBoost, Extreme Gradient Boost) 및 신경망(NN, Neural Network)이다. 모형 생성 전 인체계측 변수와 맥파 변수의 값은 표준화되었다. 최종 6개 모형에서 선택된 개별 인체계측 변수와 맥파 변수의 기여도를 식별하기 위해 상대 변수 중요도(Relative variable importance)를 계산하였다. 각 모형의 성능은 5개의 외부 분할(5 outer split)과 5개의 내부 분할(5 inner splits)이 있는 중첩된 교차검증(Nested cross-validation)을 사용하여[27] 정확도(Accuracy), Kappa, 정밀도(Precision), F1 점수(F1 score), 민감도(Sensitivity), 특이도(Specificity), 수신자 작동 특성 곡선값 아래 영역(AUC, Areas Under the receiver operating characteristic Curve) 값을 95% 신뢰구간(CI)과 함께 제시하였다. 최적 임계값은 Youden 지수에 의해 결정하였고 신뢰구간은 2000번의 부트스트랩 복제를 사용하여 계산하였다. 엘라스틱 넷 모형의 AUC와 각 모형의 AUC를 비교하기 위한 유의확률 값(P value)은 z-점수[28][29]에서 도출되었다.
Ⅳ. 실험 결과
4.1 당뇨 및 공복혈당 장애군과 정상군의 비교
표 2는 인체계측 변수와 맥파 변수를 당뇨 및 공복혈당 장애군과 정상군 간에 비교한 결과를 나타낸다. 본 연구에 포함된 40~69세 여성 215명 중 35명이 당뇨 및 공복혈당 장애군에 180명이 정상군에 포함되었다. 인체계측 변수 증 수축기 혈압(SYS)과 이완가 혈압(DIAS)이 당뇨 및 공복혈당 장애군과 정상군 사이에 유의한 차이를 보였다(수축기 혈압, p = 0.005; 이완기 혈압, p = 0.013). 맥파 변수 중에서는 수축기 면적(Sys_area), 최대 맥박 진폭(PPI), 최대 적용 압력에 대한 최대 진폭에서 적용 압력의 비율(PDI)을 제외한 모든 맥파 변수에서 유의한 차이가 나타났다.
4.2 모형 생성 및 성능 평가
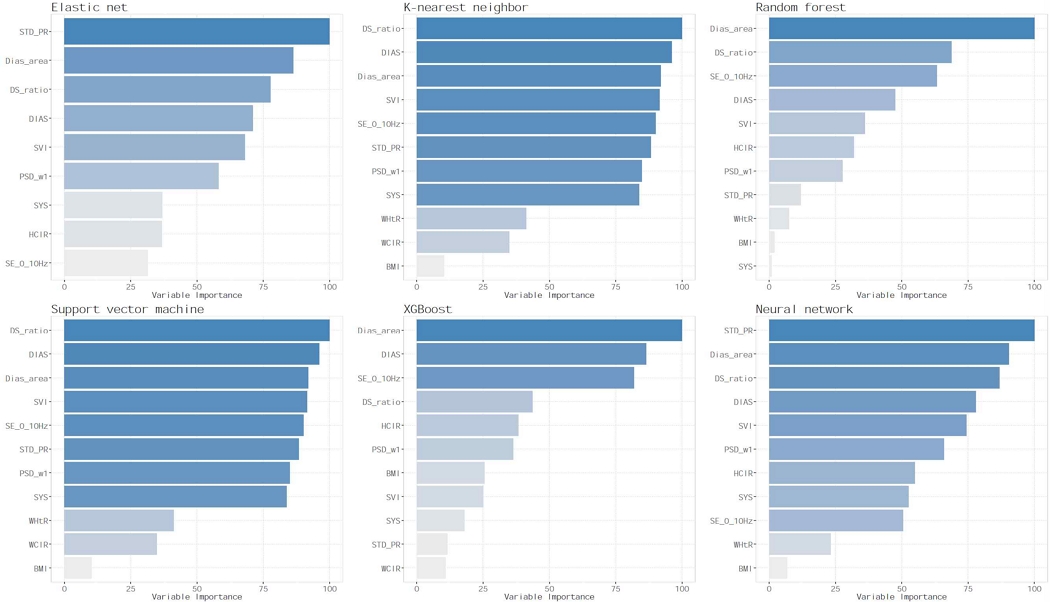
그림 1은 각 모형에서 선택된 변수의 상대적 중요도를 보여준다. 6개 모형 대부분에서 이완기의 면적(Dias_area), 이완기 면적과 수축기 면적의 비율(DS_ratio), 이완기 혈압(DIAS), 맥박수 표준편차(STD_PR) 등이 당뇨 및 공복혈당 장애에 상대적으로 강한 영향을 주는 것으로 나타났다. 표 3은 각 모형의 성능으로 정확도, Kappa, 정밀도, F1 점수, 민감도, 특이도를 나타낸다. 표 4는 AUC 값과 엘라스틱 넷 모형의 AUC와 각 모형의 AUC를 비교한 결과를 나타낸다. 그림 2는 각 모형의 AUC 값과 수신자 작동 특성(ROC, Receiver Operating Characteristic) 곡선을 보여준다.
엘라스틱 넷 로지스틱 회귀분석을 사용한 모형이 가장 높은 AUC 값, Kappa 및 F1 점수를 보고하였다(AUC = 0.724 [95% CI, 0.634-0.806]; Kappa = 0.27 [95% CI, 0.15-0.384]; F1 점수 = 0.436 [95% CI, 0.314-0.541]). 여러 가지 성능 평가 지표에서 상대적으로 우수한 성능을 나타내는 엘라스틱 넷 로지스틱 회귀분석을 사용한 최종 모형은 식 (1)과 같다. 는 인체계측 변수와 맥파 변수가 X 일 때, 당뇨 및 공복혈당 장애군으로 분류될 확률을 의미한다.
| (1) |
k-최근접 이웃 접근법을 사용한 모형이 두 번째로 높은 AUC 값, Kappa 및 F1 점수를 보였다(AUC = 0.715 [95% CI, 0.618-0.803; Kappa = 0.264 [95% CI, 0.136-0.395]; F1 점수 = 0.427 [95% CI, 0.305-0.538]). AUC 값 기준으로 성능이 높은 다음 순서는 랜덤 포레스트, 익스트림 그래디언트 부스트, 신경망, 서포트 벡터 머신 방법을 이용한 모형이었다(RF, 0.703 [95% CI, 0.616-0.783]; XGBoost, 0.694 [95% CI, 0.593-0.787]; NN, 0.689 [95% CI, 0.597-0.776]; SVM, 0.652 [95% CI, 0.544-0.746]). AUC 값이 가장 큰 엘라스틱 넷 모형과 나머지 5개의 모형의 AUC 값을 Z 검정으로 비교한 결과 통계적으로 유의하게 엘라스틱 넷 모형의 AUC 값이 크다고는 볼 수는 없었다. 신경망 방법을 사용한 모형이 정확도, Kappa, 정밀도, F1 점수에서는 상대적으로 가장 낮은 값을 보였다(정확도 = 0.512 [95% CI, 0.447-0.572]; Kappa = 0.152 [95% CI, 0.081-0.227]; 정밀도, 0.234 [95% CI, 0.167-0.31], F1 점수 = 0.371 [95% CI, 0.276-0.462]).
Ⅴ. 결론 및 향후 과제
당뇨환자의 혈당 증가는 전신에 걸쳐 합병증이 생길 수 있으며 특히 심근경색이나 뇌졸중 등과 같은 증상은 중요한 사망원인으로 알려져 있다. 따라서 당뇨의 발병 가능성을 예측하고 예방하는 것은 중요하다. 의료, 보건 산업 및 의생명공학 분야에서 통계적 기계학습 기법은 질환의 예측 및 분류 분석에 자주 사용되고 있다. 기계학습 모형은 임상의가 환자에게 발병할 수 있는 질환과 관련 지표 간의 임상 연관성을 더 잘 이해하고 발견하도록 지원하여 예방 관리 및 치료 방법을 개선하는 데 도움이 된다[30]. 그러나 기계학습 기반 40세 이상의 한국 여성을 대상으로 인체 측정변수와 맥파 변수를 사용하여 적극적인 혈당 관리를 필요로 하는 당뇨 및 공복혈당 장애를 분류하기 위한 연구는 아직 수행되지 않았다.
본 연구에서는 침습적 방법인 혈액채취 전에 비침습적 측정으로 얻을 수 있는 인체계측 정보나 맥파 정보를 이용하여 당뇨 및 공복혈당 장애 분류 모형을 6가지 기계학습 접근 방식으로 생성하였다. 각 모형의 성능을 평가한 결과 전반적인 성능은 엘라스틱 넷 방법이 다소 높게 나타났다.
본 연구는 다음과 같은 제한점을 갖는다. 첫째, 기계학습 기반 분석을 위해 더 많은 데이터 축척이 필요하다. 특히 남성의 경우, 표본 크기의 부족하여 본 연구에서는 여성만을 대상으로 분석하였다. 더 많은 데이터를 축적한 후 남성에 대한 당뇨 및 공복혈당 장애 분류 연구도 필요하다. 둘째, 분석에 사용된 데이터는 정해진 시기에 대상 집단 전체에 대하여 동시에 관찰한 횡단면 연구이므로 질환인 당뇨와 입력 변수인 인체측정 변수 및 맥파 변수 간의 인과 관계 추정이 어렵다. 종단적 데이터를 축적하여 인과 관계를 알아보는 향후 연구가 필요하다. 셋째, 불완전한 데이터에 대해 기계 학습된 모형은 왜곡된 결론을 초래할 수 있다.
그럼에도 불구하고 이 연구 결과는 혈당 관리를 필요로 하는 당뇨 및 공복혈당 장애 분류를 위한 비침습적 변수의 가능성을 보여주었다. 또한 기계학습 알고리즘을 사용한 분류는 임상의가 의사 결정을 하고 의료 서비스를 제공하는데 도움이 될 것으로 예상된다.
Acknowledgments
본 연구는 한국한의학연구원의 기본사업 과제(KSN1823130)와 정보통신기술기획평가원 과제(제2021-0-00104호)의 지원을 받아 수행되었음
References
-
A. L. Samuel, "Some Studies in Machine Learning Using the Game of Checkers", IBM Journal of Research and Development, Vol. 3, No. 3, pp. 210-229, Jul. 1959.
[https://doi.org/10.1147/rd.33.0210]

-
C. Park, C. C. Took, and J.-K. Seong, "Machine Learning in Biomedical Engineering", Biomedical Engineering Letters, Vol. 8, pp. 1-3, Feb. 2018.
[https://doi.org/10.1007/s13534-018-0058-3]

- B. Mahesh, "Machine Learning Algorithms-A Review", International Journal of Science and Research (IJSR), Vol. 9, No. 1, pp. 381-386, Jan. 2020.
-
R. Kohavi and F. Provost, "Glossary of Terms", Machine Learning—Special Issue on Applications of Machine Learning and the Knowledge Discovery Process. Machine Learning, Vol. 30, pp. 271-274. Feb. 1998.
[https://doi.org/10.1023/A:1017181826899]

-
I. Kavakiotis, O. Tsave, A. Salifoglou, N. Maglaveras, I. Vlahavas, and I. Chouvarda, "Machine Learning and Data Mining Methods in Diabetes Research", Computational and Structural Biotechnology Journal, Vol. 15, pp. 104-116, Jan. 2017.
[https://doi.org/10.1016/j.csbj.2016.12.005]

- K. Rajesh and V. Sangeetha, "Application of Data Mining Methods and Techniques for Diabetes Diagnosis", International Journal of Engineering and Innovative Technology, Vol. 2, No. 3, pp. 224-229, Sep. 2012.
-
S. Ding, et al., "Predicting heart cell types by using transcriptome profiles and a machine learning method", Life, Vol. 12, No. 2, p. 228, Jan. 2022.
[https://doi.org/10.3390/life12020228]

-
S. Devi, S. R. Gaikwad, and R. Harikrishnan, "Prediction and Detection of Cervical Malignancy Using Machine Learning Models", Asian Pacific Journal of Cancer Prevention, Vol. 24, No. 4, pp. 1419-1433, Apr. 2023.
[https://doi.org/10.31557/apjcp.2023.24.4.1419]

-
C. Küpper, et al., "Identifying predictive features of autism spectrum disorders in a clinical sample of adolescents and adults using machine learning", Scientific reports, Vol. 10, No. 1, pp. 4805, Mar. 2020.
[https://doi.org/10.1038/s41598-020-61607-w]

- World Health Organization, "Definition, diagnosis and classification of diabetes mellitus and its complications. Part 1: Diagnosis and Classification of Diabetes Mellitus", pp. 1-66, 1999.
- G.-H. Yun, "Characteristics of Diabetes Mellitus among Korean Population", The Monthly Diabetes, pp. 60-63, Jan. 2005.
- World Health Organization, "Classification of diabetes mellitus", 2019.
-
M. M. Engelgau, K. M. Narayan, and W. H. Herman, "Screening for type 2 diabetes", Diabetes Care, Vol. 23, No. 10, pp. 1563-1580, Oct. 2000. ㅣ
[https://doi.org/10.2337/diacare.23.10.1563]

-
J. H. Chi and B. J. Lee, "Risk factors for hypertension and diabetes comorbidity in a Korean population: A cross-sectional study", PLoS One, Vol. 17, No. 1, pp. e0262757, Jan. 2022.
[https://doi.org/10.1371/journal.pone.0262757]

-
B. J. Lee and J. Y. Kim, "Identification of Type 2 Diabetes Risk Factors Using Phenotypes Consisting of Anthropometry and Triglycerides based on Machine Learning", IEEE Journal of Biomedical and Health Informatics, Vol. 20, No. 1, pp. 39-46, Jan. 2016.
[https://doi.org/10.1109/JBHI.2015.2396520]

-
H. Yokoyama, et al., "Pulse Wave Velocity in Lower-Limb Arteries Among Diabetic Patients with Peripheral Arterial Disease", Journal of Atherosclerosis and Thrombosis, Vol. 10, No. 4, pp. 253-258, Oct. 2003.
[https://doi.org/10.5551/jat.10.253]

-
A. McWilliams, et al., "Probability of Cancer in Pulmonary Nodules Detected on First Screening CT", New England Journal of Medicine, Vol. 369, No. 10, pp. 910-919, Sep. 2013.
[https://doi.org/10.1056/NEJMoa1214726]

-
C. M. Lynch, et al., "Prediction of lung cancer patient survival via supervised machine learning classification techniques", International Journal of Medical Informatics, Vol. 108, pp. 1-8, Dec. 2017.
[https://doi.org/10.1016/j.ijmedinf.2017.09.013]

-
A. Rahim, et al., "An Integrated Machine Learning Framework for Effective Prediction of Cardiovascular Diseases", IEEE Access, Vol. 9, pp. 106575-106588, Jul. 2021.
[https://doi.org/10.1109/ACCESS.2021.3098688]

-
A. Dinh, et al., "A data-driven approach to predicting diabetes and cardiovascular disease with machine learning", BMC Medical Informatics and Decision Making, Vol. 19, No. 1, pp. 1-15, Nov. 2019.
[https://doi.org/10.1186/s12911-019-0918-5]

-
J. P. Li, et al., "Heart Disease Identification Method using Machine Learning Classification in E-Healthcare", IEEE Access, Vol. 8, pp. 107562-107582, Jun. 2020.
[https://doi.org/10.1109/ACCESS.2020.3001149]

-
A. K. Dwivedi, "Performance evaluation of different machine learning techniques for prediction of heart disease", Neural Computing and Applications, Vol. 29, pp. 685-693, Sep. 2016.
[https://doi.org/10.1007/s00521-016-2604-1]

-
B. Farran, et al., "Use of Non-invasive Parameters and Machine-Learning Algorithms for Predicting Future Risk of Type 2 Diabetes: A Retrospective Cohort Study of Health Data From Kuwait", Frontiers in Endocrinology, Vol 10, pp. 624, Sep. 2019.
[https://doi.org/10.3389/fendo.2019.00624]

-
Q. Zou, K. Qu, Y. Luo, D. Yin, Y. Ju, and H. Tang, "Predicting Diabetes Mellitus With Machine Learning Techniques", Frontiers in Genetics, Vol. 9, p. 515, Nov. 2018.
[https://doi.org/10.3389/fgene.2018.00515]

-
A. Dagliati, et al., "Machine Learning Methods to Predict Diabetes Complications", Journal of Diabetes Science and Technology, Vol. 12, No. 2, pp. 295-302. May 2018.
[https://doi.org/10.1177/1932296817706375]

-
R. Pal, J. Poray, and M. Sen, "Application of machine learning algorithms on diabetic retinopathy", 2nd IEEE Int Conf Recent Trends Electron Inf Commun Technology, Bangalore, India, May 2017.
[https://doi.org/10.1109/RTEICT.2017.8256959]

-
S. Bates, T. Hastie, and R. Tibshirani, "Cross-validation: what does it estimate and how well does it do it?", Journal of the American Statistical Association, pp. 1-12, May 2023.
[https://doi.org/10.1080/01621459.2023.2197686]

-
J. A. Hanley and B. J. McNeil, "The meaning and use of the area under a receiver operating characteristic (ROC) curve", Radiology, Vol. 143, No. 1, pp. 29-36, Apr. 1982.
[https://doi.org/10.1148/radiology.143.1.7063747]

-
J. A. Hanley and B. J. McNeil, "A method of comparing the areas under receiver operating characteristic curves derived from the same cases", Radiology, Vol. 148, No. 3, pp. 839-843, Sep. 1983.
[https://doi.org/10.1148/radiology.148.3.6878708]

-
N. Sambyal, P. Saini, and R. Syal, "A review of statistical and machine learning techniques for microvascular complications in type 2 diabetes", Current Diabetes Reviews, Vol. 17, No. 2, pp. 143-155, Feb. 2021.
[https://doi.org/10.2174/1573399816666200511003357]


1999년 2월 : 이화여자대학교 통계학과(이학사)
2001년 2월 : 이화여자대학교 통계학과(이학석사)
2012년 2월 : 충남대학교 통계학과(이학박사)
2017년 3월 ~ 현재 : 한국한의학연구원 기술연구원
관심분야 : 공개 빅데이터 및 임상 자료 분석, 기계학습

1999년 2월 : 인제대학교 의용공학과(공학사)
2001년 2월 : 전북대학교 의용생체공학과(공학석사)
2006년 2월 : 전북대학교 메카트로닉스공학과(공학박사)
2007년 3월 ~ 현재 : 한국한의학연구원 책임연구원
관심분야 : 생체계측, 생체신호처리, 한의 의료기기 개발

1981년 2월 : 서울대학교 계산통계학과(이학사)
1984년 5월 : 미국 사우스캐롤라이나 대학교 통계학과(이학석사)
1989년 12월 : 미국 위스콘신 대학교 통계학과(이학박사)
1993년 3월 ~ 현재 : 충남대학교 정보통계학과 교수
관심분야 : 비모수 통계학, 생물 통계학