Health-AutoML: 헬스케어 데이터 분석을 위한 자동 적응형 다층 스태킹 앙상블 학습 프레임워크
초록
헬스케어 데이터는 클래스 불균형과 특성 간 복잡한 관계를 가지고 있어, 단일모델만으로는 모델의 다양성을 보장하지 못하며 데이터 패턴을 충분히 학습하기 어렵다. 이에 따라, 헬스케어 분야의 도메인 지식을 가진 연구자들이 다층으로 구성된 복잡한 앙상블 모델 구조를 쉽게 구축할 수 있는 자동화 방법의 필요성이 증가하고 있다. 본 논문은 헬스케어 데이터에 대해 적응형 다층 스태킹 앙상블 모델을 자동으로 구축하는 새로운 학습 프레임워크 알고리즘을 제안한다. 세 가지 헬스케어 데이터를 활용하여 제안된 프레임워크로 구축한 모델을 다른 AutoML 프레임워크와 비교하였으며, 결과는 정확도, 정밀도, 재현율, F1-score, AUC를 통해 평가되었다. 제안된 방법은 다른 AutoML 프레임워크와 비교하여 AUC 기준 평균 8.25%p 향상된 결과를 얻었다.
Abstract
Healthcare data is characterized by class imbalance and complex relationships between each feature in datasets, making it challenging for a single model to adequately learn data patterns. Thus, there is an emerging need for automated methods that enable researchers with domain knowledge in healthcare to effortlessly construct sophisticated multi-layer stacked ensemble models. In this paper, we introduce a novel algorithm within a learning framework that automatically constructs adaptive multi-layer stacking ensemble models tailored for healthcare data. Using three different healthcare datasets, the models constructed with the proposed framework are compared to other AutoML frameworks. Compared to other AutoML frameworks, the proposed method achieved an average improvement of 8.25%p in terms of AUC.
Keywords:
healthcare, AutoML, machine learning, stacking ensembleⅠ. 서 론
최근 몇 년 사이 컴퓨팅 성능이 발전함에 따라 머신러닝 및 딥러닝 기술이 혁신을 반복하고 있다. 머신러닝은 통계적 기법 및 수학적 알고리즘을 이용해 데이터를 학습시켜 예측 및 분류를 하며, 딥러닝은 신경망을 이용하여 데이터를 학습시켜 예측 및 분류를 하는 기술이다. 예측 및 분류 정확도가 올라감에 따라 헬스케어, 금융, 제조 등 다양한 분야에서 이를 활용하고자 하는 연구가 늘고 있다[1][2].
특히 헬스케어 데이터는 임상진단 등을 통해 데이터가 방대하게 축적되어 있다. 또한, 다양한 특성으로 이루어져 있으며, 특정 질병이나 증상은 정상 데이터보다 적게 나타나는 불균형한 클래스 분포가 일반적이라는 특징이 있다. 특성 간의 복잡한 관계가 형성되기 때문에 단순한 단일모델만으로는 데이터의 패턴을 충분히 학습하기 어렵다. 따라서 헬스케어 데이터를 활용한 연구는 신경망과 같은 복잡한 모델을 이용한다[3]-[5].
복잡도가 높은 모델을 구축하는 대표적인 머신러닝 기법에는 앙상블 기법이 있다. 앙상블 기법은 모델의 복잡도가 높아 일반적으로 단일모델보다 성능이 높다고 알려져 있다[6]. 앙상블 모델을 구축하는 기법에는 대표적으로 배깅, 부스팅, 스태킹, 보팅이 있다. 앙상블 기법 중 보팅 기법과 스태킹 기법은 다양한 모델을 조합해 사용할 수 있다. 보팅 기법은 여러 유형의 모델을 조합해 결정할 수 있지만, 해당 방식의 경우 사용할 모델을 미리 결정하는 과정이 선행된다. 따라서 어떤 모델을 사용해야 좋을지 선택하기 위해서는 머신러닝 기반 지식이 필요하다. 반면에 스태킹 앙상블 기법은 다양한 모델들을 계층적으로 조합하고 예측을 결합하기 때문에 모델 선택을 자동화할 수 있다. 또한, 불균형한 클래스를 가진 분류 문제에서 성능이 단층 스태킹 앙상블 모델보다 다층 스태킹 앙상블 모델에서 더 나은 성능을 보였다[7][8].
복잡한 증상 간의 다양한 상호 관계를 고려하려면 헬스케어 분야에 대한 도메인 지식이 필요하다. 그러나 머신러닝 기술은 데이터 전처리, 변수 선택, 모델 선택, 학습 및 하이퍼파라미터 튜닝과 같은 복잡한 프로세스를 반복하게 되어 비효율적이다. 이 때문에 머신러닝 기술에 대한 도메인 지식이 부족한 다양한 분야의 연구자들은 사용에 어려움을 겪는다. 따라서 머신러닝 프로세스의 생산성과 효율을 높이고, 다양한 분야의 연구자들이 쉽게 머신러닝 모델 개발을 할 수 있도록 프로세스를 자동화하기 위해 등장한 기술이 AutoML(Automated Machine Learning)이다. 현재 다양한 AutoML 프레임워크들이 서비스되며 대중화되었지만, 기존의 프레임워크에서 앙상블 기법을 이용해 다양한 모델을 조합하는 방법은 여전히 한정적이다. 또한, 특정 분야의 데이터에 초점을 맞추어 자동으로 모델을 구축하고자 하는 연구는 아직 부족하다.
본 논문에서는 헬스케어 데이터에 맞춰 변수 선택과 적응형 다층 스태킹 앙상블 모델을 구축하는 과정을 자동화한 알고리즘을 제안한다. 논문의 구성은 다음과 같다. 2장에서는 관련 연구로 기존 AutoML 프레임워크 및 다양한 분야에서 AutoML 연구 동향에 관해 기술한다. 3장은 본 논문에서 제안하는 적응형 다층 스태킹 앙상블 구축 방법에 대해 자세하게 기술한다. 4장에서는 실험 환경 및 다양한 헬스케어 데이터를 이용해 제안하는 방법과 다른 AutoML 도구들을 비교한 실험에 관해 기술한다. 마지막으로 5장에서는 결론과 함께 본 연구가 갖는 한계점 및 향후 연구에 관하여 기술한다.
Ⅱ. 관련 연구
AutoML은 복잡한 머신러닝 프로세스를 자동화하며 데이터 분석가의 불필요한 반복 작업을 하지 않도록 해주고, 도메인 전문가가 머신러닝 및 통계적 지식이 없더라도 머신러닝 어플리케이션을 구축할 수 있도록 만든다. 현재 다양한 오픈소스 AutoML의 프레임워크가 제공되고 있다.
TPOT(Tree-based Pipeline Optimization Tool)[9]은 유전 알고리즘을 이용해 머신러닝 파이프라인을 최적화하는 AutoML 프레임워크이다. TPOT의 진화 전략은 불규칙한 탐색 공간을 처리하기 때문에 탐색 과정 중 많은 후보 파이프라인은 유효하지 않을 수 있다. 따라서 사용할 때 시간적 비용이 많이 들 수 있다.
Pycaret은 파이썬 기반의 머신러닝 프로세스를 자동화하는 오픈소스 프레임워크이다. 데이터 전처리, 모델 선택, 파라미터 튜닝 작업, 앙상블 모델 구축 작업들을 자동화하며 간단한 코드로도 모든 작업이 가능하다는 특징이 있다. 다양한 모델의 결과를 비교할 수 있다는 장점이 있지만 간단한 코드로 실행하기 때문에 단일모델의 성능만을 이용해 스태킹 앙상블 모델을 구축하는 등 세부적으로 커스터마이징하기 힘들다는 단점이 있다.
Autogluon[10]은 자동으로 데이터 전처리를 수행하여 결측치 처리, 특성 스케일링, 범주형 변수 인코딩 등과 같은 작업을 처리한다. 여러 모델을 구성하여 성능을 비교하고 다층 스태킹 앙상블을 만드는 기능 제공한다. ResNet과 MobileNet 등에서 사용하는 Skip-Connection 구조를 응용해 스태킹 앙상블을 구성한다. 하지만 6개의 모델만 이용하기 때문에 구축될 수 있는 모델 구조가 한정적이다.
기존 AutoML 프레임워크는 시간적 비용이 많이 들거나, 세부적으로 모델의 다양성을 확보하기 힘들다는 단점이 존재한다.
이러한 AutoML은 다양한 분야에서 이용된다. 그중에서도 헬스케어 분야에서는 질병의 진단이나 증상의 판별에 AutoML이 주로 사용된다. T. Anwar는 3D CT 스캔을 예측에 AutoML 활용해 COVID19 진단하고자 했다[11]. N. K. Tran, et al.은 화상 환자 중 패혈증 증상을 보이는 환자를 찾기 위해 AutoML 플랫폼 MILO를 제안했다[12]. K. W. Wan, et al.은 초음파 유방 이미지 데이터로부터 양성 혹은 악성 유방 병변을 판별하는 AutoML 모델을 추천했다[13].
헬스케어 데이터는 특성 간의 복잡한 관계와 불균형한 데이터 분포를 따른다. 따라서 헬스케어 데이터를 활용하는 연구에서는 인공신경망과 딥러닝 기법 등과 같은 복잡도가 높은 모델을 구축하여 사용한다[14]. A. H. Khan, et al.은 심전도 영상으로부터 심장 질환을 판별하기 위해 MobileNet v2 기반의 심층 신경망을 제안했다[15]. F. J. Díaz-Pernas, et al.은 CNN 기반의 분류 모델을 이용해 MRI 이미지에서 뇌종양 이미지를 추출하고 분류했다[16]. 딥러닝 기법을 활용한 연구들은 비정형 데이터인 이미지나 영상과 같은 데이터를 주로 다루었다. A. A. Bataineh, et al.은 심장병 발병을 예측하기 위해 MLP-PSO 하이브리드 알고리즘을 제안했다[17]. T.-H. Lim, et al.은 군 의료 데이터로부터 3가지 주요 국방 의료 질병을 진단하는 인공신경망 기반 주요 질병 진단 시스템을 제안했다[18].
그러나 기존의 헬스케어 분야 AutoML 연구와 인공신경망 기반 분류 모델 연구들은 대체로 단일모델을 중점적으로 다루어, 다양성을 보장하지 못하는 한계가 있다. 본 연구에서는 다양한 헬스케어 데이터에 대응할 수 있도록 적응적인 방법을 도입하고, 모델의 다양성을 확보하여 성능 향상을 위해 다층 스태킹 앙상블 알고리즘을 자동화하는 방법을 제안한다.
Ⅲ. 적응형 다층 스태킹 앙상블 모델 구축
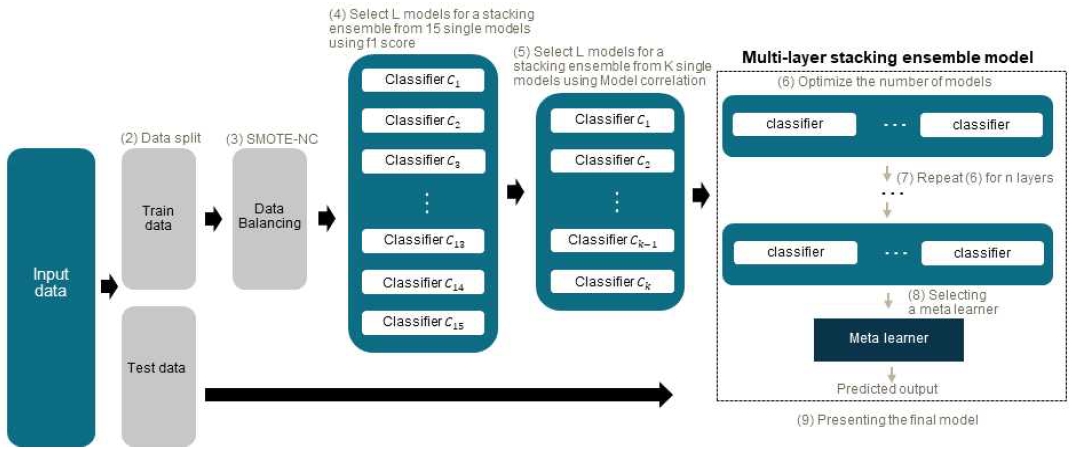
본 논문에서 제안하는 헬스케어 데이터에 맞춰 변수를 선택하는 과정과 적응형 다층 스태킹 앙상블 모델을 탐욕적인 방법을 이용해 구축하는 과정에 대한 자동화 절차는 그림 1과 같다. 입력 데이터셋에 맞추어 모델을 구축하기 위해 Step-wise feature selection과 Fisher’s score를 통해 변수를 선별해 활용한다. 불균형한 클래스 분포는 일반적인 헬스케어 데이터의 특성이다. 따라서 클래스 분포를 확인하고, 불균형한 데이터의 경우에는 SMOTE-NC (Synthetic Minority Over-sampling TEchnique-Nominal Continuous)를 활용해 소수의 데이터를 증강하는 과정을 추가해 모델의 정확도를 항상 시킨다. 이후 자주 사용되는 분류 모델을 학습 방법을 기준으로 15가지 선정해 모델의 성능과 모델들의 예측값을 통해 구한 모델 간의 상관관계를 이용해 다층 스태킹 앙상블을 구축하는 데 사용할 모델을 선정한다. 마지막으로 모델의 성능을 기준으로 탐욕적으로 모델의 개수를 늘려가며 구한 최적의 모델 조합을 한 층으로 하며, 이전 층과 현재 층의 모델 구성 조합이 같지 않을 때까지 층을 늘려가는 과정을 자동화한다.
3.1 데이터 전처리
학습을 수행하기 전 데이터 전처리는 결측치 처리, Feature selection, Oversampling 등 총 3가지 단계를 거쳐 진행된다.
먼저 데이터에 결측치 여부를 파악하고, 결측치가 존재하는 경우 결측치 비율에 따라서 결측치 처리 방법을 달리 진행한다. 전체 데이터에서 결측치가 10% 미만인 변수는 결측값 대치를 사용한다. 범주형 변수일 경우 최빈값을 이용하며, 연속형 변수일 경우 중앙값을 이용해 대체한다. 결측치 비율이 10% 이상 50% 미만일 경우에는 KNN을 이용해 모델 기반의 결측치 대치를 진행한다. K의 개수를 변경하면서 KS 검증(Kolmogorov-Smirnov Statistics test)을 이용해 결측치 처리 전후의 데이터 분포를 p-value 값을 이용해 비교한 후 K의 값을 가장 유의미한 값으로 결정한다. 결측치가 50% 이상일 경우에는 해당 변수를 삭제한다.
Feature selection을 진행하며 Filter methods 중에서 Fisher’s score, Wrapper methods 중에서 Step-wise feature selection을 함께 이용해 변수를 선택한다. 방법이 다른 두 가지를 함께 사용하며 다양한 관점에서 변수를 선택한다.
| (1) |
Fisher’s score를 구하는 식은 (1)과 같다. Si란 i 번째 특징의 점수인 Fisher’s score이다. μij와 pij는 각각 j번째 클래스에서 i 번째 특징의 평균과 분산, nj는 j 클래스의 샘플 수, μi는 i 번째 특징의 평균이다. 이를 통해 클래스 간 특징의 평균 간의 분산으로 나타나는 클래스 간 분산과 각 클래스 내에서 특징값들의 분산으로 나타나는 클래스 내 분산을 통해 각 특징에 순위를 매긴다. 즉, 특징마다 클래스 간 분산과 클래스 내 분산의 비율을 계산한다. 계산된 Fisher’s score를 기준으로 정렬한다. 상위 50%에 속하는 변수를 선택해 변수 집합을 생성한다.
모든 변수가 선택되지 않은 모델에서 출발해 변수를 하나씩 추가하는 Forward selection을 시행하고, 이후 추가된 변수에서 하나씩 제거하는 Backward elimination을 시행하는 Step-wise feature selection을 진행한다. 학습 방법이 다른 4가지 분류기(LDA, SVM, SGD, CatBoost)를 이용해 Feature selection을 진행한다. 4가지 분류기는 다양한 학습 방법을 겹치지 않도록 하여 한 가지 학습 방법에 과도하게 적합하지 않도록 한다. 이 중 세 가지 분류기에서 공통으로 선택된 변수를 이용해 최종적으로 집합을 만든다.
Fisher’s score에서 선택된 변수 집합과 Step-wise feature selection을 통해 선택된 변수 집합에서 공통으로 선택한 변수를 선택해 최종적으로 모델의 학습에 사용한다.
클래스 불균형을 해결하는 샘플링 방법에는 크게 두 가지 방법이 있다.
Oversampling은 소수 범주의 레이블의 데이터 수를 증강해 데이터 불균형을 해소하는 방법을 말한다. Undersampling은 다수 범주의 레이블의 데이터 수를 감소시키는 방법을 말한다. Undersampling을 이용하면 학습에 이용할 데이터 수가 급격하게 줄어 성능이 떨어질 가능성이 존재한다. 따라서 본 연구에서는 소수 범주 레이블의 학습 데이터에 Oversampling을 사용한다. 데이터에 범주형 변수가 포함되어 있을 것을 고려해 Oversampling 기법 중에서 SMOTE-NC(Synthetic Minority Over-sampling Technique for Nominal and Continuous)를 적용한다. SMOTE-NC란 낮은 비율 클래스 데이터들의 근접 이웃을 이용해 데이터를 증강하는 방법으로, SMOTE 방식으로 데이터를 증강하고 그룹화한 후 해당 클래스에 가까운 범주형 변숫값을 붙이는 방식이다. 본 연구에서는 다수의 레이블의 데이터에서는 데이터 증강을 시행하지 않고 소수의 레이블에서만 Oversampling을 진행한다.
3.2 적응형 다층 스태킹 앙상블 모델
모델을 자동으로 구성해 주는 알고리즘을 구축하기 전에 스태킹 앙상블을 구성하기 위해 사용될 분류 모델을 선정한다. 일반적으로 자주 이용되는 기존의 분류기 중 15가지 모델을 사용한다. 사용한 모델은 표 1과 같다.
이렇게 기존에 설정해 둔 15개의 분류 모델 중에서 사용할 데이터에 적합한 모델 집합을 자동으로 선정한다. 만약 단일모델의 성능이 다른 단일모델의 성능에 비해 지나치게 낮다면 스태킹 앙상블을 통해 성능을 향상시키기 어렵다. 따라서 우선적으로 단일모델의 성능을 기준으로 일차적으로 모델을 선정한다. 이때, 데이터의 불균형성을 고려하여 정밀도와 재현율을 동시에 고려하는 F1-score를 사용한다. K-fold 교차 검증을 수행하며 모델의 평균 F1-score를 계산한다. 그 후, 평균 F1-score가 미리 설정한 임곗값 이상의 성능을 나타내는 모델들만을 자동으로 K개 선정한다. 이 과정은 알고리즘 1과 동일하다.
스태킹 앙상블에서는 다양한 모델로부터 예측을 도출하고 결합하여 사용한다. 이는 각 모델의 예측 오차를 서로 상쇄시켜 전체 앙상블의 성능을 높이고 모델의 다양성을 유지하는 강점을 가진다. 선택한 K개의 모델의 예측값을 비교하여 모델 간의 상관관계를 기준으로 다시 L개의 모델을 선택하는 과정은 알고리즘 2와 같다. 먼저, K개의 모델 중에서 두 모델씩 모든 가능한 조합을 만들어 각 조합에 대해 두 모델의 예측값을 구한다. 그런 다음, 두 모델의 예측값을 비교하여 상관계수를 계산한다. 여기서는 피어슨 상관계수를 사용하며, 일반적으로 값이 0.7 이상이면 두 변수 간에 높은 상관관계가 있다고 판단한다.
따라서 상관계수가 0.7 이상인 두 모델을 찾아 성능이 낮은 모델을 K개의 모델에서 제외한다. 이러한 두 단계를 통해 최종적으로 사용할 모델을 L개 선정한다.
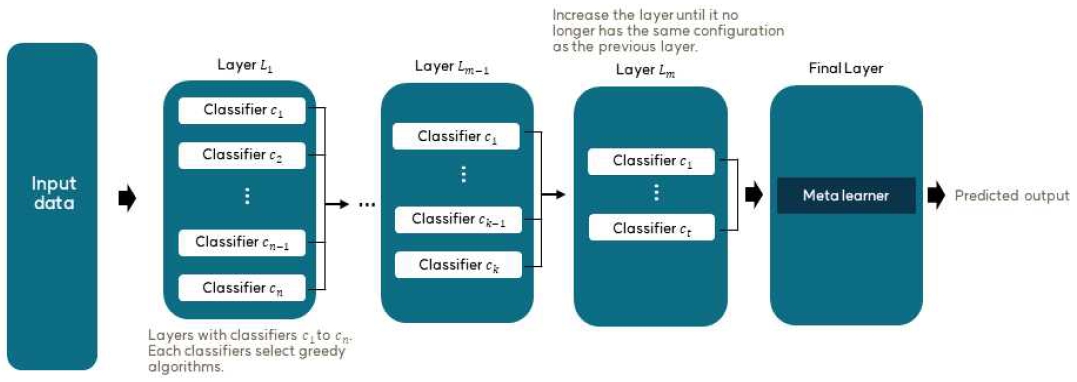
본 연구에서 제안하는 방법으로 구축될 다층 스태킹 앙상블 모델의 전체구조도는 그림 2와 같다. 또한, 다층 스태킹 앙상블 모델을 자동으로 구축하는 세부적인 과정은 알고리즘 3과 같다. 아래는 알고리즘 3의 주요 단계에 대한 설명이다.
먼저 알고리즘 1과 알고리즘 2를 통해 구한 L개의 모델을 성능 순으로 정렬한다. 정렬된 모델 중에서 성능이 가장 좋은 모델부터 시작하여 모델의 개수를 점진적으로 증가시키며 최적의 성능을 가지는 모델 조합을 탐색한다. 사용자가 설정한 임곗값만큼 모델을 추가했는데도 성능 향상이 없다면 가장 성능이 좋았던 조합을 최적이라고 판단하고, 해당 조합을 그 층의 최적 단일 층으로 선택한다. 이후, 층수를 한 층 늘리고 앞의 과정을 반복한다. 생성된 층의 모델 조합이 이전 층의 모델 조합과 같다면 반복을 종료하고 최적의 다층 스태킹 앙상블 모델 구조를 반환한다.
마지막 층은 각 모델의 결과를 결합해 최종적인 결괏값을 도출하는 Meta classifier로 구성한다. 본 연구에서 구축한 다층 스태킹 앙상블 모델은 다층 스태킹 앙상블이라는 특징상 모델의 복잡도가 높을 것으로 예상된다. 따라서 모델의 복잡도가 높아지면서 발생할 수 있는 데이터의 과적합 문제를 방지하고자 Meta classifier로는 Logistic regression을 활용한다.
Ⅳ. 실험 및 성능 평가
본 논문에서는 Kaggle에 공개된 헬스케어 데이터세트 두 가지와 한국인 유전체역학조사의 지역사회 기반 코호트(안산, 안성) 데이터세트를 이용해 실험을 진행했다. 학습과 평가 데이터는 클래스 분포를 고려하며 7:3 비율로 나누었다. 또한, 모델의 성능 평가 지표로 정확도, 정밀도, 재현율, F1-score를 이용했고 사용한 데이터가 전부 이진 분류이기 때문에 AUC(Area under the ROC Curve)를 추가로 사용했다. 실험은 Feature selection 유무, Oversampling 유무, 기존 AutoML 프레임워크 세 가지와의 성능 비교까지 세 가지 측면에서 수행하였다.
4.1 데이터
본 연구에서 이용한 데이터세트의 분포는 표 2와 같다. 첫 번째 데이터는 Kaggle에서 제공하는 “흡연신체 신호”[19]이다. 해당 데이터는 기본적인 건강 생체 신호를 이용해 흡연 여부를 분류하는 이진 분류 데이터며 레이블의 비율이 균형을 이루는 데이터이다. 전체 데이터는 55,692개로, 건강 생체 신호 및 정보로 구성된 26가지 독립변수와 흡연 여부를 알 수 있는 종속변수로 구성된다.
두 번째 데이터는 Kaggle에서 제공하는 “심장 질환 건강 지표 데이터셋”[20]을 이용했다. 해당 데이터는 심장 질환 유무의 이진 분류에 사용하기 위해 구성되었으며 2015년 BRFSS(Behavioral Risk Factor Surveillance System)의 전화 설문조사 응답 데이터를 활용했다. BRFSS는 CDC(Centers for Disease Control and Prevention)에서 매년 건강 관련 위험 행동, 만성 건강 상태 및 예방 서비스 이용에 대한 내용을 조사하는 미국의 건강 관련 전화 설문조사 자료이다. 원본 데이터는 441,455명의 응답과 330개의 특징으로 구성되어 있다. 사용할 데이터는 253,680명의 응답이며 21개의 독립변수와 1개의 종속변수로 구성된다. 레이블의 비율은 불균형하게 나타난다.
세 번째 데이터는 한국인 유전체역학조사의 지역사회 기반 코호트(안산, 안성) 데이터이다. 해당 데이터는 당뇨 발생 여부를 관측한 데이터로, 안산과 안성 두 지역의 40대 이상의 중년 주민을 대상으로 조사한 데이터이며 당뇨 가족력 등의 기초자료 및 혈액검사 자료와 같은 검사자료 등으로 구성되어 있다. 해당 데이터는 10,030명의 조사 결과이며 118개의 독립변수와 1개의 종속변수로 구성된다. 구성하는 레이블의 비율이 차이 나는 불균형 데이터이다.
4.2 Feature selection 실험
본 연구에서 이용하는 다층 스태킹 앙상블 모델은 모델의 복잡도가 높고, 데이터세트가 다양한 검사나 신상 정보와 같은 다양한 변수들로 구성됐다. 이러한 특성상 모델이 데이터세트에 과적합 될 가능성이 높다. 사전에 사용할 변수를 선정해 데이터의 복잡도를 낮추고 과적합을 방지하고자 했다. 변수 간의 복잡한 관계가 얽혀있는 헬스케어 데이터의 특성상 아무 변수나 제거하게 되면 모델의 성능이 오히려 떨어질 수 있다. 따라서 Feature selection을 시행한 경우와 시행하지 않았을 때를 비교하는 실험을 진행해 해당 과정의 타당성을 검증했다.
본 실험에서는 제안하는 방법을 통해 구축한 모델을 각기 다른 데이터로 학습시킨 후 Fisher’s score와 Step-wise feature selection을 이용해 변수를 추출했다. 정확한 비교를 위해 그 외의 실험 환경은 전부 동일하게 진행하였다.
흡연 신체 신호 데이터세트에서 종속변수를 포함한 27가지 변수 중 Feature selection 과정을 통해 표 3과 같이 종속변수를 포함해 13가지를 선택했다. 표 4는 Feature selection을 진행한 실험과 시행하지 않은 실험의 비교 결과이다. 재현율(0.7766, 0.7674)을 제외한 모든 평가 지표에서 Feature selection을 시행한 모델의 결과에서 시행하지 않은 모델의 결과보다 좋게 나온 것을 확인할 수 있었다.
심장 질환 건강 지표 데이터세트에서 선택된 변수는 표 5와 같다. 기존의 22개의 변수 중 종속변수를 포함해 11가지 선택되었다. 표 6은 Feature selection 시행 여부에 따른 성능 비교표이다. 성능 비교 실험 결과 정확도(0.8454, 0.7971)를 제외한 모든 평가 지표상으로 Feature selection을 시행했을 때 성능이 올라간 것을 확인할 수 있었다.
한국인 당뇨병 코호트 데이터세트에서는 기존의 119개의 변수 중 종속변수를 포함해 28가지가 선택되었다. 구성된 변수 목록은 표 7과 같다.
성능 비교 결과는 표 8과 같다. 정확도(0.8711, 0.8518)와 정밀도(0.6500, 0.4762)를 제외한 대부분의 평가 지표에서 Feature selection을 진행한 결과가 더 좋은 것을 확인 할 수 있었다.
세 가지 데이터세트를 통한 비교 실험 결과 헬스케어 데이터에서 Feature selection을 시행했을 때 대다수의 평가 지표에서 성능이 더 좋게 나오는 것을 알 수 있었다.
4.3 Oversampling 실험
본 연구에서는 불균형한 레이블 문제를 해소하고자 Oversampling 기법의 하나인 SMOTE-NC를 이용했다. 하지만 헬스케어 데이터의 특성상 데이터 증강을 할 시 성능이 떨어지거나 데이터의 특성을 잘 반영하지 못할 것이라는 우려가 있다. 따라서 불균형한 레이블의 데이터세트 두 가지를 이용해 Oversampling 시행 여부에 대한 비교 실험을 진행하고 성능을 평가하였다.
심장병 발생 예측 데이터세트와 당뇨병 코호트 데이터세트에서 SMOTE-NC 시행 여부를 비교한 결과는 표 9, 표 10과 같다. 심장 질환 건강 지표 데이터세트의 경우 정확도(0.8506, 0.7971) 외에 모든 지표에서 성능이 좋았다. 당뇨병 코호트 데이터세트의 경우 모든 지표에서 좋은 성능을 보여주었다.
또한, 표 9 및 표 10의 결과를 살펴보면 두 가지 데이터에서 재현율이 높게 나온 것을 볼 수 있었다. 헬스케어 데이터에서는 양성 레이블에서 질병의 발병 여부를 탐지한다. 따라서 불균형한 레이블의 데이터에서 실제 양성 레이블을 잘 탐지하는 것은 이점을 가진다.
4.4 흡연 여부 예측 데이터세트
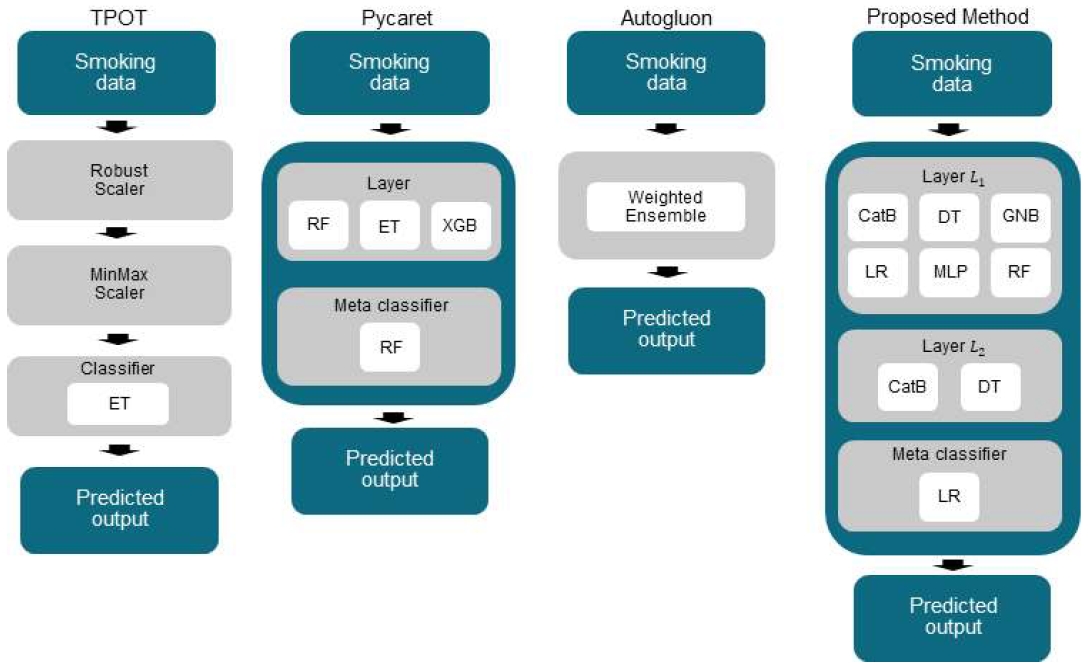
제안하는 방법의 성능 평가를 위해 세 가지 AutoML 프레임워크(TPOT, Pycaret, Autogluon)를 이용해 비교 실험을 진행했으며 모두 같은 데이터를 사용했다. 흡연 여부 예측 데이터는 두 가지 레이블의 비율이 비슷하므로 학습 데이터에 Oversampling을 진행하지 않았다.
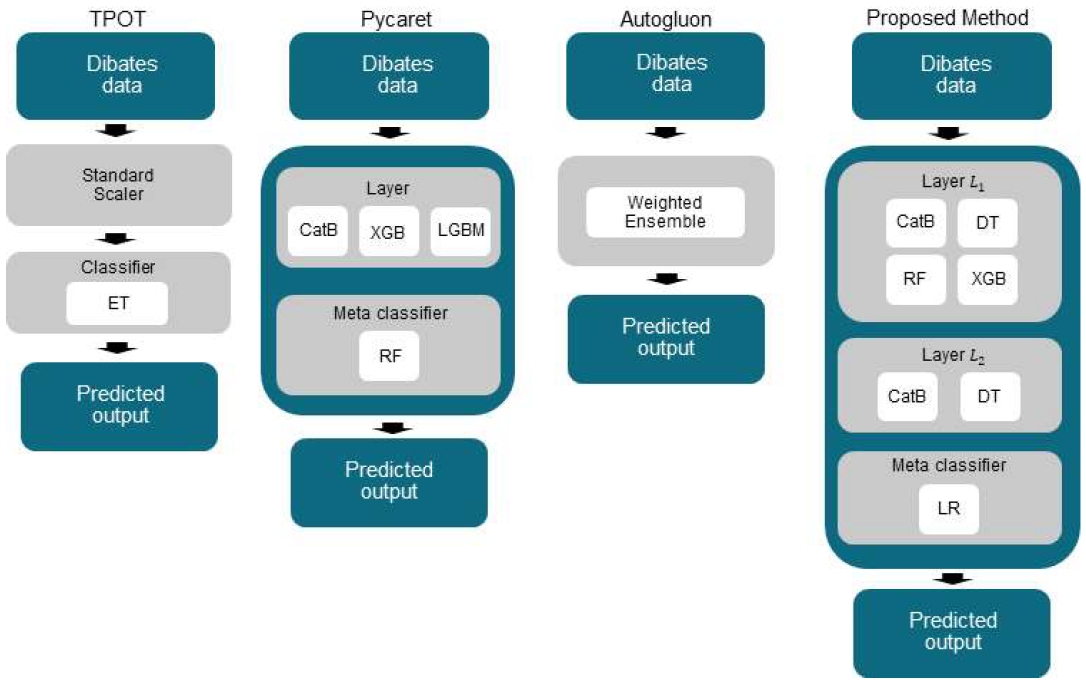
그림 3은 타 AutoML 프레임워크 및 제안하는 방법을 통해 구축된 최적의 모델 구조이다. TPOT에서 도출한 최적의 모델은 Robust Scaler와 MinMax Scaler를 거친 데이터를 Extra Tree를 이용해 분류했다. Pycaret은 단일모델에서 가장 성능이 좋았던 Random Forest, Extra Tree, XGBoost를 한 층으로 하는 단층 스태킹 앙상블을 도출했다. 세 가지 모델에서 나온 예측값을 Meta classifier인 Random Forest를 이용해 결합해 최종적으로 분류했다. Autogluon의 경우는 프레임워크의 내부에서 자체 알고리즘을 이용해 구축한 weighted ensemble 모델을 최적의 모델로 도출했다. 본 논문에서 제안하는 방법은 다층 스태킹 앙상블을 구축했다. 첫 번째 층에서 6가지 모델(CatBoost, Decision Tree, Gaussian NB, Logistic regression, MLP, Random Forest)의 결괏값을 결합해 다음 충의 입력으로 이용했으며, 두 번째 층에서 CatBoost, Decision Tree를 사용했다. 마지막 층에서 Meta classifier로 Logistic regression을 이용해 예측값을 결합해 최종 결괏값을 도출하였다.
표 11은 각 AutoML 프레임워크와 제안하는 방법에서 도출된 모델을 이용해 성능을 평가한 결과이다. 실험 결과 Autogluon에서 도출된 모델이 재현율(0.7934)과 F1-score(0.7679)에서 가장 좋은 성능을 보였다. 재현율과 F1-score를 제외한 나머지 평가 지표에서는 제안하는 모델이 가장 좋은 성능을 보였다. 재현율과 F1-score에서도 Autogluon 다음으로 좋은 성능을 보여주었다.
4.5 심장병 발생 예측 데이터세트
앞에서 실험한 흡연 여부 예측 데이터와 다르게 심장병 발생 예측 데이터는 클래스의 분포가 불균형한 데이터이다. 따라서 학습 데이터와 평가 데이터를 나눈 후, 학습 데이터에 대해서만 SMOTE-NC를 시행했다. 타 AutoML 프레임워크를 사용할 때도 SMOTE-NC를 적용한 학습 데이터로 학습시켰다.
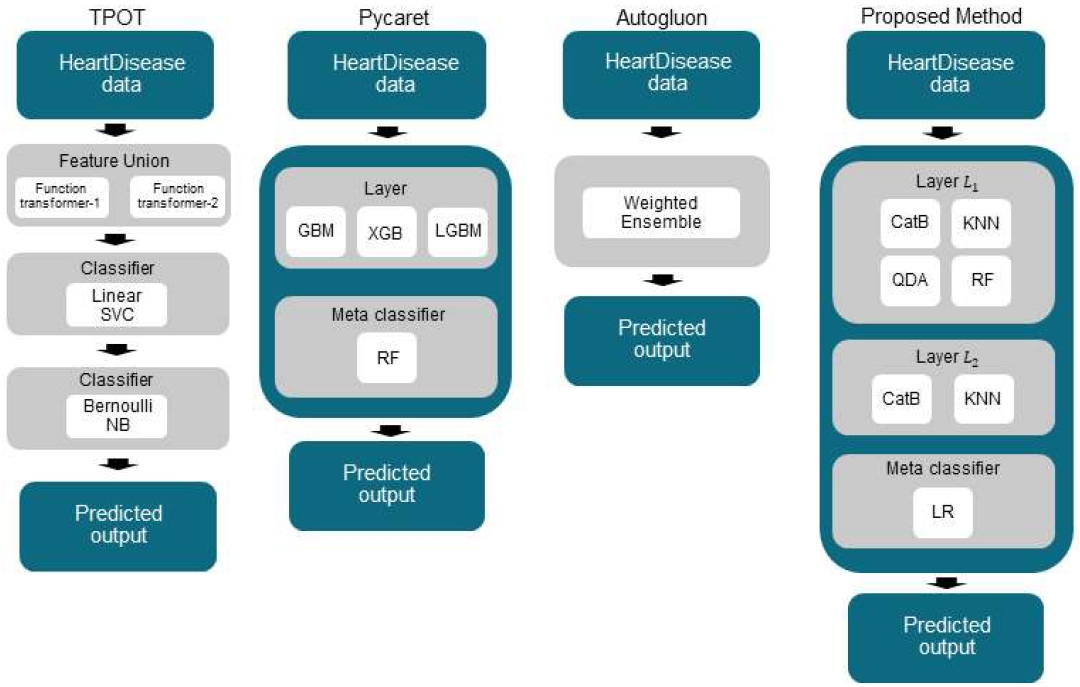
실험을 통해 구축한 모델은 그림 4와 같다. TPOT에서 구축한 모델은 입력으로 들어온 데이터에서 Feature union을 이용해 각기 다른 Feature를 추출하고, Linear SVC와 Bernoulli Naīve Bayes를 차례로 이용해 최종 예측값을 도출한다. Pycaret을 이용해 자동으로 머신러닝 프로세스를 진행한 결과로 구성된 모델은 단층 스태킹 앙상블 모델이다. 스태킹 앙상블은 총 세 가지 모델(Gradient Boosting Machine, XGBoost, Light Gradient Boosting Machine)로 층이 구성되어 있다. 세 모델을 통해 나온 예측값을 결합하는 Meta classifier로는 Random Forest을 이용했다. Autogluon은 weighted ensemble 모델을 최종적으로 제안했다. 제안하는 방법은 다층 스태킹 앙상블 모델을 구축했다.
첫 번째 층에서는 4개의 모델(CatBoost, KNN, Quadratic Discriminant Analysis, Random Forest)의 조합으로 구성되었으며, 두 번째 층에서는 2개의 모델(CatBoost, KNN)을 최적의 층으로 사용했다. Meta classifier로는 Random Forest를 사용했다.
비교 실험의 성능 평가 결과는 표 12와 같다. 정확도(0.9078)에서는 Autogluon, 정밀도(0.5763)에서는 Pycaret이 가장 좋은 성능을 보여주었다. 불균형한 레이블의 데이터세트에서 중요한 재현율(0.5696)과 F1-score(0.3459), AUC(0.6998)에서는 제안하는 모델이 가장 높은 성능을 보였다.
4.6 한국인 당뇨병 코호트 데이터세트
한국인 당뇨병 코호트 데이터는 클래스의 분포가 불균형하며, 다수의 결측치 등이 포함된 실제 헬스케어 데이터이다. 따라서 학습을 진행하기 전 결측치 비율에 따라 결측치 대치를 시행했다. 결측치 비율이 10% 이상 50% 미만인 경우, KNN을 활용하여 결측치를 대체했다. 이때 KS 검증을 통해 p-value 값이 유의미했던 3을 K 값으로 선택하였다. 결측치 대치 후 학습 데이터에 SMOTE-NC를 적용해 불균형한 클래스를 완화했다.
그림 5는 한국인 당뇨병 코호트 데이터 세트를 통해 AutoML 프레임워크와 제안하는 방법에서 자동으로 생성한 모델을 나타낸다. TPOT은 입력 데이터를 Standard Scaler를 활용하여 적절한 스케일로 조정했다. 이후 조정된 데이터를 Extra Tree의 입력으로 활용하여 최종 결괏값을 도출하는 것을 최적화된 과정으로 제안했다. Pycaret은 단층 스태킹 앙상블 모델을 도출했다. 이 모델은 단일 모델로 사용했을 때 가장 성능이 좋았던 세 가지 모델(CatBoost, XGBoost, Light Gradient Boosting Machine)을 스태킹한 구조를 가지고 있다. 최종 분류기로 Random Forest를 이용했다. Autogluon은 최적화된 모델로 자체 내부 알고리즘으로 생성한 weighted ensemble 모델을 제안했다. 제안하는 방법은 다층 스태킹 앙상블 모델을 도출했다. 생성한 모델은 총 세 층으로 구성되어 있다. 앞 층에서 얻은 각 모델의 예측값이 다음 레이어의 입력값으로 활용된다. 첫 번째 레이어는 CatBoost, Decision Tree, Random Forest, XGBoost의 조합으로 이루어진 층을 형성했다. 두 번째 층에서는 CatBoost와 Decision Tree로 구성했다. 최종 레이어에서는 Logistic regression을 Meta classifier로 활용하여 예측을 수행했다.
표 13은 세 가지의 기존 AutoML 프레임워크를 이용하여 도출한 최적의 모델과 제안하는 방법을 통해 구축한 최적의 다층 스태킹 앙상블 모델의 비교 실험 결과이다. Autogluon이 정확도(0.8671), 정밀도(0.5596)에서 가장 좋은 성능을 보인 것을 알 수 있다. 불균형한 레이블의 데이터세트에서 중요한 재현율(0.6221)은 TPOT에서 가장 좋은 성능을 보였으며, 제안하는 방법을 통해 구축한 다층 스태킹 앙상블(0.4762)이 그 뒤를 이었다. 다만 TPOT의 경우에는 재현율에서 가장 좋은 수치를 보였지만, 정밀도(0.3125)는 네 가지 비교군 중 가장 떨어지는 수치를 보였다. 반면에 제안하는 방법을 통해 만든 다층 스태킹 앙상블의 경우에 정밀도(0.4762)와 재현율(0.4762)이 비슷하게 좋은 성능을 보였다. 또한 F1-score(0.4728)와 AUC(0.7008)에서 네 가지 모델 중에서 가장 우수한 성능을 나타냈다.
Ⅴ. 결론 및 향후 과제
본 논문에서는 헬스케어 데이터를 이용해 이진 분류를 하는 다층 스태킹 앙상블 모델 자동화 방법을 제안했다. 제안된 방법은 데이터의 특성을 고려하여 결측치 처리, 특성 선택, 클래스 불균형 처리, 그리고 최적의 모델 조합 탐색을 자동화하였다. 실험 결과, 제안된 방법은 다양한 헬스케어 데이터에서 기존 AutoML 프레임워크와 비슷하거나 높은 예측 성능을 보여주었다.
제안된 방법의 자동화 과정에서는 먼저 데이터의 결측치 비율을 확인하고, 결측치에 대한 대치를 자동으로 시행하였다. 이어서 Step-wise feature selection과 Fisher’s score를 결합하여 데이터의 특성을 줄이고자 하는 Feature selection이 이루어졌다. 데이터세트의 클래스 분포가 불균형할 경우에만 학습 데이터에 SMOTE-NC를 적용하여 Oversampling을 진행했다. 미리 선정한 모델 후보군 중 F1-score와 모델 간 상관관계를 고려하여 최적의 모델 조합을 자동으로 찾아내었다. 마지막으로, 최적의 모델 조합에서 성능을 기준으로 점진적으로 하나씩 늘려가며 최적의 조합의 레이어와 레이어의 수를 탐색했다.
실험에서는 세 가지 다른 헬스케어 데이터를 활용하여 모델을 평가하였다.
균형 데이터와 불균형 데이터 모두에서 제안하는 모델은 AutoML 프레임워크에 버금가는 정확도를 보여주었다. 그러나 불균형 데이터의 경우 낮은 F1-score를 보인다는 한계점이 나타났다.
본 연구에서 제안하는 방법은 하이퍼 파라미터 튜닝까지 진행하는 타 AutoML 프레임워크와 다르게 모델 선택까지만 시행한다. 또한, 이진 분류 데이터에서만 사용할 수 있다.
본 연구의 방법은 하이퍼 파라미터 튜닝까지 진행하는 다른 AutoML 프레임워크와 구별되며, 현재는 이진 분류에만 활용 가능하다. 따라서 향후 연구 방향으로는 알고리즘의 발전을 통해 다진 분류도 가능하게 하고, 하이퍼 파라미터 튜닝까지 자동화할 수 있는 범용적인 방법을 모색할 것이다.
Acknowledgments
본 논문은 정부(교육부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임(No. 2021R1G1A1006381)
References
- M. Amini and A. Rahmani, "How Strategic Agility Affects the Competitive Capabilities of Private Banks", International Journal of Basic and Applied Sciences, Vol. 10, No. 1, pp. 8397-8406, Apr. 2023.
- K. Sharifani and M. Amini, "Machine Learning and Deep Learning: A Review of Methods and Applications", World Information Technology and Engineering Journal, Vol. 10, No. 07, pp. 3897-3904, May 2023.
-
J. Ha, K. Kong, and D. Park, "Deep Learning Framework for Predicting Alzheimer‘s Disease using Multi-omics Data", The Journal of Korean Institute of Information Technology, Vol. 20, No. 7, pp. 29-37, Jul. 2022.
[https://doi.org/10.14801/jkiit.2022.20.7.29]

-
J.-H. Ha, "Autoencoder-based Disease-related miRNA Prediction Research using Deep Learning", The Journal of Korean Institute of Information Technology, Vol. 20, No. 6, pp. 33-40, Jun. 2022.
[https://doi.org/10.14801/jkiit.2022.20.6.33]

-
J. Mun, S. Kim, M. J. Kim, J. Ryu, S. Kim, and M. Chung, "Automatic detection and severity prediction of chronic kidney disease using machine learning classifiers", Phonetics and Speech Sciences, Vol. 14, No. 4, pp. 45-56, Dec. 2022.
[https://doi.org/10.13064/KSSS.2022.14.4.045]

- The 10 main takeaways from MLconf SF, https://tryolabs.com/blog/2016/11/18/10-main-takeaways-from-mlconf, [accessed: Sep. 08, 2023]
-
L. R. Kalabarige, R. S. Rao, A. Abraham, and L. A. Gabralla, "Multilayer stacked ensemble learning model to detect phishing websites", IEEE Access, Vol. 10, pp. 79543-79552, Jul. 2022.
[https://doi.org/10.1109/ACCESS.2022.3194672]

-
M. Zulfiker, N. Kabir, A. A. Biswas, and P. Chakraborty, "Predicting Insomnia Using Multilayer Stacked Ensemble Model", Sleep Research Journal, Vol. 1440, pp. 338-350, Apr. 2021.
[https://doi.org/10.1007/978-3-030-81462-5_31]

-
R. S. Olson, N. Bartley, R. J. Urbanowicz, and J. H. Moore, "Evaluation of a tree-based pipeline optimization tool for automating data science", Proceedings of the genetic and evolutionary computation conference, Denver Colorado USA, pp. 485-492, Jul. 2016.
[https://doi.org/10.1145/2908812.2908918]

-
N. Erickson, J. Mueller, A. Shirkov, H. Zhang, P. Larroy, M. Li, and A. Smola, "Autogluon-tabular: Robust and accurate automl for structured data", arXiv:2003.06505, , Mar. 2020.
[https://doi.org/10.48550/arXiv.2003.06505]

-
T. Anwar, "COVID19 Diagnosis using AutoML from 3D CT scans", Proc. of the IEEE/CVF International Conference on Computer Vision, Montreal, BC, Canada, Oct 2021.
[https://doi.org/10.1109/ICCVW54120.2021.00061]

-
N. K. Tran, S. Albahra, T. N. Pham, J. H. Holmes, D. Greenhalgh, T. L. Palmieri, J. Wajda, and H. H. Rashidi, "Novel application of an automated-machine learning development tool for predicting burn sepsis: proof of concept", Scientific Reports, Vol. 10, Jul. 2020.
[https://doi.org/10.1038/s41598-020-69433-w]

-
K. W. Wan, C. H. Wong, H. F. Ip, D. Fan, P. L. Yuen, H. Y. Fong, and M. Ying, "Evaluation of the performance of traditional machine learning algorithms, convolutional neural network and AutoML Vision in ultrasound breast lesions classification: a comparative study", Quantitative imaging in medicine and surgery, Vol. 11, No. 4, pp. 1381-1393, Apr. 2021.
[https://doi.org/10.21037/qims-20-922]

-
M. Mirbabaie, S. Stieglitz, and N. R. J. Frick, "Artificial intelligence in disease diagnostics: A critical review and classification on the current state of research guiding future direction", Health and Technology, Vol. 11, No. 4, pp. 693-731, May 2021.
[https://doi.org/10.1007/s12553-021-00555-5]

-
A. H. Khan, H. Muzammil, and M. K. Malik, "Cardiac disorder classification by electrocardiogram sensing using deep neural network", Complexity, pp. 1-8, Mar. 2021.
[https://doi.org/10.1155/2021/5512243]

-
F. J. Díaz-Pernas, M. Martínez-Zarzuela, M. Antón-Rodríguez, and D. González-Ortega, "A deep learning approach for brain tumor classification and segmentation using a multiscale convolutional neural network", Healthcare, Vol. 9. No. 2. pp. 153, Feb. 2021.
[https://doi.org/10.3390/healthcare9020153]

-
A. A. Bataineh and S. Manacek, "MLP-PSO Hybrid Algorithm for Heart Disease Prediction", Journal of Personalized Medicine, Vol. 12, No. 8, Jul. 2022.
[https://doi.org/10.3390/jpm12081208]

-
T.-H. Lim, K.-O. Lim, S. Chung, and S.-C. Han, "Disease diagnosis research using deep learning based on military medical data", Journal of Digital Contents Society, Vol. 22, No. 9, pp. 1359-1367, Sep. 2021.
[https://doi.org/10.9728/dcs.2021.22.9.1359]

- Body signal of smoking, https://www.kaggle.com/datasets/kukuroo3/body-signal-of-smoking, [accessed: Sep. 14, 2023]
- Heart Disease Health Indicators Dataset, https://www.kaggle.com/datasets/alexteboul/heart-disease-health-indicators-dataset, [accessed: Sep. 14, 2023]

2019년 3월 ~ 현재 : 경상국립대학교 컴퓨터과학부 학사과정
관심분야 : 인공지능, 헬스케어, 멀티모달

2020년 3월 ~ 현재 : 경상국립대학교 컴퓨터과학부 학사과정
관심분야 : 인공지능, 자연어처리, 이미지 분류

2023년 3월 ~ 현재 : 경상국립대학교 AI 융합공학과 석사과정
관심분야 : 생성 모델, 인공지능

2006년 12월 : 호주뉴캐슬대학교 컴퓨터공학과(공학사)
2007년 9월 : 호주뉴캐슬대학교 정보공학과(공학석사)
2017년 8월 : 한양대학교 컴퓨터공학과(공학박사)
2021년 9월 ~ 현재 : 경상국립대학교 컴퓨터과학부 조교수
관심분야 : 인공지능, 시멘틱 헬스케어, 데이터마이닝