온톨로지 기반의 이기종 네트워크를 이용한 질병-유전자 상관관계 예측
초록
데이터를 의미론적으로 관리하고 분석하기 위한 온톨로지의 중요성이 최근에 크게 부각됨에 따라, 생명정보학 분야에서도 바이오 온톨로지의 활용이 급속히 증가하고 있다. 이 중, 유전자 온톨로지(GO)와 인간 표현형 온톨로지(HPO), 질병 온톨로지(DO)는 질병과 유전자 간의 상관관계를 예측하는 데에 활용될 수 있다. 본 논문에서는 온톨로지에 의미론적 유사성 측정 방법들을 적용하여, 가중치가 부여된 질병 네트워크와 유전자 네트워크를 구성하고, 이를 통합한 이기종 네트워크로부터 질병-유전자 상관관계를 예측하는 실험을 진행하였다. ROC 곡선과 AUC 값으로 실험 결과를 평가하였을 때, DO보다 HPO를 이용한 질병 네트워크에서 더 우수한 예측 정확도를 보였다. 또한, 기존에 주로 사용되는 MimMiner 또는 단백질 상호작용 데이터로 이기종 네트워크를 구성할 경우보다 온톨로지를 활용할 경우 더 우수한 예측 정확도를 보였다.
Abstract
As the importance of ontologies for semantically managing and analyzing data has recently been highlighted, the use of bio-ontologies is also rapidly increasing in Bioinformatics. For example, Gene Ontology(GO), Human Phenotype Ontology(HPO), and Disease Ontology(DO) can be used to predict associations between diseases and genes. In this paper, we applied semantic similarity measures to ontologies to construct weighted disease and gene networks, and performed experiments to predict disease-gene associations from heterogeneous networks incorporating them. When we evaluated the predictive performance using ROC curves and AUC values, the experimental results showed higher accuracy on the disease network constructed by HPO than DO. They also showed higher accuracy when using a heterogeneous network weighted by ontologies than that constructed by MimMiner or PPIs which are frequently used in previous studies.
Keywords:
ontology, semantic similarity, disease-gene association prediction, heterogeneous networks, disease networksⅠ. 서 론
최근 데이터를 의미론적으로 관리하고 분석하기 위한 목적으로 온톨로지의 활용이 확산되고 있다. 특히 빅데이터를 다루는 생명정보학 분야에서 각 도메인 별로 요소들의 속성 분석을 위한 온톨로지의 활용이 급속히 증가하고 있다[1]. BioPortal을 통하여 현재 1,000개 이상의 도메인에 해당하는 바이오 온톨로지가 구축되어 다양한 생명과학 또는 생명정보학 분야의 연구에 활용되고 있음을 확인할 수 있다[2]. 대부분의 바이오 온톨로지는 OBO(Open Biological and Biomedical Ontologies) 구조와 규정을 따르므로, 이 분야의 연구자들이 관련 데이터를 취득하고 분석하는 데에 편리성을 제공한다[3]. OBO 형태의 온톨로지 구조는 지정된 도메인 내의 개념들에게 의미론적인 부모-자식 관계를 부여한 구조로, 방향성을 가진 비순환 그래프(Directed acyclic graph) 형태로 표현될 수 있다. 즉, 그 그래프상에서 임의의 개념 Ci에 더 구체적인 의미를 포함하는 개념 Cj는 Ci의 자식 노드로 표현되고, 반대로 Ci는 Cj의 부모 노드로 표현된다. 하지만, Cj가 여러 부모 노드들을 가질 수 있는 측면에서 온톨로지 구조는 트리 구조와 구분되며, 트리의 상위 개념이라 할 수 있다.
현재 구축된 바이오 온톨로지 중에서 가장 널리 활용되는 예로 유전자 온톨로지(GO, Gene Ontology)를 들 수 있다[4]. GO는 모든 종에 걸쳐서 유전자 또는 그 외 생물학적 요소들의 속성에 대한 서술을 통합하여 온톨로지 형태로 구축한 빅데이터로 속성의 종류에 따라서 생물학적 과정(BP, Biological Process), 생체분자들의 기능(MF, Molecular Function), 세포 내의 요소(CC, Cellular Component)로 분류된다. 지난 20년간 GO 데이터는 지속적으로 추가되고 개선되어, 현재 BP 온톨로지에서는 대략 30,000개, 그리고 MF 온톨로지에서는 대략 12,000개의 속성을 포함하고 있다. GO가 다양한 연구에서 효과적으로 활용되는 주된 이유는 각 속성을 나타내는 유전자 또는 유전자 산물에 대한 정보를 주석(Annotation)으로 제공하기 때문이다. 이러한 주석 정보는 이미 출판된 논문에서의 실험 결과를 기반으로 구축되었고, 각 속성에 대한 유전자 정보 외에 그 정보가 어떤 실험을 통하여 획득되었는지를 가리키는 확증 코드(Evidence code)도 함께 제공된다.
질병 관련 연구를 위하여 널리 활용되는 온톨로지로 인간 표현형 온톨로지(HPO, Human Phenotype Ontology)[5] 와 질병 온톨로지(DO, Disease Ontology)[6]가 있다. HPO는 인간의 표현형을 온톨로지 형태로 구축한 빅데이터로 현재 15,000개 이상의 표현형을 포함한다. HPO의 장점은 GO와 유사하게 각 표현형에 대한 질병과 유전자 정보를 주석으로 제공하는 점이고, 이 주석 정보는 OMIM, OrphaNet, DECIPHER 데이터베이스로부터 지속적으로 추출하고 통합하여 구성되었다. DO는 인간에게 발병하는 질병들을 온톨로지 형태로 구축한 빅데이터로 약 10,000개의 질병 정보를 포함한다. DO는 특히 다른 질병 데이터베이스와 연결성을 외부 참조(External reference)로 제공하며, OMIM, SNOMED, MeSH, UMLS 등의 데이터베이스가 이를 지원한다.
이러한 유전자 및 질병 정보와 함께 네트워크를 기반으로 질병을 일으키는 유전자를 예측하는 연구가 활발히 진행되고 있다. 본 연구에서는 각 질병에 대해 그 질병과 관련된 유전자를 예측하는 데에 GO, HPO, DO 및 그 주석 정보를 활용하여 구축한 네트워크를 이용한다. 온톨로지 구조에서 속성 간의 의미론적 유사성(Semantic similarity)을 측정하고[7], 주석처리 된 유전자 사이의 유사성과 질병 사이의 유사성을 계산하여, 이를 통하여 가중치가 부여된 이기종 네트워크(Weighted heterogeneous network)를 구축한다. 동종 네트워크(Homogeneous network)는 모든 노드가 같은 속성만을 가진 그래프 구조를 의미하는 반면, 이기종 네트워크(Heterogeneous network)[8][9]는 다른 종류의 속성을 가진 노드가 혼재된 그래프 구조를 의미한다. 질병-유전자 네트워크에서 일부 노드는 질병을, 나머지는 유전자를 의미하므로 전형적인 이기종 네트워크라고 할 수 있다. 구축된 질병-유전자 네트워크에 그래프 이론 기반의 알고리즘을 적용하여 질병-유전자 상관관계(Disease-gene association)를 예측한다.
이러한 네트워크 기반의 질병-유전자 상관관계 예측 방법은 임의의 질병을 일으키는 후보 유전자 그룹을 짧은 시간 내에 효율적으로 찾아낸다는 장점이 있다.
다시 말해서, GO, HPO, DO의 정보를 활용하여 질병 관련 유전자 분석을 유전체 빅데이터 규모로 진행할 수 있다. 본 논문에서는 네트워크 기반의 질병-유전자 상관관계 예측을 위하여 온톨로지 내 의미론적 유사성 측정 방법들을 활용하여 구축된 유전체 규모의 유전자 및 질병 네트워크가 예측 성능에 미치는 영향을 분석한다.
본 논문의 구성은 다음과 같다. 2장은 관련 연구로 온톨로지 속성 간의 의미론적 유사성을 측정하는 방법들을 체계적으로 분류하고, 의미론적 유사성 값을 이용하여 주석처리 된 유전자 사이의 유사도 및 질병 사이의 유사도를 계산하는 방법을 소개한다. 3장에서는 의미론적 유사성 측정 방법들을 질병-유전자 상관관계 예측에 적용한 실험을 통하여 온톨로지를 활용하여 구성된 네트워크를 비교 평가한다. 또한, 이를 위한 실험 데이터, 실험 방법, 실험 결과를 기술한다. 마지막으로 4장에서는 본 연구의 중요성 및 향후 연구에 관해 기술한다.
Ⅱ. 관련 연구
2.1 온톨로지 속성 간 의미론적 유사성 측정
온톨로지 속성 간 의미론적 유사성을 측정하는 것은 온톨로지에 주석 처리된 정보 사이의 유사도를 측정하는 근간이 된다. 온톨로지 구조에서 각 속성은 노드로 표현되고 속성 간 부모-자식 관계는 엣지로 표현된다. 온톨로지 속성 간 의미론적 유사성을 측정하는 방법들을 정리하면, 온톨로지 엣지, 노드, 주석 정보 기반의 방법으로 분류될 수 있다.
엣지 기반의 유사성은 방향성을 가진 비순환 그래프 구조상에서 두 속성에 해당하는 노드 사이의 최단 경로 거리를 이용하여 측정된다. 그래프상에서 두 노드가 서로 가까울수록 그 속성은 서로 더 유사하다고 할 수 있다. 두 노드 사이의 최단 거리는 두 노드의 공통 조상 노드들 중에서 가장 근접한 조상 노드까지의 경로의 길이 합으로 계산된다. 또한, 가장 근접한 공통 조상 노드와 루트(Root) 사이의 거리 정보도 반영하여, 두 속성 Ci와 Cj 사이의 유사성을 식 (1)과 같이 정규화된 수치로 정량화할 수 있다.
| (1) |
이 식에서 Ck는 Ci와 Cj의 가장 근접한 공통 조상 노드이고, Cr은 온톨로지의 루트 노드를 의미하고, l(Ci, Ck)는 두 속성 Ci와 Ck 사이의 최단 경로 길이를 의미한다.
노드 기반의 유사성은 방향성을 가진 비순환 그래프 구조상에서 두 속성에 해당하는 노드의 공통 조상 노드들의 개수를 이용하여 측정한다. 공통 조상 노드들이 더 많을수록 그 속성은 서로 더 유사하다고 할 수 있다. 두 속성 Ci와 Cj의 유사성을 정규화된 수치로 정량화하기 위하여, 식 (2)와 같이 자카드 인덱스(Jaccard index)를 이용하여 두 속성의 조상의 합집합의 크기에 대한 두 속성의 조상의 교집합의 크기, 즉 공통 조상의 비율을 계산할 수 있다.
| (2) |
이 식에서 Sp(Ci)는 Ci의 조상 노드들의 집합을 의미하고, |Sp(Ci)|는 집합 Sp(Ci)의 크기, 즉 그 집합 내 구별되는 원소의 개수를 의미한다.
주석 정보 기반의 유사성은 온톨로지가 정확하고 충분한 양의 주석 정보를 포함하는 경우에 적용이 가능하다. 이 방법에서는 정보학 이론에서의 정보의 양(IC, Information Content)을 계산하는 아래의 공식을 활용하여 온톨로지 내 각 속성에 대한 의미론적 구체성(Semantic specificity)을 측정한다.
| (3) |
여기서 Ci는 의미론적 구체성을 측정하려는 속성이고, P(Ci)는 온톨로지에 주석 처리된 모든 유전자 중에서 Ci에 주석 처리된 유전자의 비율을 의미한다. 즉, 속성이 더 구체적일수록 그 속성에 대한 IC 값이 더 크다. 주석 정보 기반의 유사성은 두 속성에 해당하는 노드 Ci와 Cj의 공통 조상 노드 중에서 의미론적으로 가장 구체적인 노드 Ck의 정보의 양 IC(Ck)를 이용하여 측정한다. 또한, IC(Ci)와 IC(Cj)의 평균값을 이용하여, 두 속성 간의 유사성을 식 (4)와 같이 정규화된 수치로 정량화할 수 있다.
| (4) |
최근에 더 정확한 유사성 측정을 위하여, 이러한 주석 정보 기반의 방법을 온톨로지 구조, 즉 온톨로지 엣지 또는 노드를 기반으로 하는 방법과 통합한 새로운 방법을 개발하는 시도가 이루어져 왔다. 대표적인 예로 주석 정보 기반의 방법과 온톨로지 노드 기반의 방법을 통합하여, 식 (2)와 같이 두 속성 Ci와 Cj의 공통 조상 노드들의 개수만을 고려하지 않고, 식 (5)와 같이 공통 조상 노드들의 의미론적 구체성, 즉 공통 조상 노드들의 IC 값의 합을 이용하여 두 속성 간 유사성을 측정할 수 있다[10].
| (5) |
또한 최근 연구[11][12]에서는 온톨로지 속성 사이의 의미론적 유사성을 측정하기 위해 주석 정보 기반의 방법과 엣지 기반의 방법을 통합한 방법을 제시하거나, 주석 정보 기반의 방법과 그래프의 위상정보를 통합한 방법을 제시하였다. 이는 주석 정보를 기반으로 다양한 온톨로지 구조를 통합한 방법으로 의미론적 유사성을 측정하는 것이 최근의 연구 동향임을 알 수 있다.
2.2 주석 간 기능적 유사도 측정
온톨로지 주석 간 유사도를 측정하는 것은 그 주석이 달린 속성들의 집합 사이의 유사성으로 표현될 수 있다. 속성들의 집합 사이의 유사성을 측정하기 위하여 가장 널리 활용되는 방법은 두 집합 사이의 각 속성에 대해 가장 높은 유사성을 가지는 속성 쌍을 찾아 그들의 평균을 계산하는 방식(Best-match averaging)이다.
예를 들어, 유전자들이 온톨로지의 속성에 주석 처리되었을 경우, 두 유전자 g1과 g2 간의 기능적 유사도(Functional similarity)를 측정하기 위하여, 식 (6)과 같이 g1과 g2가 주석 처리된 각 속성에 대한 최적의 속성 쌍들의 유사성 평균값을 이용한다.
| (6) |
이 식에서, Ci는 유전자 g1이 주석 처리된 속성들의 집합이고, m은 그 속성들의 개수이며, Cj는 g2가 주석 처리된 속성들의 집합이고, n은 그 속성들의 개수를 의미한다.
또한, 식 (2)와 식 (5)에서는 두 집합 사이의 모든 속성 쌍들에 대한 의미론적 유사성을 고려하지 않고, 집합 연산을 확장하여 바로 두 집합 사이의 유사도를 측정하는 방식을 사용하기도 한다.
Ⅲ. 실험 방법 및 결과
본 장에서는 GO를 이용하여 구성한 유전자 네트워크와 HPO 또는 DO를 이용하여 구성한 질병 네트워크를 기반으로, 질병-유전자 이기종 네트워크를 구축하는 방법에 대해 설명한다. 또한, 각 네트워크의 밀도를 변경하면서 질병-유전자 상관관계 예측 실험을 진행하여 네트워크의 성능을 평가한다. 특히, HPO와 DO 중에서 어느 온톨로지를 통해 질병 네트워크를 구성하는 방법이 질병-유전자 상관관계 예측에 더 유리한지 평가한다.
3.1 실험 데이터
실험에 사용된 질병-유전자 상관관계 정보는 인간의 유전자 정보와 유전자 발현에 의한 장애 정보를 제공하는 OMIM(Online Mendelian Inheritance in Man)[13] 데이터베이스로부터 수집되었다. OMIM에서 추출한 질병-유전자 상관관계 정보는 질병 7,338개와 유전자 13,759개 사이에 25,477개의 상관관계를 포함한다. 이 중에서 질병은 HPO와 DO에 모두 주석 처리된 질병만을 남기고, 유전자는 GO의 BP 혹은 MF에 주석 처리된 유전자만을 남겨, 4,849개의 질병과 3,839개의 유전자 사이에 5,414개의 질병-유전자 상관관계를 기준으로 실험하였다.
유전자 네트워크를 구성하기 위하여 3.1.1장에서 언급한 3,839개의 유전자에 대해, GO의 BP와 MF에서 식 (5)의 방법을 이용하여 유전자 간 유사도를 계산하였다. 두 유전자 중 적어도 하나가 BP와 MF에 모두 주석 처리되지 않은 경우에는 그들의 유사도를 계산하지 않았고, BP와 MF 중 하나에는 주석 처리되지 않은 경우에는 주석 처리된 곳에서의 유사도 값을 사용하였으며, 두 유전자가 BP와 MF에 모두 주석 처리된 경우에는 BP와 MF에서 계산된 유사도 값 중 더 큰 값을 선택하였다.
본 실험에서는 두 개의 질병 네트워크를 구성하여 네트워크 간 성능을 비교하였다. 첫 번째 질병 네트워크는 HPO를 이용하여 구성하였고, 두 번째 질병 네트워크는 DO를 이용하였다. 질병 간의 유사도는 식 (5)를 이용하여 계산되었다. HPO로 구성된 질병 네트워크는 phenotypic abnormality의 하위 속성에 주석 처리된 질병 중 질병-유전자 상관관계 데이터에 존재하는 4,849개의 질병 간 모든 쌍에 대해 유사도를 계산하였다. DO로 구성된 질병 네트워크는 DO의 외부 참조(External reference) 정보 중 OMIM 정보를 주석으로 간주하여 계산하였으며, 질병-유전자 상관관계 정보에 포함되는 4,849개의 질병 간 모든 쌍에 대해 유사도를 계산하였다.
3.2 실험 방법
이 실험에서는 질병-유전자 이기종 네트워크에 랜덤워크[14] 기술을 적용하여 질병-유전자 상관관계를 예측하였다. 예측 정확도를 평가하기 위하여, 리브원아웃 교차검증(Leave-one-out cross-validation) 방법을 이용하였다. 리브원아웃 교차검증에서는 각 질병과 유전자 간의 상관관계를 이기종 네트워크로부터 삭제한 후 이를 예측하는 실험을 4,849개의 모든 질병에 대해 교차검증으로 진행하였다. 예측 점수가 임계값(Threshold) 이상인 유전자를 양성 데이터(Positives)로, 반대로 임계값 미만인 경우를 음성 데이터(Negatives)로 간주하였다. 이러한 실험 조건에서 임계값을 최대 예측 점수에서부터 최소 예측 점수로 감소시키면서 참의 양성율(TPR, True Positive Rate)과 거짓 양성율(FPR, False Positive Rate)의 변화를 추적하여 거짓 양성율이 증가함에 따른 참의 양성율의 변화를 보여주는 ROC(Receiver Operating Characteristic) 곡선과 그 곡선을 이용하여 예측 정확도를 정량화한 AUC(Area Under the Curve) 값을 비교하였다. 특정 네트워크에서 높은 예측 정확도를 보인다는 것은 그 네트워크의 가중치가 질병-유전자 상관관계 예측에 유리하게 작용하였다고 판단할 수 있다.
3.3 실험 결과
3.1장에서 구성한 질병 네트워크와 유전자 네트워크는 기본적으로 모든 노드 쌍에 가중치가 부여된 완전 연결 그래프로 구성되지만, 유사도가 높은 노드 쌍에만 엣지로 연결하기 위하여 질병 네트워크와 유전자 네트워크의 밀도(Density)를 각각 1%에서 10%까지 조절하면서 실험을 진행하였다. 여기서 네트워크 밀도는 그래프의 모든 노드 쌍에서 엣지의 비율을 의미한다.
표 1과 2는 HPO-기반 이기종 네트워크와 DO-기반 이기종 네트워크에서 질병 네트워크와 유전자 네트워크의 밀도가 각각 1%, 5%, 10%일 때 질병-유전자 상관관계 예측 실험에 대한 AUC 값을 보여준다. 표 1과 2를 비교하였을 때, DO-기반 네트워크에서 보다, HPO-기반 네트워크를 이용한 실험에서 더 높은 AUC 값을 보였다.
또한, 두 이기종 네트워크에서 유전자 네트워크의 밀도가 1%일 때 예측 정확도가 현저하게 낮아지는 결과를 확인할 수 있었고, 유전자 네트워크의 밀도가 증가함에 따라 예측 정확도는 대체적으로 증가하였다. 반면에, 질병 네트워크의 밀도에 대해서는 표 1의 HPO-기반 네트워크를 이용한 실험에서 질병 네트워크의 밀도가 증가함에 따라 예측 정확도는 약간씩 감소하였고, 표 2의 DO-기반 네트워크를 이용한 실험에서는 질병 네트워크의 밀도가 1%일 때 낮은 예측 정확도를 확인할 수 있었다. 네트워크의 밀도를 1%에서 10%까지 변화시킴에 따라서 AUC 값의 변동 폭을 분석하면, 질병 네트워크보다 유전자 네트워크의 밀도를 변화시킴에 따른 AUC 값의 변동 폭이 더 컸다. 이 결과로부터 높은 질병-유전자 상관관계 예측을 위하여 유전자 네트워크의 밀도를 더 세밀히 조절하는 것이 중요함을 알 수 있다.
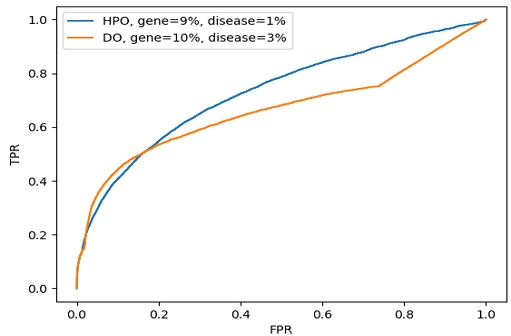
최종적으로 HPO-기반 네트워크를 이용한 실험에서는 유전자 네트워크의 밀도가 9%, 질병 네트워크의 밀도가 1%일 때 가장 높은 예측 정확도를 보였고, DO-기반 네트워크를 이용한 실험에서는 유전자 네트워크의 밀도가 10%, 질병 네트워크의 밀도가 3%일 때 가장 높은 예측 정확도를 보였다. 해당 조합에서의 ROC 곡선은 그림 1과 같다. HPO-기반 네트워크에서의 AUC 값은 0.731, DO-기반 네트워크에서의 AUC 값은 0.666으로, 표 1과 2에서도 이미 확인했던 것처럼, DO를 사용하는 것보다 HPO를 사용하여 질병-유전자 이기종 네트워크를 구성할 때 질병-유전자 상관관계 예측 정확도가 더 높았다.
질병-유전자 상관관계 예측을 위한 질병 네트워크를 구성할 때, 기존 연구에서는 MimMiner[15]를 활용하여 질병 간 유사도를 구하는 방법이 가장 널리 사용된다. MimMiner는 OMIM에서 제공하는 질병정보에 대해 텍스트 마이닝 기법을 이용하여 질병 간의 유사도를 구한다. HPO를 통해 구성한 질병 간 유사도와 MimMiner로 구성한 질병 간 유사도를 평균 또는 최댓값으로 통합하여 HPO-통합 질병 네트워크를 구성한 다음, 질병-유전자 상관관계 예측 실험을 진행하였다.
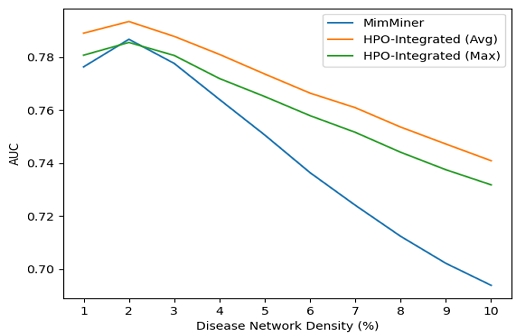
그림 2는 MimMiner로 구성한 질병 네트워크와 HPO-통합 질병 네트워크에서 밀도를 조절하며 질병-유전자 상관관계를 예측했을 때 AUC 값의 변화를 보여준다. MimMiner 만을 사용했을 때보다 HPO-통합 네트워크를 사용했을 때, 전반적으로 더 높은 AUC 값을 확인할 수 있으며 질병 네트워크의 밀도가 변함에 따라 AUC 값의 변동 폭이 작은 것을 확인할 수 있다. 이 결과를 통하여, MimMiner 만을 사용하는 것보다 HPO-통합 질병 네트워크를 사용하는 것이 질병-유전자 상관관계 예측에 더 유리함을 알 수 있다.
기존의 질병-유전자 상관관계 예측에서는 단백질 상호작용(PPI, Protein-Protein Interactions) 데이터가 유전자 네트워크로 주로 사용된다. 두 개의 단백질에 대해 상호작용이 있을 경우에는 그 단백질에 해당하는 유전자가 기능적으로 서로 유사하다는 것을 암시한다. 본 실험에서는 GO를 통해 구성한 유전자 네트워크를 사용할 때와 PPI를 유전자 네트워크로 사용할 때의 질병-유전자 상관관계 예측 실험을 진행하였다. 이 실험을 위한 유전체 수준의 PPI 데이터는 BioGRID[16] 데이터베이스로부터 추출하였다.
표 3은 GO를 활용하여 구성한 유전자 네트워크와 PPI를 사용한 유전자 네트워크로 각각 질병-유전자 상관관계를 예측했을 때 AUC 값을 보여준다. 질병 네트워크를 MimMiner로 구성했을 때 GO와 PPI에 따른 AUC 값의 차이는 거의 없지만, HPO-통합 네트워크에서는 GO를 통해 구성한 유전자 네트워크를 사용하는 것이 PPI를 사용할 경우보다 상관관계 예측에 근소하게 더 유리함을 알 수 있다.
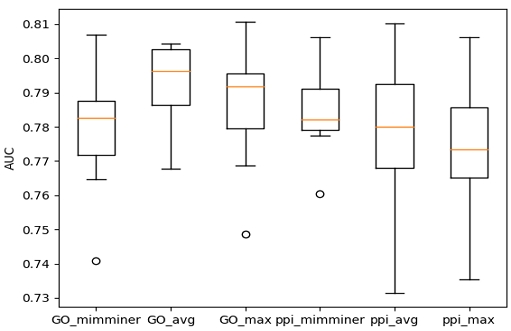
또한, 질병-유전자 상관관계 예측에 대한 오차율을 분석하기 위하여, 표 3에서의 6가지 실험 조건에 대해 10배 교차검증(10-fold cross-validation) 방법을 이용하여 각 폴드 별 AUC 값의 분포를 조사하였다. 그림 3은 각 조건에 대한 AUC 값의 분포도를 상자그림으로 보여준다. 표 3에서의 결과와 유사하게 GO를 유전자 네트워크로 사용하고 HPO-통합 네트워크를 질병 네트워크로 사용하였을 때 가장 우수한 예측 정확도를 보이는 것을 재확인할 수 있다. 반면에 PPI를 유전자 네트워크로 사용하고 HPO-통합 네트워크를 질병 네트워크로 사용하였을 때에는 폴드 별 AUC 값의 편차가 상당히 큰 것을 확인할 수 있다. 따라서 이 조건에서는 훈련 데이터에 따른 예측 오차율이 높다는 결과를 추론할 수 있다.
Ⅳ. 결 론
최근 다양한 바이오 온톨로지의 출현에 따라, 이를 활용하기 위하여 온톨로지의 개념이나 속성에 해당하는 데이터를 의미론적으로 분석하기 위한 다양한 방법이 제안되어왔다. 본 연구에서는 온톨로지의 개념을 바탕으로 온톨로지를 통해 의미론적 유사도를 구하는 방법을 체계적으로 분류하고, 바이오 온톨로지 중에서 GO, HPO, DO를 활용하여 질병-유전자 상관관계 예측 실험을 유전체 단위에서 진행하였다. GO와 HPO, GO와 DO를 사용하여 두 개의 질병-유전자 이기종 네트워크를 구성하였고, 각 네트워크에 랜덤워크 기법을 적용하여 질병-유전자 상관관계를 예측하였다.
실험 결과, DO를 사용하여 질병 네트워크를 구성하는 경우보다 HPO를 사용할 경우 더 높은 예측 정확도를 보였다. 또한, 질병-유전자 이기종 네트워크 구성을 위하여 적절한 유전자 네트워크의 밀도를 선택하는 것이 중요하였다. 또한, 기존의 질병-유전자 상관관계 예측에 주로 사용되는 MimMiner와 PPI를 적용한 경우보다 온톨로지를 통해 네트워크를 구성하는 방법이 질병-유전자 상관관계 예측에 조금 더 유리하게 작용하는 결과를 보였다. 특히, GO를 유전자 네트워크로 사용하고 HPO와 MimMiner를 통합한 방법으로 질병 네트워크를 구축한 이기종 네트워크에서 질병-유전자 상관관계를 예측할 경우에 정확도가 가장 높았으며 입력되는 훈련 데이터에 따른 예측 정확도의 차이, 즉 오차율이 비교적 작았다.
본 연구를 통하여 네트워크의 밀도를 조절하는 것이 질병-유전자 상관관계 예측 실험에 영향을 미칠 수 있다는 점과 온톨로지를 사용하여 질병 네트워크 및 유전자 네트워크를 구성하는 것이 질병-유전자 상관관계 예측 실험에 유리함을 입증한 점에서 그 의의를 찾을 수 있다. 향후 과제로, 80% 수준의 질병-유전자 상관관계 예측 정확도를 더욱 향상시키기 위하여 더욱 정밀한 이기종 네트워크의 구성 및 더욱 정확한 상관관계 예측 방법의 제시가 필요하다.
Acknowledgments
이 논문은 2022년도 과학기술정보통신부의 재원으로 한국연구재단 기초연구사업의 지원(2021R1A2C101194612)과 교육부의 재원으로 한국연구재단 지자체-대학 협력기반 지역혁신사업의 지원(2022RIS-005)을 받아 수행된 연구임
References
-
O. Bodenreider and R. Stevens, "Bio-ontologies: current trends and future directions", Briefings in Bioinformatics, Vol. 7, No. 3, pp. 256-274, Sep. 2006.
[https://doi.org/10.1093/bib/bbl027]

-
P. L. Whetzel, N. F. Noy, N. H. Shah, P. R. Alexander, C. Nyulas, T. Tudorache, and M. A. Musen, "BioPortal: enhanced functionality via new Web services from the National Center for Biomedical Ontology to access and use ontologies in software applications", Nucleic Acids Research, Vol. 39, pp. W541-W545, Jul. 2011.
[https://doi.org/10.1093/nar/gkr469]

-
J. B. L. Bard and S. Y. Rhee, "Ontologies in Biology: design, applications and future challenges", Nature Reviews: Genetics, Vol. 5, pp. 213-222, Mar. 2004.
[https://doi.org/10.1038/nrg1295]

-
The Gene Ontology Consortium, "The Gene Ontology resource: enriching a GOld mine", Nucleic Acids Research, Vol. 49, No. D1, pp. D325–D334, Dec. 2021.
[https://doi.org/10.1093/nar/gkaa1113]

-
S. Köhler, et al., "The Human Phenotype Ontology in 2021", Nucleic Acids Research, Vol. 49, No. D1, pp. D1207–D1217, Jan. 2021.
[https://doi.org/10.1093/nar/gkaa1043]

-
L. M. Schriml, et al., "The Human Disease Ontology 2022 update", Nucleic Acids Research, Vol. 50, No. D1, pp. D1255–D1261, Jan. 2022.
[https://doi.org/10.1093/nar/gkab1063]

-
C. Pesquita, D. Faria, A. O. Falcao, P. Lord, and F. M. Couto, "Semantic similarity in Biomedical Ontologies", PLoS Computational Biology, Vol. 5, No. 7, Jul. 2009.
[https://doi.org/10.1371/journal.pcbi.1000443]

-
S. K. Ata, M. Wu, Y. Fang, L. O. Yang, C. K. Kwoh, and X. L. Li, "Recent advances in network-based methods for disease gene prediction", Briefings in Bioinformatics, Vol. 22, No. 4, Jul. 2021.
[https://doi.org/10.1093/bib/bbaa303]

-
H. Yu and Y. Yoon, "Drug repositioning through drug-disease bipartite network", Journal of KIIT, Vol. 18, No. 12, pp 1-9, Dec. 2020.
[https://doi.org/10.14801/jkiit.2020.18.12.1]

-
C. Pesquita, D. Faria, H. Bastos, A. E. N. Ferreira, A. O. Falcao, and F. M. Couto, "Metrics for GO based protein semantic similarity: a systematic evaluation", BMC Bioinformatics, Vol. 9, pp. 54, Apr. 2008.
[https://doi.org/10.1186/1471-2105-9-S5-S4]

-
C. Zhao and Z. Wang, "GOGO: An improved algorithm to measure the semantic similarity between gene ontology terms", Scientific Reports, Vol. 8, Oct. 2018.
[https://doi.org/10.1038/s41598-018-33219-y]

-
P. Dutta, S. Basu, and M. Kundu, "Assessment of Semantic Similarity between Proteins Using Information Content and Topological Properties of the Gene Ontology Graph", IEEE/ACM Transactions on Computational Biology and Bioinformatics, Vol. 15, No. 3, pp. 839-849, May 2018.
[https://doi.org/10.1109/TCBB.2017.2689762]

-
J. S. Amberger, C. A. Bocchini, A. F. Scott, and A. Hamosh, "OMIM.org: Leveraging knowledge across phenotype–gene relationships", Nucleic Acids Research, Vol. 47, No. D1, pp. D1038-D1043, Jan. 2019.
[https://doi.org/10.1093/nar/gky1151]

-
Y. Li and J. C. Patra, "Genome-wide inferring gene–phenotype relationship by walking on the heterogeneous network", Bioinformatics, Vol. 26, No. 9, pp. 1219–1224, May 2010.
[https://doi.org/10.1093/bioinformatics/btq108]

-
M. A. Driel, J. Bruggeman, G. Vriend, H. G. Brunner, and J. A. M. Leunissen, "A text-mining analysis of the human phenome", European Journal of Human Genetics, Vol. 14, pp. 535–542, Feb. 2006.
[https://doi.org/10.1038/sj.ejhg.5201585]

-
R. Oughtred, et al., "The BioGRID database: A comprehensive biomedical resource of curated protein, genetic, and chemical interactions", Protein Science, Vol. 30, pp. 187–200, Jan. 2021.
[https://doi.org/10.1002/pro.3978]


2022년 8월 : 연세대학교 미래캠퍼스 컴퓨터공학과(공학사)
2022년 9월 ~ 현재 : 연세대학교 미래캠퍼스 전산학과 통합과정
관심분야 : 데이터마이닝, 바이오인포매틱스, 빅데이터, 인공지능

1994년 2월 : 연세대학교 공과대학(공학사)
2003년 12월 : 일리노이주립대학 컴퓨터공학 졸업(공학석사)
2009년 6월 : 뉴욕주립대학 컴퓨터공학 졸업(공학박사)
2009년 8월 ~ 2019년 12월 : 베일러대학교 컴퓨터공학과 부교수
2020년 3월 ~ 현재 : 연세대학교 미래캠퍼스 소프트웨어학부 부교수
관심분야 : 바이오인포매틱스, 데이터마이닝, 빅데이터, 인공지능