코로나19 환자 조기 분류를 위한 다층 스태킹 앙상블 기반 임상 중증도 예측 모델
초록
코로나19 확산 상황에서는 환자의 임상 중증도에 따라 신속하게 치료순위를 정하고 입원 치료가 필요한 환자들을 빠르게 선별하는 것이 중요하다. 본 논문에서는 5,651명의 코로나19 확진자 임상역학정보 데이터를 기반으로 다양한 기계학습 모델의 특성을 활용할 수 있는 다층 스태킹 앙상블 기반 임상 중증도 예측 모델을 제안한다. 모델의 정확도를 확보하기 위해 다양한 기계학습 기반 모델들 간의 상관분석을 실시하여 제안모델에 포함되는 서브모델들을 도출하였다. 또한 실제 코로나19 임상 중증도에 영향을 미치는 특성들을 분석하여 선별된 특성 적용을 통해 모델의 성능을 향상시켰다. 실험 결과 본 논문에서 제안한 모델의 F1(0.9373) 및 AUROC(0.9545) 점수는 기존 단일 모델들의 평균 점수 대비 각각 7.25%, 6.21% 향상되었다.
Abstract
In the context of the COVID-19 pandemic, it is crucial to prioritize treatment sequences based on the clinical severity of patients and select patients requiring inpatient treatment. This paper proposes a multi-layer stacking ensemble-based clinical severity prediction model that can utilize the characteristics of various machine learning models based on the clinical epidemiologic data of the 5,628 confirmed COVID-19 cases. To ensure the accuracy of the model, the sub-models included in the proposed model were derived through correlation coefficient analysis between various machine learning-based models. Also, The performance of the model was improved by applying the selected features by analyzing the features affecting the actual COVID-19 clinical severity. The experimental results improved the F1-Score(0.9373) and AUROC-Score(0.9545) of the proposed model by up to 7.25% and 6.21% compared to the existing single models, respectively.
Keywords:
COVID-19, early triage, clinical servility prediction, stacking ensembleⅠ. 서 론
2019년 12월 코로나19가 처음 출현한 이후 전 세계적으로 사망자가 급증하였으며, 유례없는 높은 전염성으로 현재까지 완전히 종식되지 않고 신규확진자가 계속해서 발생하고 있다. 코로나19와 같은 신종 감염병 상황에서는 확진자의 급속한 확산과 사망 위험성으로 인해 의료시설과 의료진들이 심각히 부족해지며, 이에 따라 적절한 치료를 받지 못해 증상이 악화하여 사망에 이르는 환자들이 다수 발생할 수 있다[1]. 따라서, 제한된 의료진과 시설을 통해 의료시스템 전달체계를 효율적으로 제공하기 위해서는 코로나19 확진자의 임상 중증도 예측하여 신속하게 치료순위를 정하고 입원 치료가 필요한 환자들을 빠르게 선별하는 것이 중요하다[2].
현재 임상 중증도 분류는 질병관리청 중앙방역대책본부에서 규정한 경증, 중등증, 중증, 최중증으로 이루어져 있으며 수축기혈압, 맥박, 호흡수, 체온, 의식 수준 등 5가지의 지표를 기준으로 분류된다[3]. 경증의 경우 저위험군 환자로서 무증상 또는 경증 호흡기 증상을 가진 경우이며, 중등증의 경우 발현되는 경증 호흡기 증상을 안정화하는 대증치료 또는 증상 경과 모니터링 등을 수행하는 단계이다. 중등증의 경우부터는 고위험군의 환자로서 호흡 불안으로 인공호흡기를 통한 기계호흡 치료가 필요한 단계이며 마지막으로 최중증의 경우 호흡부전, 패혈증성 쇼크 등으로 인해 에크모와 같이 환자의 혈액에 산소를 공급하기 위해 인공 폐 및 혈액 펌프 등기구를 활용하는 단계로 정의된다.
우리나라의 경우 코로나19를 비교적 잘 통제하였다고 알려졌지만, 중증 또는 최중증 환자 분류의 경우 X-Ray, Chest CT 등의 주요 검사장비를 갖춘 병원에서 검진 결과를 통해 임상 중증도를 분류하기 때문에 이러한 고위험군의 환자들을 관리하는 데 어려움을 겪었다. 실제 이러한 이유로 자택에서 대기하거나 생활치료센터에 격리되어 있던 환자가 대응 지연으로 인해 임상 상태 악화로 사망한 사례가 있다[4]. 따라서 환자의 인터뷰나 간단한 검사를 통해 얻은 특징을 기반으로 환자의 상태를 신속하고 정확하게 식별하는 임상 중증도 예측 모델이 필요하다.
본 논문에서는 PCR 검사를 통해 양성판정을 받은 코로나19 확진 환자들을 대상으로 의료진의 신속한 진단에 도움을 줄 수 있는 코로나19 환자 조기 분류를 위한 임상 중증도 예측 모델을 제안한다. 이를 위해 제안모델은 많은 인력과 주요 의료 장비를 필요로 하는 기존 임상 중증도 판별 방법과 달리 환자의 문진 데이터 또는 건강 애플리케이션을 통해 확보할 수 있는 키, 몸무게, 체온, 심박수, 혈압 및 기저질환과 같은 데이터를 활용한다. 또한 실제 임상 환경의 경우 환경적 요인으로 인하여 데이터 획득에 제한이 발생할 수 있으므로, 데이터 사용 전략 및 재귀적 특징제거 방법(RFE, Recursive Feature Elimination)을 통해 선별된 특징만을 활용하여 임상 중증도 예측 모델의 정확도를 향상시켰다. 마지막으로 대부분의 기계학습 기반의 관련 연구들과 같이 특정한 단일 모델만을 기반으로 예측하였을 때는 모델 자체에 존재하고 있는 편향이 예측 결과에 반영될 위험이 있으므로 다양한 기계학습 모델들이 각각 독립적이라고 가정하여 임의의 조합을 통해 오분류율을 최소화하는 다층 스태킹 앙상블 기반의 학습 방법을 제안한다.
본 논문의 구성은 다음과 같다. 2장은 관련연구로 코로나19 임상 중증도 예측 관련 연구들을 살펴보며, 3장에서는 본 논문에서 제안하는 다층 스태킹 앙상블 기반 임상 중증도 예측 모델에 대해 자세히 설명한다. 4장에서는 다양한 실험을 통해 본 논문에서 제안하는 방법론의 성능을 분석하고 제안하는 모델의 우수성을 증명한다. 마지막으로 5장에서는 결론에 관해 기술하고 본 연구가 갖는 한계점 및 향후 연구에 관하여 기술한다.
Ⅱ. 관련 연구
현재 코로나19의 세계적 확산과 대응 시급성으로 인해 임상 중증도 예측을 위한 많은 연구들이 제시되고 있다. 대부분의 관련 연구들은 코로나19 양성판정을 받은 환자들을 대상으로 하며 효과적으로 환자들의 예후 예측을 위해 기계학습 모델을 활용하여 중증도 예측을 수행하였다.
Yan, L et al.,(2020)[5]와 Shang, W et al.,(2020)[6]은 코로나19 발생 초기 중국 우한의 한 지역병원에서 획득한 데이터를 활용하여 코로나19 환자의 임상 중증도 예측을 수행하였다. 두 연구 모두 환자의 임상 특성 및 입원 중 기록 정보(e.g. 인적정보, 혈액검사결과, 병력 등)을 기반으로 코로나19의 중증도에 주요하게 영향을 줄 수 있는 임상 변수들이 무엇인지 검출하고 이를 활용하여 중증도 예측의 정확도를 높이고자 하였다. 하지만 사용된 환자의 데이터 수가 각각 375명, 443명으로 제한된 수의 데이터만을 기반으로 제안되었기 때문에 해당 방법을 일반화하여 적용하는 데에는 문제가 있다.
Liang W et al.,(2020)[7]은 예측 모델의 일반화 가능성을 높이기 위해 중국의 우한, 후베이, 광둥 지역의 병원들에서 획득한 1,590명의 데이터를 활용하여 임상 중증도 예측을 수행하였다. 해당 연구에서는 코로나19 환자의 생존율을 예측할 수 있는 딥러닝 기반의 Cox Proportional Hazard Model을 제안하였지만, 각 환자당 사건(Event)이 발생한 시간과 관련된 시계열 기반의 추적 코호트 데이터가 학습에 필요하므로 지속적인 데이터 확보가 매우 어렵고 데이터 확보 비용 또한 많이 드는 단점이 발생한다.
Jin C et al.,(2020)[8], Xu Q et al.,(2021) [9], Al Rahhal MM et al.,(2021)[10]은 흉부 X-Ray, Chest CT 이미지를 활용하여 코로나19 환자의 사망률 및 시간에 따른 환자의 중증도 변화를 예측하였다. 특히 컴퓨터 비전 분야에서 높은 성능을 보이는 Vision Transformer(ViT)의 기법을 활용하여 학습 이미지와 증강 이미지를 분할 후 처리하거나 텍스트 기반 환자의 임상 정보 데이터를 추가로 모델에 반영하여 정확도를 높이는 결과를 제시하였다. 해당 연구들은 시간에 따른 중증도 변화를 시각적인 정보로 분류할 수 있다는 데 의의가 있지만, 학습에 활용 이미지 데이터들의 경우 주요 검사장비를 통해 획득할 수 있으므로 신속한 초기 분류가 요구되는 상황에서는 채택하기 어렵다.
An C et al.,(2020)[11], Zoabi Y et al.,(2021)[12], Kim J et al.,(2021)[13]은 비교적 쉽게 확보할 수 있는 환자의 문진 데이터, 기저질환, 임상 소견 데이터를 기반으로 Logistic Regression, Gradient Boosting Machine, XGBoost 등 특정한 기계학습 방법을 활용하여 임상 중증도 예측을 수행하였다. 해당 연구들에서는 각 모델의 예측과정에서 중증도 예측의 주요한 임상 변수들을 확인하고 최적의 하이퍼파라미터 튜닝을 통해 예측이 가능한 최적의 단일 모델을 제시하였다. 하지만 기계학습에서 특정한 예측 모델만을 기반할 경우 편향(Bias)-분산(Variance) trade off로 인하여 모델 자체적으로 존재하고 있는 편향이 예측에 반영될 위험이 존재한다. 또한 An C et al.,(2020)[11]의 모델 비교실험과정에서 제시된 바와 같이 중증도 예측의 주요한 임상 변수들이 모델마다 상이한 결과를 제시하기도 한다, 예를 들어 LASSO의 경우 중요 임상 변수로 당뇨병 또는 암을 포함했지만, RandomForest의 경우 감염 경로(집단 감염 또는 개인 접촉으로 인한 감염), 기저 고혈압이 포함되었다. 이러한 이유로 해당 연구들에서는 경증, 중등중, 중증, 최중증과 같이 세분화된 임상 중증도 예측이 아니라 생존, 사망과 관련된 단순화된 이진 분류 형태로 중증도 예측을 진행하였다.
본 논문에서는 일반화된 임상 중증도 예측 모델 제시를 위해 질병관리청 및 고려대학교 안산병원을 통해 획득한 5,651명의 코로나19 환자 데이터를 기반으로 중증도 예측을 수행한다. 또한 신속한 중증도 분류를 위해 비교적 쉽게 획득될 수 있는 환자의 기본 인적정보, 활력징후, 임상 증상, 기저질환 등의 데이터만을 활용하며, 중증도 예측의 주요한 임상 변수 파악과 모델의 영향성을 복수의 기계학습 기반 모델들로 파악한 후 각 모델을 다층 스태킹 앙상블 학습 모형으로 결합하여 모델 편향 위험을 경감시키는 새로운 중증도 예측 방법을 제시한다.
Ⅲ. 다층 스태킹 앙상블 기반 임상 중증도 예측 모델
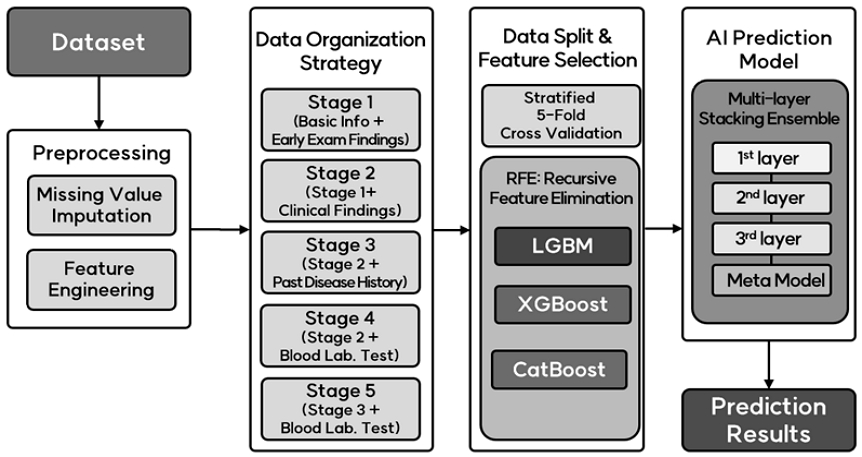
본 논문에서 제안하는 다층 스태킹 앙상블 모델 기반 임상 중증도 예측 모델 구축 절차는 그림 1과 같다. 먼저 획득한 코로나19 환자 데이터를 활용하여 데이터 전처리 기법을 시행하고, 최소의 임상 변수만을 사용하여 예측을 최적화할 수 있도록 환자의 임상 특성을 고려한 5가지 형태의 데이터 구성 전략을 적용하여 데이터를 구성한다. 이후 데이터 분할 및 특징 선택 과정을 통해 예측에 영향력이 있는 특징만 선별하고 마지막으로 제안모델을 통해 예측을 수행한다.
3.1 데이터셋
본 논문에서는 질병관리청 중앙방역대책본부를 통해 2020년 4월 30일까지 우리나라 전역 100개 이상의 병원에서 획득한 5,628명의 코로나19 임상역학정보 데이터와 2020년 8월 30일까지 고려대학교 안산병원에서 획득한 50명의 환자데이터를 결합한 총 5,678명의 코로나19 환자데이터를 활용하였다. 본 데이터들은 PCR 검사 후 양성판정을 받은 환자를 대상으로 총 42개의 임상 변수로 구성되어 있다. 본 논문에서는 환자 아이디, 격리해제상태, 격리기간 등 예측과정에서 활용성이 없는 데이터들은 제외하여 표 1과 같이 총 38개의 임상 변수만 활용하였다. 또한 5,678명의 데이터 중 27명의 환자의 경우 예측에서 목표변수로 활용되는 임상중증도점수(CSS, Clinical Severity Score)가 누락되어 본 연구에서는 총 5,651명의 환자데이터만 활용하였다.
3.2 데이터 전처리
데이터 전처리 단계에서는 먼저 일부 임상 변수들의 결측값들을 처리하는 Missing Value Imputation을 실시한다. 데이터셋 내부 결측값 중 Yes, No와 같은 범주형 변수인 경우 빈도(Frequency)를 기반으로 결측값들을 대체하였으며, 수치형 변수의 경우 중앙값(Median)을 기반으로 대체하였다. 마지막으로 임신과 관련된 임상변수인 임신여부(PREG) 및 임신주수(PREGW)의 경우 임신인 경우를 제외한 나머지 값들을 모두 0으로 처리하였다.
Feature Engineering의 경우 초기 검사 정보 중 심박수(HRI) 및 체온(TEMPI) 변수에 대해 연령별 정상 심박수 범위와 정상체온 구간 범위를 참고하여 수치형 변수를 이산형 변수로 변경하였다[14][15]. 또한 기계학습 모델에서 일반적인 요구사항인 표준화(Standardization)를 위해 목표변수인 임상중증도점수(CSS)를 제외한 학습에 사용되는 변수들은 모두 평균이 0이고 표준편차가 1인 가우시안 정규분포를 가진 형태로 각 변수들의 분포를 변경하였다.
| (1) |
마지막으로 목표변수인 임상중증도점수(CSS)의 경우 표 2와 같이 총 8개의 클래스로 분류되었지만 5,651명의 환자 데이터 중 4,456명(78.9%)이 1번 클래스인 ‘일상생활 지장 없음’이었으며 1,195명(21.1%) 환자 데이터만이 2번부터 8번 클래스로 구성되어 있다. 또한 5번 클래스인 ‘비침습인공호흡기’와 6번 클래스인 ‘침습인공호흡기’의 경우 각각 21명(0.4%) 및 18명(0.3%)의 환자 데이터만 존재하므로 모델 학습에 활용하기에는 매우 부족하다.
이러한 문제를 해결하기 위해 1번과 2번 클래스는 ‘경증’(4,791명, 84.6%), 3번부터 6번 클래스는 ‘중증’(601명, 10.6%), 7번 및 8번 클래스는 ‘최중증’(259명, 4.6%)으로 목표변수를 재구성하였다. 클래스 재구성 시 발생되는 불균형 문제는 모델 학습 시 활용되는 하이퍼파라미터인 class weight를 통해 balanced로 설정하여 한쪽 클래스로 편향되지 않게 학습이 진행되도록 하였다.
3.3 데이터 구성 전략
실제 임상 환경의 경우 제한된 의료진이나 시설 사용 현황으로 인해 신속하게 환자의 모든 임상 변수 정보를 확보하기 어려울 수 있다. 데이터 구성 전략 단계에서는 환자가 병원에 방문했을 때 확보하기 쉬운 임상 변수들을 우선으로 학습데이터를 구성하여 예측 정확도를 확인해보기 위해 총 5개의 단계로 학습데이터를 구성하였다.
Stage 1의 경우, 연령(AGE), 성별(SEX), 체질량지수(BMI) 등으로 구성된 환자의 기본 정보와 수축기혈압(SBP), 심박수(HRI), 체온(TEMPI) 등 환자가 병원에 방문하였을 때 빠르게 확보할 수 있는 초기 검사 정보와 관련된 총 9개의 임상 변수로 구성된다. Stage 2의 경우, Stage 1 데이터에서 발열(FEVER), 기침(COUGH)와 같은 임상 소견 정보가 추가되어 총 21개의 임상변수로 구성된다. Stage 3에서는 Stage 2 데이터에서 당뇨(DM), 고혈압(HTN) 등 환자의 현재/과거 병력과 관련된 기저질환을 추가하여 총 32개의 임상 변수로 구성된다. Stage 4에서는 환자의 현재/과거 병력의 조회가 어려울 경우를 대비하여 Stage 2 데이터에 헤모글로빈(HGB), 림프구(LYMPHO)등의 혈액검사 수치 정보를 추가한 총 26개의 임상 변수로 구성되며, 마지막으로 Stage 5에서는 환자의 현재/과거 병력과 관련된 기저질환이 포함된 Stage 3 데이터를 기준으로 혈액검사 정보를 추가하여 총 37개의 임상 변수로 구성된다.
3.4 데이터 분할 및 특징 선택
특징 중요도에 따른 특징 선택 과정 및 중증도 예측 모델 개발을 수행하기 위해 먼저 5,651명의 환자데이터를 층화추출방법(Stratified Sampling)을 활용하여 학습데이터셋(3,956/5,651, 70%)과 테스트 데이터셋(1,695/5,651, 30%)으로 분할 하였다. 학습데이터의 경우 모델 학습 시 과적합(Overfitting)을 방지하기 위해 Randomly Shuffle 알고리즘을 통해 무작위로 섞었으며 층화추출방법으로 5개의 fold로 분할되어 구성하였다. 5개의 fold 중 1개는 교차검증을 위한 검증(validation) 데이터셋으로 활용되었으며 4개의 fold만 학습 데이터로 활용하였다.
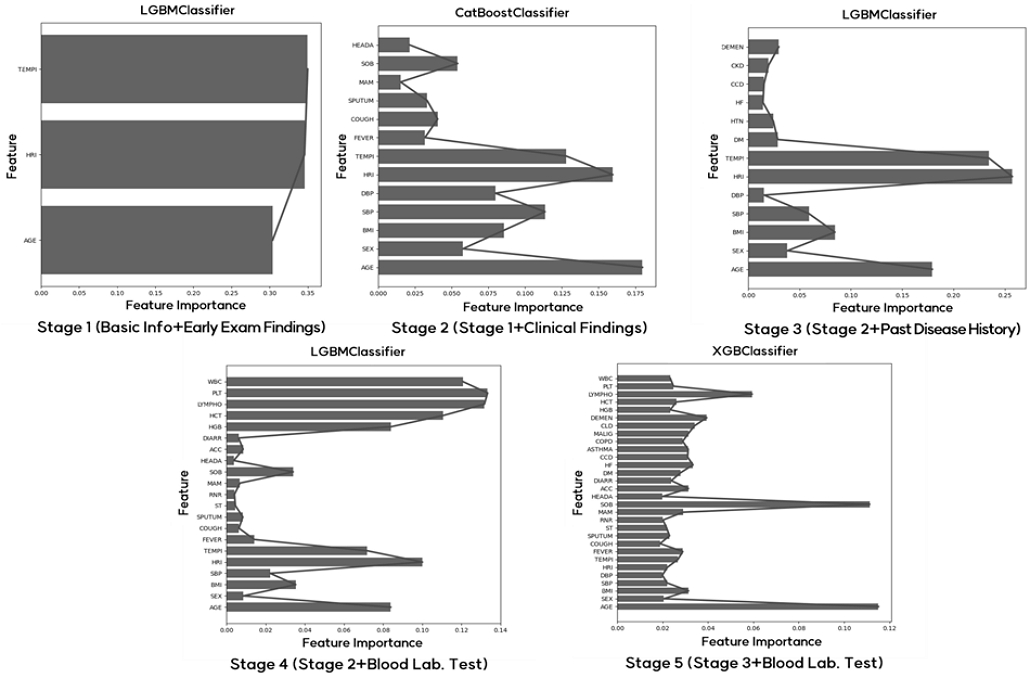
이후 교차검증 방법을 통해 반복적으로 모델을 학습하여 특징의 중요도가 낮은 특징들을 제거해나가는 재귀적 특징 제거 방법을 활용하여 예측에 영향력이 있는 특징만을 선별하였다. 특징 선택을 위한 반복적 모델학습을 위해 트리 기반 앙상블 모델인 LGBM, XGBoost, CatBoost 모델을 활용하였으며, 학습데이터 내에 있는 각각의 특징들이 중증도 예측에 미치는 영향에 대한 특징 중요도 값을 분석하여 그림 2와 같이 중요 특징들을 선별하였다.
Stage 1의 경우 LGBM모델을 통해 총 9개의 임상 변수 중 3개의 변수들이 선별되었고 체온(TEMPI), 심박수(HRI), 연령(AGE)순으로 중증도 예측에 대한 임상변수의 중요도 값을 나타냈다. Stage 2의 경우 CatBoost 모델을 통해 총 21개의 임상 변수 중 13개의 임상 변수들이 선별되었으며 Stage 1에서 활용한 연령(AGE), 심박수(HRI) 등이 높은 임상 변수 중요도 값을 나타내었다. 임상 소견 정보 중에서는 호흡곤란(SOB)가 높은 중요도를 나타내었다. Stage 3의 경우 LGBM 모델을 통해 총 32개의 임상 변수들 중 13개의 임상 변수들이 선별되었으며 현재/과거 병력과 관련된 기저질환 변수들보다는 Stage 1에서 활용한 연령(AGE), 심박수(HRI) 등과 같은 환자 기본 정보 및 초기 검사 정보 관련 변수들이 높은 임상 변수 중요도 값을 나타내었다.
Stage 4에서는 LGBM모델을 통해 총 26개 변수 중 21개의 변수가 선별되었고 혈소판(PLT), 림프구(LYMPHO)와 같은 혈액검사 임상 변수가 중증도 예측에 큰 영향을 미치는 높은 특징 중요도 값을 보였다. 마지막으로 Stage 5에서는 XGBoost 모델을 통해 총 37개의 임상 변수 중 30개의 변수가 선별되었고 연령(AGE), 호흡곤란(SOB), 림프구(LYMPHO) 순으로 임상 변수의 중요도 값을 나타냈다.
반면 임신여부(PREG), 임신주기(PREGGW), 피로(FM), 구토(VN)은 중증도 예측에 있어 예측 모델 기여 효과가 없는 임상 변수로 확인되었다.
3.5 다층 스태킹 앙상블 기반 중증도 예측 모델
앙상블(Ensemble) 기법은 다수의 약한 분류기(Weak classifier)들을 조합하여 하나의 강한 분류기(Strong classifier)를 생성하는 기법으로, 단일 모델 사용 시 발생 될 수 있는 모델 편향에 의한 오차를 줄일 수 있는 방법이다. 앙상블 기법에서는 대표적으로 데이터를 복원 추출하여 병렬적으로 학습한 후 투표(Voting) 방법을 통해 분류하는 배깅(Bagging)기법[16]과 데이터를 순차적으로 학습하여 오답에 대해 높은 가중치를 부여하는 부스팅(Boosting)기법[17]이 있지만 대부분 하나의 단일 모델을 기준으로 여러 개의 약한 분류기의 조합을 통해 진행된다. 반면 스태킹 앙상블 기법은 두 개 이상의 학습 서브 모델을 활용하여 각각의 모델의 예측 결과를 데이터로 학습하여 메타 분류기(Meta classifier)를 생성하는 기법으로, 개별 모델이 독립적이라고 가정하기 때문에 단일모델의 장단점을 보완할 수 있으며, 특히 단일모델에서 발생할 수 있는 이상치(Outlier)에 대응력이 높아 일반적으로 단일모델보다 성능이 좋은 것으로 알려져 있다.
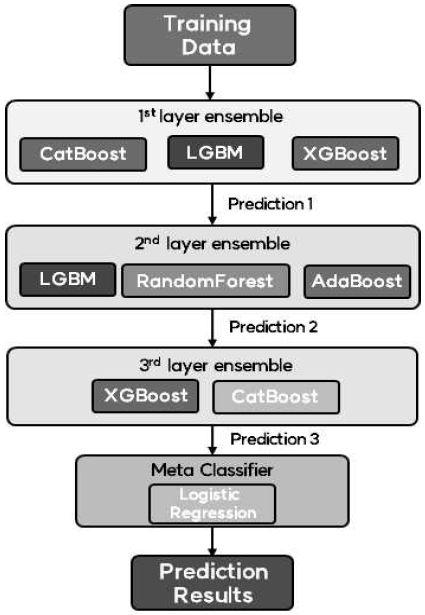
본 논문에서는 여러 개별 모델들의 학습 결과들이 각각 독립적이라고 가정하고 여러 개의 계층을 통해 서브 모델들을 조합한 다층 스태킹 앙상블 기반의 학습 방법을 활용한다. 본 논문에서 활용한 다층 스태킹 앙상블 모델은 그림 3과 같이 3-Layer 형태의 구조를 가지며, 서브 모델로는 XGBoost, LGBM, CatBoost, AdaBoost, RandomForest 등의 모델을 조합하여 구성되었다. 각 모델들의 결과를 결합하는 메타 분류기로는 Logistic Regression을 사용하였다.
먼저 각 층에서 조합될 모델들을 도출하기 위해 5개의 데이터 구성 전략에 대한 개별 모델 x, y 간의 중증도 예측 결과에 대한 상관계수를 구하고, 표 3과 같이 각 모델의 평균 상관계수를 기반으로 다양한 예측을 만들 수 있게 낮은 상관관계를 가지는 모델들끼리 조합될 수 있도록 구성하였다. 이후 재귀적 탐색을 통해 각 층에서 조합될 수 있는 모델의 종류 및 개수를 변화해가며 최적의 층의 개수를 탐색하였다.
| (2) |
Ⅳ. 실험 및 성능 평가
본 논문에서 제안하는 다층 스태킹 앙상블 기반 중증도 예측 모델을 평가하기 위해 데이터 구성 전략에서 구성된 5개의 각 Stage 학습데이터를 활용하여 학습을 수행하였으며 각 Stage 별로 데이터 분할 및 특징 선택과정에서 분리된 1,695개의 환자데이터를 활용하여 성능을 평가하였다.
또한 최종 다층 스태킹 앙상블 모델을 훈련할 때 설정되는 각각의 서브 모델들의 하이퍼파라미터(Hyperparameter)는 표 4와 같이 설정하였다.
성능 평가 지표로는 목표변수로 ‘경증’, ‘중증’, ‘최중증’ 등 3개의 임상 중증도를 예측하기 때문에 3개 클래스에 대한 평균 정밀도(Average Precision), 평균 재현율(Average Recall), 평균 F1 점수(Average F1-score)를 사용하였고 모델의 분류 성능을 추가로 확인하기 위해 평균 AUROC(Average Area Under the Receiver Operating Characteristic Curve)를 활용하여 검증하였다.
표 5는 데이터 구성 전략에 따른 6개의 단일모델 기반 중증도 예측 모델과 제안된 다층 스태킹 앙상블 기반 중증도 예측 모델의 비교 실험 결과를 보여준다. 전체적인 비교 실험 결과에서 단일 모델들은 각각의 모델 구조에 따라 서로 다른 성능들을 보여주고 있으며, 제안된 다층 스태킹 앙상블 모델은 모든 데이터 구성 전략에서 높은 성능을 보여주었다.
구체적으로 5개의 Stage로 구분된 데이터 구성 전략 중 환자의 기본 정보와 초기 검사 정보만 활용한 Stage 1의 중증도 예측 성능이 가장 낮았으며 환자의 임상 소견 정보 및 현재/과거 병력정보를 추가한 Stage 2와 Stage 3로 갈수록 중증도 예측 성능이 높아지는 것을 알 수 있었다. 따라서 환자의 기본 정보와 초기 검사 정보에서 임상 소견 정보 및 현재/과거 병력정보를 확보할 수 있다면 중증도 예측의 정확도를 증가시킬 수 있음을 알 수 있었다. 하지만 Stage 2와 Stage 3의 성능 평가 결과만 비교하였을 때, Stage의 3의 경우 Stage 2보다 환자의 현재/과거 병력과 관련된 기저질환 정보와 관련된 데이터들이 더 추가되었지만, 전체적으로 평균 F1 점수, 평균 AUROC 점수 등의 성능 평가 지표상승이 미비하여 환자의 기저질환과 관련된 현재/과거 병력정보는 정확도 상승에 큰 영향을 주는 변수로 활용되지는 못함을 알 수가 있었다.
또한 혈액검사를 활용한 Stage 4와 Stage 5의 경우 Stage 2와 Stage 3보다 단일모델 및 제안모델 모두 크게 정확도가 상승하여 헤모글로빈(HGB), 림프구(LYMPHO)등의 혈액검사 수치 정보가 코로나 중증도 예측에 있어 정확도를 향상시키는 주요 변수임을 알 수 있었다. 하지만 환자의 현재/과거 병력의 조회가 어려울 경우를 대비하여 Stage 2 데이터에 혈액검사 정보만 추가한 Stage 4 데이터가 Stage 5보다는 높은 정확도를 보여주었고, 제안된 모델 역시 Stage 4에서 가장 좋은 평균 F1 점수(0.9373) 및 AUROC(0.9545)를 보여주었다. 따라서, 혈액검사 수치 정보는 코로나 중증도 예측에 중요한 예측 변수로 활용될 수 있으나 환자의 현재/과거 병력정보는 크게 영향력을 미치는 변수는 아님을 확인할 수 있었다.
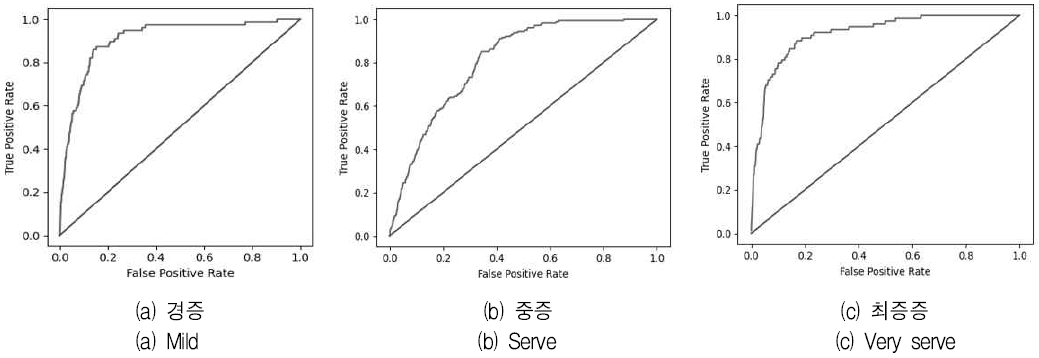
그림 4는 제안된 다층 스태킹 앙상블 모델에서 가장 좋은 AUROC 점수를 보여주었던 Stage 4 데이터의 ‘경증’, ‘중증’, ‘최중증’ 등 3개의 임상 중증도에 대한 AUROC Curve 그래프이며, 표 6은 3개의 임상 중증도에 대한 정밀도, 재현율, F1 점수이다.
3개의 임상 중증도에 대한 결과에서는 일상생활에 지장이 없거나 산소치료가 필요 없어 자가격리로 치료가 가능한 ‘경증’ 클래스와 사망 또는 인공 폐, 혈액 펌프 등의 기구를 이용한 중환자 치료가 필요한 ‘최중증’ 클래스의 경우 비교적 높은 F1 점수 및 AUROC를 보였으나 ‘중증’ 클래스의 경우에는 상대적으로 낮은 결과를 보였다. 이는 1,695개의 테스트 데이터셋 대부분이 ‘경증’ 클래스에 분포되어 있으며, 최중증의 경우에는 사망 또는 심각한 치료와 관련된 환자로서 데이터의 수치적 패턴이 비교적 명확하기 때문으로 판단된다.
Ⅴ. 결론 및 향후 과제
본 논문에서는 의료 자원이 제한된 코로나19 확산 상황에서 환자의 문진 데이터, 임상 소견 데이터 등 비교적 빠르게 확보할 수 있는 데이터를 기반으로 환자의 조기 분류가 가능한 임상 중증도 예측 모델을 제시하였다. 특히 5,651명의 대규모 코로나19 환자 데이터를 기반으로 모델을 구축하였으며, 데이터 사용 전략 및 재귀적 특징 제거 방법을 통해 선별된 특징만 사용하는 방법을 제시하였다. 또한 단일 모델의 편향성을 줄이기 위해 다층 형태의 스태킹 앙상블 모델을 제시하여 단일 모델보다 코로나19의 중증도를 정확하게 예측할 수 있었다. 하지만 ‘경증’, ‘중증’, ‘최중증’으로 구분된 임상 중증도 예측에서 극도로 치우친 데이터로 인해 중증 사례를 예측하는 성능이 예상보다 낮다는 한계점이 발견되었다. 또한 국내 환자의 데이터만 활용하였기 때문에 다른 인종 환자의 경우에 대한 일반화 가능성이 포함되지 않았다.
향후 연구에서는 다른 인종의 환자 데이터를 비롯한 다양한 데이터 세트에 대하여 AI 모델을 적용할 계획이다. 또한 일반 사용자가 쉽게 사용할 수 있으며 데이터 업데이트를 통해 추가 데이터 확보가 가능한 웹 애플리케이션 기반의 시스템 확장연구를 진행할 예정이다.
Acknowledgments
본 논문은 2021~2022년도 경상국립대학교 대학회계 연구비 지원사업에 의해 연구되었음
References
-
C. Kooli, "COVID-19: Public health issues and ethical dilemmas", Ethics, Medicine and Public Health, Vol. 17, pp. 1-9, Jun. 2021.
[https://doi.org/10.1016/j.jemep.2021.100635]

-
J. Park, G. Kim, H. Seok, H. Shin, and D. Lee, "Early triage of COVID-19 patients exploting Data-Driven Strategies and Machine Learning Techniques", In Proceedings of the 2022 International Conference on Electronics, Information, and Communication(ICEIC), Jeju, Korea, pp. 234-237, Feb. 2022.
[https://doi.org/10.1109/ICEIC54506.2022.9748839]

- Korea Disease Control and Prevention Agency (KCDA), Response Guidelines of Coronavirus Infectious Disease-19(For Local Government), 13-1 Edition, https://www.kdca.go.kr/board/board.es?mid=a20507020000&bid=0019&act=view&list_no=720440, [accessed: Sep. 04. 2022]
-
K. Hwang, "Physician’s Role in Community Treatment Center", Human Insurance Review & Assent(HIRA) Service Research, Vol. 2, No. 2, pp. 131-137, Feb. 2022.
[https://doi.org/10.52937/hira.22.2.1.131]

-
L. Yan, et al., "An interpretable mortality prediction model for COVID-19 patients", Nature Machine Intelligence, Vol. 2, pp. 283-288, May 2020.
[https://doi.org/10.1038/s42256-020-0180-7]

-
W. Shang, J. Dong, Y. Ren, M. Tian, W. Li, J. Hu, and Y. Li, "The value of clinical parameters in predicting the severity of COVID-19", Journal of Medical Virology, Vol. 92, No. 10, pp. 2188-2192, Oct. 2020.
[https://doi.org/10.1002/jmv.26031]

-
W. Liang, et al., "Early Triage of critically ill COVID-19 patients using deep learning", Nature Communication, Vol. 11, No. 3543, pp. 1-7, Jul. 2020.
[https://doi.org/10.1038/s41467-020-17280-8]

-
C. Jin, et al., "Development and evaluation of an artificial intelligence system for COVID-19 diagnosis", Nature Communication, Vol. 11, No. 5088, pp. 1-14, Oct. 2020.
[https://doi.org/10.1038/s41467-020-18685-1]

-
Q. Xu, et al. "AI-based analysis of CT images for rapid triage of COVID-19 patients", npj Digital Medicine, Vol. 4, No. 75, pp. 1-11, Apr. 2021.
[https://doi.org/10.1038/s41746-021-00446-z]

-
M. M. Al Rahhal, et al., "COVID-19 Detection in CT/X-ray Imagery Using Vision Transformers", Journal of Personalized Medicine, Vol. 12, No. 2, pp. 1-17, Feb. 2022.
[https://doi.org/10.3390/jpm12020310]

-
C. An, H. Lim, D. Kim, J. Chang, Y. Choi, and S. Kim, "Machine learning prediction for mortality of patients diagnosed with COVID-19: a nationwide Korean cohort study", Scientific Reports, Vol. 10, No. 18716, pp. 1-11, Oct. 2020.
[https://doi.org/10.1038/s41598-020-75767-2]

-
Y. Zoabi, S. Deri-Rozov, and N. Shomron, "Machine learning-based prediction of COVID-19 diagnosis based on symptoms", npj Digital Medicine, Vol. 4, No. 3, pp. 1-5, Jan. 2021.
[https://doi.org/10.1038/s41746-020-00372-6]

-
J. Kim, H. Lim, J. Ahn, K. Lee, K. Lee, and K. Koo, "Optimal Triage for COVID-19 Patients Under Limited Health Care Resources With a Parsimonious Machine Learning Prediction Model and Threshold Optimization Using Discrete-Event Simulation: Development Study", JMIR Medical Informatics, Vol. 9, No. 11, pp. 1-15, Nov. 2021.
[https://doi.org/10.2196/32726]

- Cleveland Clinic, "Pulse & Heart Rate", https://my.clevelandclinic.org/health/diagnostics/17402-pulse—heart-rate, [Accessed: Aug. 15, 2022]
- Healthline, "What is the Normal Body Temperature Range?", https://www.healthline.com/health/what-is-normal-body-temperature, [accessed: Aug. 03, 2022]
-
L. Breiman, "Bagging Predictors", Machine Leaning, Vol. 24, No. 2, pp. 123-140, Aug. 1996.
[https://doi.org/10.1007/BF00058655]

- RE. Schapire, "A brief introduction to boosting", In Proceedings of the IJCAI International Joint Conference on Artificial Intelligence, pp. 1401-1406, Aug. 1999. https://dl.acm.org/doi/10.5555/1624312.1624417, .

2006년 : 호주뉴캐슬대학교 컴퓨터공학과(공학사)
2007년 : 호주뉴캐슬대학교 정보공학과(공학석사)
2017년 : 한양대학교 컴퓨터공학과(공학박사)
2021년 9월 ~ 현재 : 경상국립대학교 컴퓨터과학부 교수
관심분야 : 인공지능, 시멘틱 헬스케어, 데이터마이닝