UNETR 기반 3D MRI 뇌 영상에서 다발성 경화증 병변 검출
초록
다발성 경화증 진단은 매우 어렵고 병리사들의 노고가 많이 든다. 최근에 딥러닝 기술을 이용한 의료 영상 분석 방법의 발전으로 특정 질환 분류 및 진단 효율성과 정확도가 높아지고 있다. 본 논문에서는 트랜스포머를 인코더로 대체한 UNETR 딥러닝 모델을 기반으로 다발성 경화증 병변의 검출 방법을 제안한다. 특히, 의료 분야 특성상 영상 데이터 수가 적기 때문에 모델의 충분한 학습과 과적합을 방지하기 위해 데이터 증강 기법을 적용하였다. 제안하는 방법의 성능은 2008 MICCAI MS Lesion Segmentation Challenge 데이터셋을 사용하여 검증하였고, 그 결과 75.22% DSC와 56.40% mean IoU 정확도를 달성하여, Residual 3D U-Net 기반 방법에 대비하여 각각 4.54%, 3.61% 향상하였다.
Abstract
Diagnosis of multiple sclerosis is difficult and requires a lot of effort by pathologists. Recently, with the development of medical image analysis methods using deep learning, the efficiency and accuracy of specific disease classification and diagnosis are increasing. In this paper, we propose a method for detecting multiple sclerosis lesions based on the UNETR deep learning model that replaces a transformer with encoders. In particular, since the number of image data is small due to the characteristics of the medical field, a data augmentation technique is applied to prevent sufficient training and overfitting of the model. The performance of the proposed method was verified using the 2008 MICCAI MS Lesion Segmentation Challenge dataset and achieved 75.22% DSC and 56.40% mean IoU accuracy, which improved 4.54% and 3.61% respectively compared to the Residual 3D U-Net-based model.
Keywords:
multiple sclerosis lesion, brain MRI, UNETR, transformer, deep learningⅠ. 서 론
다발성 경화증(MS, Multiple Sclerosis)은 중추신경계에 발생하는 만성 질환으로 환자의 면역체계가 건강한 세포와 조직을 공격하는 자가면역 질환이다[1]. 초기 증상이 대부분 뚜렷하지 않지만, 우울증 및 기억력 장애가 나타날 수 있고, 질환이 진행되면 인지기능 장애가 나타나 전문의에게 정확하게 진단 및 치료를 받는 것이 중요하다[2].
그러나 임상에서 다발성 경화증 환자의 진단은 매우 어렵고 정확한 진찰과 뇌척수 MRI, 시신경 자극유발 전위검사 등으로 병변(Lesion)이 공간적으로 적어도 2개 이상이 있는지 확인해야 한다[3]. 이 진단 과정에서 병리사들의 인적 및 시간적 노고가 많이 들어서 컴퓨터 비전 분야에서는 의료 영상 분석 기술들이 개발되어 다양한 분석에 도움을 주고 있다.
기존 의료 영상 분석에서는 SIFT, Harr Wavelet, HOG, LBP 등 고정적 특징 추출 방법이나 각각의 작업 목표나 영상에 특화된 특징 추출하는 방법을 사용했지만, 영상별로 성능 편차가 크거나 복잡하고 내재된 특징을 추출하지 못하는 한계가 있다[4].
최근에는 딥러닝 기술로 고정적 특징 추출 방법의 성능 한계를 극복하며 의료 영상을 자동 분석해서 특정 질환 분류 및 진단 효율성과 정확도를 높이고 있다. 또한 NLP(Natural Language Processing) 분야에서 사용되는 트랜스포머(Transformer)를 이미지 분류 분야에 맞게 변형하여 CNN(Convolutional Neural Network) 구조 대부분을 대체하는 ViT(Vision Transformer) 연구도 증가하는 추세이다. 그러나 딥러닝 모델 학습에 필요한 영상 데이터와 레이블은 병리사들의 수작업과 개인 정보 문제로 다량 수집하기 어렵다. 이와 같은 데이터 부족은 학습 과정에서 모델의 과적합을 야기하며 이를 방지하기 위해서는 데이터 증강으로 다양한 변이를 만들어 모델을 학습시켜야 한다[5][6].
본 논문에서는 3D U-Net에서 스킵 연결로 디코더에 트랜스포머 스택으로 구성된 인코더를 연결한 수축 확장 패턴을 적용한 UNETR 모델을 이용하여 3D MRI 뇌 영상에 대해 다발성 경화증 병변의 검출 방법을 제안한다[7]. 제안한 방법은 데이터를 증강한 후에 다양한 실험과 파라미터 설정을 통해 다발성 경화증 병변 검출에 맞게 최적화하였고, 기존 3D U-Net 수축 경로에 잔차 신경망을 추가한 Residual 3D U-Net 모델보다도 성능을 향상시켰다[6].
본 논문은 다음과 같이 구성되어 있다. 2절에서 ViT 및 트랜스포머 U-Net에 관해 설명한다. 3절은 뇌 3D MRI를 이용한 데이터 증강 기법과 UNETR 기반 다발성 경화증 병변 검출 방법을 제안한다. 4절에서는 실험 결과를 제시하고 5절에서 결론을 짓는다.
Ⅱ. 관련 연구
2.1 Vision 트랜스포머
ViT는 NLP에서 자주 사용되는 트랜스포머를 비전 분야에 적용한 것이다. Dosovitsky et. al.은 NLP의 트랜스포머 스케일링이 이미지 분류에 적용할 수 있고, 최신 CNN 구조보다 좋은 결과를 보이며 모델 훈련에 필요한 계산량이 적은 것을 보였다[8]. 그러나 ViT는 컨볼루션 계층과 다르게 귀납적 편향이 부족하므로 작은 데이터셋에서 좋은 성능을 내지 못한다.
Zhou et. al은 트랜스포머 블록이 깊어질수록 ViT에 비효율적이므로 단순히 Multi-Head Self-Attention 모듈을 Re-Attention으로 교체하여 깊은 구조를 갖는 ViT를 개선하였다[9]. 이와 다르게, Zhang et. al은 다중 스케일 이미지에서 계산 및 메모리 복잡성을 관리하고 포괄적 인코딩을 제공하여 ResNet과 같은 기존 CNN과 동일한 네트워크 구조를 가진 모델을 제안하였다[10].
이미지뿐만 아니라 비디오에 대해서도 트랜스포머를 적용하고 있다. Arnab et. al은 추출된 비디오 클립을 Uniform 프레임 샘플링과 Tublet 임베딩의 두 가지 방법으로 처리하고 위치 임베딩을 통해 트랜스포머 입력 차원에 맞게 재구조화하였다. 특히, 모델에 Regulariser를 추가할수록 점진적으로 성능이 향상되며 ViT와 다르게 작은 데이터셋에서도 좋은 성능을 나타내는 것을 보였다[11]. 기존 비디오 행동 인식에서는 다중-모달 정보를 활용하지 않고, 프레임별 광학 흐름 획득에 시간이 오래 걸려서 계산적 병목 현상을 일으키는 문제점이 있다.
이를 해결하기 위해 Chen and Ho는 비디오 행동 인식에 트랜스포머를 활용하여 이미지 공간에서 시공간 모달리티 4D 볼륨으로 Self-Attention을 확장하는 모델을 제안하였다[12].
2.2 Transformer U-Net for medical image segmentation
딥러닝 기술의 출현 이후 FCNN(Fully CNN) 모델 및 U자 모양의 인코더-디코더 구조를 갖는 U-Net 모델이 다양한 의료 영상의 의미론적 분할에서 좋은 결과를 보였다[13].
FCNN 기반 방법은 강력한 표현 학습 성능을 갖고 있지만 장거리 의존을 학습하는데 제한이 있다. 이를 극복하기 위해 Hatamizadeh et. al은 컴퓨터 비전에서 트랜스포머를 백본 인코더로 사용하는 것이 장거리 의존 및 전역 컨텍스트에서 유용한 것을 이용하여 U-Net의 인코더 부분에 트랜스포머를 사용하는 구조를 제안하였고 해당 모델이 의료 영상에 해부학적 관계를 나타내는데 잠재력이 있는 것을 보였다[7]. 또한 트랜스포머로 인한 특징 해상도 손실 부분은 CNN의 고해상도 공간 정보와 트랜스포머에 인코딩된 전역 컨텍스트를 활용하여 보완하였다[14].
Cao et. al은 CNN 기반 방법들이 컨볼루션에서 과분할 문제가 발생 경향이 있어서 트랜스포머를 컨볼루션 없이 스킵 연결이 있는 U자형 순수 트랜스포머 구조를 제안했다. 이는 Swin Transformer[15]블록 기반으로 구축되며, 2D 의료 영상에서 전역 및 장거리 의미 정보를 더 잘 학습하여 좋은 분할 성능을 보였다[16]. 그러나 현재 의료 영상 대부분이 3D 형태로 제공되고, 2D 기반 방법은 단일 슬라이스 내에서 어텐션 인코딩만 고려하여 3D 볼륨에 의해 제공되는 축 정보를 활용하지 못한다. Yan et. al은 축에 따른 어텐션과 단일 슬라이스 내에 어텐션을 각각 계산해주며, 이 둘을 합쳐 최종 분할맵을 제공하는 방식을 제안하였고, 이는 3D 영상으로 인해 제한되는 GPU 메모리 문제를 해결하였다[17].
Ⅲ. UNETR 기반 3D MRI 뇌 영상에서 다발성 경화증 병변 검출 방법
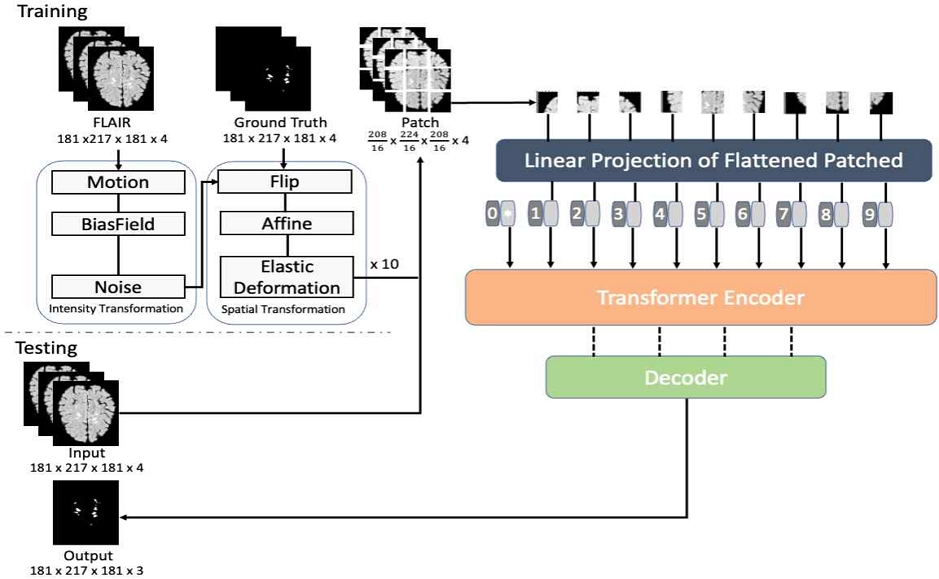
본 논문에서 제안하는 UNETR 딥러닝 모델에서 다발성 경화증 병변 검출 방법의 전반적인 구조는 그림 1과 같다.
학습 및 평가 과정으로 구분할 수 있으며, 각 과정에 데이터 전처리 및 증강 처리, 이미지 패치 후 임베딩 처리와 인코더, 스킵 연결, 디코더 모듈을 포함한다.
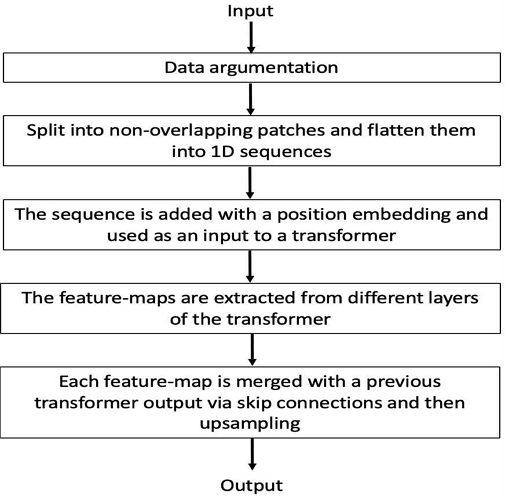
다발성 경화증 병변 검출 과정은 그림 2의 플로우차트와 같이 이루어진다. 의료 분야 특성상 학습 과정에 있어서 다발성 경화증 병변 환자의 MRI 영상 데이터 수가 부족하므로 먼저 입력으로 들어오는 데이터를 증강한다. 각 영상 데이터에 대해서 밝기값 변환 및 공간 변형 순서로 10번의 패치를 반복하여 랜덤하게 증강한다. 증강이 완료된 데이터는 트랜스포머에 입력에 맞게 겹치지 않는 패치 단위로 분리하고 1D 시퀀스로 평탄화 후에 공간 정보를 보존하기 위해 위치 임베딩을 더해준다. 이후 최종적으로 나온 특징맵을 업샘플링하며 스킵 연결을 통해 트랜스포머의 각 계층 구간에서 나온 특징맵과 결합하며 결과를 출력한다.
3.1 UNETR 아키텍쳐
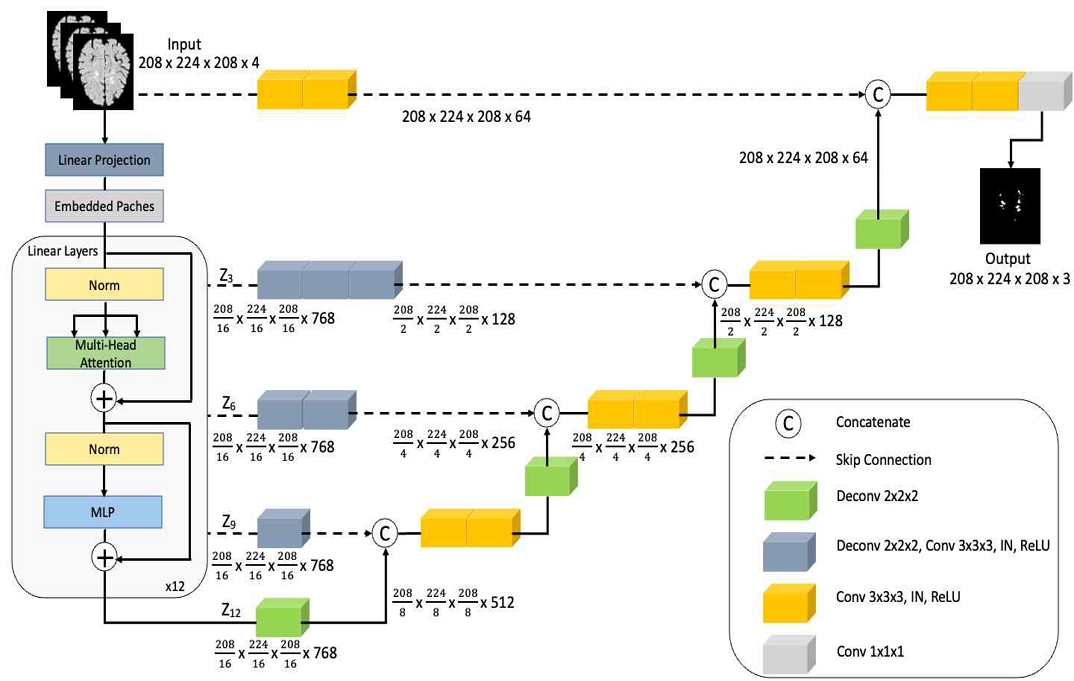
제안하는 모델은 그림 3과 같이 UNETR 구조를 이용하였다. UNETR은 전반적으로 트랜스포머 인코더와 CNN 기반 디코더가 스킵 연결을 통해 구성된 U자형 모형의 수축 확장 패턴을 활용한다[7].
수축 경로에서는 이미지를 겹치지 않는 패치 단위로 구분하고, 각 패치들에 대해 평탄화를 수행한 후에 1D 시퀀스로 만들어 인코더로 입력한다. 그 후에 MSA(Multi-Head Self-Attention)와 MLP(Multi Layer Perceptron)를 포함하는 트랜스포머 블록이 선형 계층 수만큼 수행되고 최종적으로 선형 연산을 통해 특징맵을 추출한다.
확장 경로로 전환되는 전환 구간에서는 최종적으로 나온 특징맵에 디컨볼루션 계층을 적용하여 해상도를 2배 증가시킨 다음, 이전에 Z9 구간의 결과로 나온 특징맵을 연결한다. 그리고 디코더 구간의 3×3×3 컨볼루션 계층을 수행한 후 다시 디컨볼루션 계층을 통해 업샘플링을 하며 해상도를 2배 증가시킨다. 이 과정을 Z9, Z6, Z3 구간에서도 반복하여 최종적으로 1×1×1 컨볼루션을 수행하여 의미론적 분할 예측 결과를 획득한다.
3.2 인코더
인코더는 트랜스포머를 이미지 분류에 맞게 구조를 변형한 ViT-Base 모델[8]을 사용하였고, 계층 정규화[18], MSA, MLP로 구성된 트랜스포머 블록이 선형 계층 수만큼의 스택 구조를 이루고 있다.
트랜스포머는 3D 영상을 패치별로 자른 후 1D 시퀀스로 변환시키고 트랜스포머 블록에 입력한다. 또한 패치 과정에서 공간 정보의 손실 방지를 위해 각 패치별로 768 크기의 위치 임베딩을 추가한다. 기존 구조와 다르게 MLP에서 활성화 함수로 ReLU 대신 2계층 GELU를 사용하고 계층 정규화가 MSA와 MLP 이전에 수행되면서 깊은 계층에서도 학습을 잘되도록 한다.
Vit-Base 모델은 12개의 선형 계층으로 구성되어 트랜스포머 블록이 총 12번 수행되며 각 구간(Z12, Z9, Z6, Z3)에서 크기 특징맵을 출력한다. 출력된 특징맵은 스킵 연결을 통해 N개의 2×2×2 디컨볼루션 계층을 수행하여 해상도를 2N배 증가시키고, 최종 출력 구간 Z12를 제외한 나머지 구간들은 인스턴스 정규화 계층[19] 및 ReLU 활성화 함수로 이어진 3×3×3 컨볼루션 계층을 수행하여 임베딩 크기를 연결 공간의 입력 크기에 맞게 각 512, 256, 128, 64 크기의 텐서로 만든다.
3.3 디코더
디코더는 CNN 기반의 컨볼루션 계층 및 디컨볼루션 계층으로 구성되어 있으며 인코더에 스킵 연결을 통해 연결되어 있다.
컨볼루션 계층 블록은 2가지 커널 크기 종류가 있다. 3×3×3 컨볼루션 계층은 인코딩 컨볼루션 계층과 동일한 형태로 이전 업샘플링 특징맵과 스킵 연결의 특징맵을 결합하여 출력된 특징맵에 대해서 두 번씩 반복 수행하여 임베딩 크기를 다음 결합 공간에 맞는 512, 256, 128, 64 크기의 텐서로 재구성한다. 1×1×1 컨볼루션은 최종적으로 업샘플링된 값과 원본 입력값이 연결된 후 컨볼루션 계층을 수행한 208×224×208×32 크기의 특징맵에 소프트맥스 활성화 함수를 통해 3차원의 복셀별로 의미론적 예측 결과를 출력한다.
디컨볼루션 계층은 2×2×2 커널 크기로 두 차례의 3×3×3 컨볼루션을 수행한 특징맵에 해상도를 2배씩 증가시키는 업샘플링을 수행하여 스킵 연결에서의 특징맵과 동일한 크기의 텐서로 만든다.
3.4 스킵 연결
스킵 연결은 인코더에서 트랜스포머 블록을 수행하여 구간마다 나온 특징맵에 대해서 2×2×2 디컨볼루션 계층과 잔차 블록을 사용한 3×3×3 컨볼루션 계층을 순차적으로 수행한다. 이후 업샘플링된 특징맵과 결합하여 인코더 구간에서 다운샘플링을 수행하는 동안 손실된 공간 정보를 복구시킨다.
3.5 데이터 증강 방법
의료 분야의 특성상 영상 데이터 수가 적어 모델을 충분히 학습시키지 못하고 과적합이 나타나는 문제가 있다. 이를 해결하기 위해 MRI 영상에 TorchIO가 제공하는 기능을 활용하여 랜덤 패치, 정규화, 다양한 증강 기법을 통한 전처리를 수행하여 모델 학습에 충분한 데이터를 생성하였다.
증강 과정에서 영상 데이터는 다발성 경화증 병변 영역이 포함되도록 208×224×208 크기의 패치를 생성하고 각 패치마다 증강 기법을 적용하였다. 생성된 패치는 Z 정규화를 적용한 후 밝기 변환과 공간 변형을 순차적으로 수행하였다. 밝기 변환은 모션, 바이어스필드, 노티스를 사용하였고 공간 변형에는 플립, 어파인, 탄성 변형을 사용하였다. 영상 데이터와는 다르게 레이블 데이터에 대해서는 공간 변형만 수행하였다.
Ⅳ. 실험 결과 및 분석
본 실험은 Intel i7-7700, nVidia RTX 3090, 32GB RAM 하드웨어 및 Windows 10 Pro 운영체제 환경에서 진행했으며 데이터로더는 파이토치로 구현하였고, 모델은 MONAI 깃허브[21]를 참고하였다.
4.1 실험 데이터셋
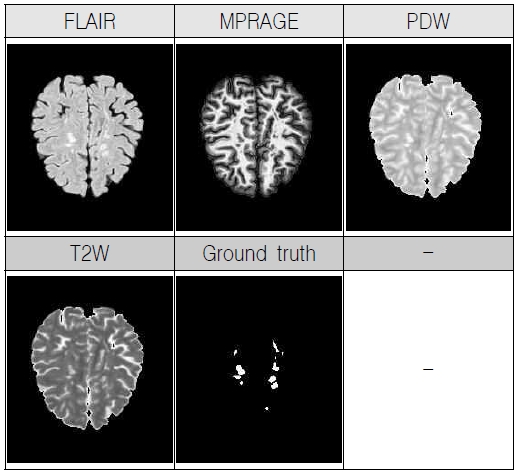
제안하는 UNETR 모델 기반 다발성 경화증 병변 검출 방법의 성능 평가를 위해, 2008 MICCAI MS Lesion Segmentation Challenge 데이터셋을 사용하였다[20]. 그림 4에 각 채널별 단면 영상을 도시하였다. 데이터셋은 임상에서 병리사가 다발성 경화증 병변 환자에서 검출한 것으로 훈련 5명, 테스트 14명으로 구분되어 있다.
각 데이터는 181×217×181 크기의 전처리 상태로 제공되며 MRI 뇌 영상 및 레이블된 마스크 데이터로 FLAIR, MPRAGE, PDW, T2W 4개 채널과 Ground Truth 채널이 포함된다. Ground Truth 채널은 병변 부분인 다발성 경화증 병변과 그 외 나머지 영역의 2가지 레이블로 구성된다.
4.2 제안하는 모델 구현 및 실험
제안하는 모델의 실험을 위해 훈련 데이터셋의 학습 및 검증 데이터 비율을 8 : 2로 사용하였고 테스트는 따로 제공된 데이터셋을 사용하였다. 데이터 증강은 학습 데이터셋에만 적용하였고 검증 데이터셋은 Z 정규화만 적용해 모델을 훈련시켰다.
학습 과정에서 배치 크기는 1로 설정하였고 UNETR 모델의 파라미터로는 특징 크기는 12로 설정하고 인코더로 트랜스포머 기반 ViT-Base 모델을 사용하기 위해 계층은 12, 은닉 크기는 768, 다층 퍼셉트론 크기는 3072, 어텐션 헤드는 12로 설정하였다. 또한 패치 크기는 16×16×16으로 설정하였으며, 이는 입력되는 이미지 데이터를 해당 크기의 겹치지 않는 패치로 변환한다.
학습 데이터는 I/O 단일 채널과 Adam 최적화 알고리즘을 사용하였고, 학습률은 10-3, 가중치 감쇠는 10-10로 설정했으며, 손실 함수는 Soft Dice Loss[22]와 Cross-Entropy Loss를 조합한 DiceCELoss를 사용하였다. 학습에 사용한 파라미터는 표 1과 같다.
Residual 3D U-Net 모델과 성능 비교를 위해 학습 파라미터에서 학습률을 제외한 나머지 파라미터는 동일한 값을 설정하였다. 비교를 위해서 유사도 메트릭으로 DSC(Dice Similarity Coefficient)와 mean IoU(Intersection over Union)를 사용하였고 학습은 200 epoch까지 진행하였다.
4.3 실험 결과
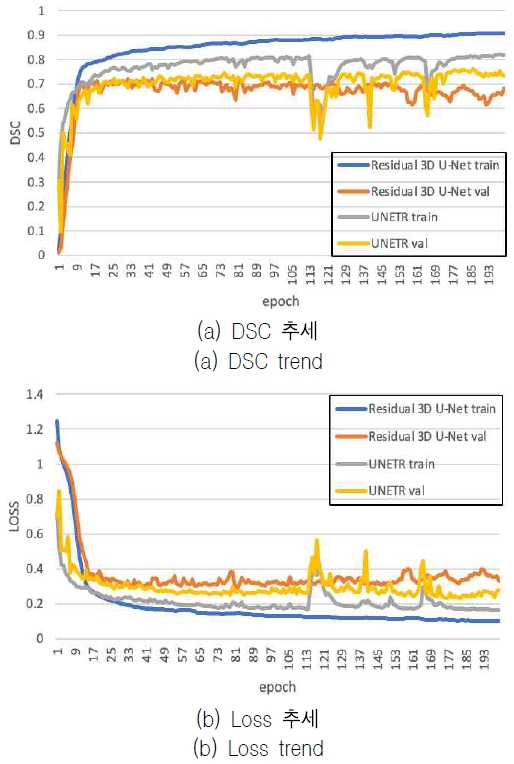
UNETR 모델과 Residual 3D U-Net 모델[6]의 성능 비교를 위해 동일한 학습 및 검증 데이터로 실험을 하였다. 그림 5에 MRI 영상 채널 중 FLAIR 채널의 epoch에 따른 정확도 추세를 도시하였다.
Residual 3D U-Net 모델의 훈련 정확도가 UNETR 모델보다 높고 향상되고 있지만 검증 정확도는 일정한 추세를 보였다. 그러나 UNETR 모델은 훈련 정확도가 낮지만, 검증 정확도는 Residual 3D U-Net 모델보다 높고 미세하지만 조금씩 향상하는 추세를 보였다.
이는 데이터 증강으로 데이터 수를 늘렸으나 Residual 3D U-Net 모델의 학습에는 부족하여 학습을 계속할수록 훈련 데이터셋에 과적합되지만, UNETR 모델은 적은 데이터에서도 안정적이고 과적합되지 않으며 훈련과 검증 데이터셋 모두 학습이 잘되는 것을 알 수 있다.
그러나 UNETR 모델도 특정 epoch에서 학습률의 보폭 간격 차이로 오버슈팅(Over-shooting)이 발생한 것을 알 수 있다.
표 2에는 UNETR과 Residual 3D U-Net 모델뿐만 아니라 3D U-Net[23]에 대하여 DSC와 mean IoU 유사도 메트릭을 이용하여 성능을 비교한 결과를 제시하였다. 3D U-Net과 Residual 3D U-Net 모델의 실험에서는 3D U-Net 모델이 리소스를 많이 요구하여 메모리 문제로 데이터 증강 단계에서 하나의 영상에 대해 랜덤으로 144×144×144 크기의 패치로 만들어 실험했고, UNETR 모델에서는 인코더에서 트랜스포머를 통해 패치 단위로 분할하기 때문에 208×224×208 크기의 패치로 만들어 실험했다[6].
Residual 3D U-Net 모델이 3D U-Net 모델보다 FLAIR와 MPRAGE 채널에서 DSC와 mean IoU가 향상되었지만 T2W와 PDW 채널은 3D U-Net 모델이 더 높게 나타났다. UNETR 모델의 경우 대체로 네 개의 채널 정확도가 이전 두 모델보다 향상되었지만 T2W와 PDW 채널의 mean IoU가 여전히 3D U-Net 모델보다 낮은 것을 알 수 있다. 실험 결과로 UNETR 모델은 loss가 가장 낮은 지점에서 DSC와 mean IoU 정확도가 각각 75.28%, 56.40%를 달성하였고, Residual 3D U-Net 모델보다 4.54%, 3.61% 향상되었지만, FLAIR 채널을 제외한 나머지 채널에서는 mean IoU가 근소하게 향상되었다.
세 모델에서 공통적으로 FLAIR 채널이 다른 채널보다 비교적 높은 정확도를 나타낸 것을 알 수 있다. 이를 통해 다발성 경화증 병변 검출을 위한 의미론적 분할에는 세 모델 모두 FLAIR 채널이 적합하며 본 논문에서 제안하는 UNETR 모델이 가장 높은 정확도를 얻을 수 있다는 것을 알 수 있다.
Ⅴ. 결론 및 향후 과제
다발성 경화증 병변 검출을 위해 컴퓨터를 이용한 의료 영상 분석 방법이 개발되어 진단 과정에 도움을 주고 있다. 최근에는 딥러닝 기술로 기존 고정적 특징 추출 성능의 한계를 극복하며 질환의 분류 및 진단 정확도를 향상하고 있다.
본 논문에서는 3D U-Net에 트랜스포머 인코더를 적용한 UNETR 딥러닝 모델을 통해 3D MRI 뇌 영상에서 다발성 경화증 병변 검출 방법을 제안하였다. 제안한 모델의 과적합을 방지하기 위해 데이터셋에 밝기값 변환 및 공간 변형의 증강 기법을 적용하였다. 특히, 의미론적 분할의 좋은 성능을 위해 UNERT 모델을 도입해 수축 경로에서 트랜스포머를 인코더로 사용함으로써 컨볼루션에 의존하지 않고 트랜스포머가 이미지 패치들에 사용되도록 하였다.
또한 3D 영상을 1D 시퀀스로 변환해 장거리 의존 문제를 해결하였고, 기존 3D U-Net 모델보다 요구하는 리소스가 적어서 효율성을 향상하였다. 제안하는 모델의 성능은 2008 MICCAI MS Lesion Segmentation Challenge 데이터셋을 사용하여 검증하였고 FLAIR 채널 기준 DSC와 mean IoU 정확도가 75.22%, 56.40%로 Residual 3D U-Net 대비하여 각각 4.54%, 3.61% 향상하였다. 이를 통하여 제안한 모델이 기존 3D U-Net과 Residual 3D U-Net 모델보다 높은 의미론적 분할 정확도를 달성할 수 있음을 보였다.
그러나 실험에서 epoch 수가 증가할수록 Residual 3D U-Net 모델은 과적합 경향을 보이고 UNETR 모델은 오버슈팅이 발생한다. 추후 연구에서는 모델 구조 및 데이터 전처리 개선을 통해 정확도를 향상하거나 GPU 메모리 사용량을 줄여 학습에 사용된 파라미터를 높이고, 학습률을 조정하거나 다른 손실 함수를 사용하여 오버슈팅을 줄일 예정이다.
Acknowledgments
This work was supported by the Basic Science Research Program through the National Research Foundation of Korea (NRF) funded by the Ministry of Education (2020R1F1A1057742)
References
- Asan Medical Center, "Multiple Sclerosis", https://www.amc.seoul.kr/asan/healthinfo/disease/diseaseDetail.do?contentId=32005, . [accessed: Aug. 23, 2022]
- Seoul National University Hospital, "Multiple Sclerosis", http://www.snuh.org/health/nMedInfo/nView.do?category=DIS&medid=AA000105, . [accessed: Aug. 23, 2022]
-
K. K. Kim, "Multiple sclerosis", Journal of Korean Medical Association, Vol. 56, No. 8, pp. 702-708, Aug. 2013.
[https://doi.org/10.5124/jkma.2013.56.8.702]

- Y. H. Lee, H. J. Kim, G. B. Kim, and N. K. Kim, "Deep Learning-based Feature, Extraction for Medical Image Analysis", Journal of KSIIM, Vol. 20, pp. 1-12, 2014.
- J. J. Lee, H. T. Han, Y. U. Choi, B. H. Choi, C. M. Kim, H. S. Kim, S. J. Eom, and H. Y. Lee, "Multiple Sclerosis Lesion Segmentation using Deep Learning", Proc. of 2021 KIIT Conf., Jeju, Korea, pp. 523-525, Jun. 2021.
-
B. H. Choi, J. J. Lee, Y. U. Choi, H. T. Han, and H. Y. Lee, "Semantic Segmentation of Multiple Sclerosis Lesion using Residual 3D U-Net", Journal of KIIT, Vol. 19 No. 11, pp. 81-90, Nov. 2021.
[https://doi.org/10.14801/jkiit.2021.19.11.81]

-
A. Hatamizadeh, Y. Tang, V. Nath, D. Yang, A. Myronenko, B. Landman, H. R. Roth, and D. Xu, "UNETR: Transformers for 3d Medical Image Segmentation", Proc. of IEEE. Conf. on Applications of Computer Vision, Waikoloa, HI, USA, pp. 1748-1758, Jan. 2022.
[https://doi.org/10.1109/WACV51458.2022.00181]

- A. Dosovitskiy, L. Beyer, A. Kolesnikov, D. Weissenborn, X. Zhai, T. Unterthiner, M. Dehghani, M. Minderer, G. Heigold, S. Gelly, J. Uszkoreit, and N. Houlsby, "An image is worth 16x16 words: Transformers for image recognition at scale", arXiv:2010.11929, , Jun. 2021.
- D. Zhou, B. Kang, B. Jin, L. Yang, X. Lian, Z. Jiang, Q. Hou, and J. Feng, "Deepvit: Towards deeper vision transformer", arXiv preprint arXiv:2103.11886, , Apr. 2021.
-
P. Zhang, X. Dai, J. Yang, B. Xiao, L. Yuan, L. Zhan, and J. Gao, "Multi-Scale Vision Longformer: A New Vision Transformer for High-Resolution Image Encoding", Proc of IEEE Int. Conf. on Computer Vision, Montreal, QC, Canada, pp. 2978-2988, Oct. 2021.
[https://doi.org/10.1109/ICCV48922.2021.00299]

-
A. Arnab, M. Dehghani, G. Heigold, C. Sun, M. Lučić, and C. Schmid, "ViViT: A Video Vision Transformer", Proc. of IEEE Int. Conf. on Computer Vision, Montreal, QC, Canada, pp. 6816-6826, Oct. 2021.
[https://doi.org/10.1109/ICCV48922.2021.00676]

-
J. Chen and C. M. Ho, "MM-ViT: Multi-Modal Video Transformer for Compressed Video Action Recognition", Proc of IEEE Winter Conf. on Applications of Computer Vision, Waikoloa, HI, USA, pp. 786-797, Jan. 2022.
[https://doi.org/10.1109/WACV51458.2022.00086]

-
O. Ronneberger, P. Fischer, and T. Brox. "U-net: Convolutional networks for biomedical image segmentation", Proc. of Int. Conf. on Medical image computing and computer-assisted intervention, pp. 234-241, Nov. 2015.
[https://doi.org/10.1007/978-3-319-24574-4_28]

- J. Chen, Y. Lu, Q. Yu, X. Luo, E. Adeli, Y. Wang, L. Lu, A. L. Yuille, and Y. Zhou, "Transunet: Transformers make strong encoders for medical image segmentation", arXiv preprint arXiv:2102.04306, , Feb. 2021.
-
Z. Liu, Y. Lin, Y. Cao, H. Hu, Y. Wei, Z. Zhang, S. Lin, and B. Guo, "Swin transformer: Hierarchical vision transformer using shifted windows", Proc of IEEE Int. Conf. on Computer Vision, Montreal, QC, Canada, pp. 9992-10002, Oct. 2021.
[https://doi.org/10.1109/ICCV48922.2021.00986]

- H. Cao, Y. Wang, D. Jiang, X. Zhang, Q. Tian, and M. Wang, "Swin-unet: Unet-like pure transformer for medical image segmentation", arXiv preprint arXiv:2105.05537, , May 2021.
-
X. Yan, H. Tang, S. Sun, H. Ma, D. Kong, and X. Xie, "AFTer-UNet: Axial Fusion Transformer UNet for Medical Image Segmentation", Proc of IEEE Winter Conf. on Applications of Computer Vision, Waikoloa, HI, USA, pp. 3270-3280, Jan. 2022.
[https://doi.org/10.1109/WACV51458.2022.00333]

- J. L. Ba, J. R. Kiros, and G. E. Hinton, "Layer normalization", arXiv preprint arXiv:1607.06450, , Jul. 2016.
- D. Ulyanov, A. Vedaldi, and V. Lempitsky, "Instance normalization: The missing ingredient for fast stylization", arXiv preprint arXiv:1607.08022, , Jul. 2016.
-
A. Carass, S. Roy, A. Jog, J. L. Cuzzocreo, E. Magrath, A. Gherman, J. Button, J. Nguyen, P. L. Bazin, P. A. Calabresi, C. M. Crainiceanu, L. M. Ellingsen, D. S. Reich, J. L. Prince, and D. L. Pham, "Longitudinal multiple sclerosis lesion segmentation data resource", Data in Brief, Vol. 12, pp. 346-350, Jun. 2017.
[https://doi.org/10.1016/j.dib.2017.04.004]

- Project-MONAI (2021) UNETR [Source code]. https://monai.io/research/unetr, .
-
F. Milletari, N. Navab, and S. A. Ahmadi, "V-net: Fully convolutional neural networks for volumetric medical image segmentation", Proc. of IEEE Int. Conf. on 3D Vision, Stanford, CA, USA, pp. 565-571, Oct. 2016.
[https://doi.org/10.1109/3DV.2016.79]

-
C. M. Kim, J. Y. Kim, H. S. Kim, S. J. Eom, and H. Y. Lee, "Multiple Sclerosis Lesion Detection in MRI Brain Image using 3D U-Net", Journal of KIIT, Vol. 19 No. 9, pp. 95-105, Sep. 2021.
[https://doi.org/10.14801/jkiit.2021.19.9.95]


2017년 3월 ~ 현재 : 금오공과대학교 컴퓨터소프트웨어공학과 학사과정
관심분야 : 이미지 처리, 딥러닝

1997년 : 성균관대학교 정보공학과(학사)
1999년 : KAIST 전산학과(공학석사)
2006년 : KAIST 전자전산학과(공학박사)
2008년 ~ 현재 : 금오공과대학교 컴퓨터소프트웨어공학과 교수
관심분야 : Digital Forensics, Image Processing, IoT