멀티오믹스 데이터를 활용한 알츠하이머 질병 예측 딥러닝 모델 개발 연구
초록
알츠하이머 질병(AD, Alzhemer’s Disease)은 치매를 일으키는 원인으로 뇌의 구조적 변화로 인해 인지능력이 저하되는 퇴행성 뇌질환이다. 인간에 있어서 치명적인 신경 퇴행성 질환 임에도 불구하고, AD에 대한 유전적 위험인자와 질병 기전은 아직 명확하게 밝혀지지 않고 있다. 따라서, AD 진단 및 예측은 치매 방지를 위한 필수적인 요소가 되고 있으며, 이로 인해, AD 예측을 위한 많은 전산학적 기반 모델들이 제안되고 있다. 본 논문에서는 멀티오믹스 데이터를 기반으로 하여 딥러닝을 활용한 AD 예측 모델을 제안하고자 한다. 유전자 발현 값을 기반으로 AD 특징 벡터를 추출하였고, 그래프 임베딩(Embedding) 기반의 Node2vec 알고리즘을 활용하여 질병 특징 벡터를 계산하였다. 추출된 특징 벡터들을 결합(Concatenate)하여 딥러닝 예측 모델의 입력값으로 사용하여 AD를 효과적으로 예측하였다. 결과적으로, 다양한 실험 결과에서 제안하는 모델의 성능이 우수한 것을 정량적으로 확인할 수 있었다.
Abstract
Alzheimer’s Disease(AD) is a type of common dementia that causes brain cells to die. However, genetic risk factor and disease mechanism of AD is not clearly revealed yet. Therefore, growing number of computational models have been proposed, which can serve as effective tool for the diagnosis and the detection of Alzheimier’s disease (AD). This paper proposes deep learning-based AD prediction model based on multi-omics dataset. We calculated AD feature vector based on gene expression data and disease feature vector based on Node2vec algorithm. Then, we concatenated each vector for the input of the deep learning-based prediction model. As a result, experimental results based on the various evaluation metrics clearly demonstrated the reliable performance of our model.
Keywords:
alzheimer disease, deep learning, computational biology, bioinforamaticsⅠ. 서 론
알츠하이머 질병(AD, Alzheimer’s Disease)은 인간의 치매를 일으키는 질병의 형태로서, 뇌의 구조적 변화로 인해 인지능력 저하와 행동 장애를 일으키는 퇴행성 뇌질환이다[1]-[3]. 이 질환은 세계의 건강, 공중 보건 및 인구 건강 측면에서 중요한 건강 문제로 대두되고 있으며, 이로 인해 AD 메커니즘에 대한 이해와 조기 진단은 유전체학(Genomics)에서 중요한 연구 과제로 남겨져 있다[4][5]. 최근 분야를 막론하고 인공지능 기술이 막강한 성능을 도출함에 따라, 인공지능을 기반으로 AD의 기전을 예측하기 위한 많은 연구가 진행되고 있다[6]-[10]. 이러한 관점에서 기계학습, 딥러닝을 기반으로 한 전산학적 모델들이 신경 영상(Neuroimaging), 유전체학(Genomics), 질병 진단 등 다양한 분야에서 주목할 만한 성과를 도출하고 있다[11]-[14]. AD 진단 및 예측 분야의 경우, 기계학습 기반 예측 모델의 목표는 신경 영상 및 게놈(Genome) 데이터를 효과적으로 활용하여 AD를 진단하는 데이터 기반 알고리즘을 설계하는 것이다. 최근 딥러닝 모델의 한 종류인 합성곱 신경망(CNN, Convolutional Neural Network)을 활용한 다양한 생물학적 연구들이 제안되고 있다. CNN은 이미지 처리, 영상 처리에서 주로 쓰이는 인공 신경망 모델이며, 뇌신경영상(Neuroimage)을 비롯한 여러 자가공명영상(MRI) 데이터를 효과적으로 처리하여 AD를 예측하는데 큰 성능을 보이고 있다[6][7]. 일반적으로 딥러닝 모델은 가공되지 않은(Raw) 데이터를 여러 계층적 표현으로 나타낸다. 이러한 특징으로 인해, 딥러닝은 여러 뇌 신경 영상과 유전체 데이터를 효율적으로 처리하여, AD를 진단하고 예측하는 연구 분야에서 유망한 기술로 손꼽히고 있다. 또한, 최근 컴퓨터 하드웨어와 소프트웨어 기술의 발달로 인해, 딥러닝 모델들의 성능은 더욱 증가하고 있으며, AD를 진단하고 예측하는 연구와 더불어 여러 생물정보학(Bioinforamtics) 문제를 다루는 데 있어서 중요한 핵심 기술로 자리매김을 하고 있다[15]-[18].
전 세계적으로 AD 환자의 수는 크게 증가하여, 공중 보건에 큰 위협이 되는 요소가 되어 가고 있다. 이에, AD의 요인을 설명할 수 있는 유전적 위험 요소를 밝혀내기 위한 여러 연구가 진행되어왔다. Desikan 외 GWAS(Genome- Wide Association Study) 데이터를 기반으로 AD 관련 단일 염기 다형성(SNP)을 밝혀내는 방법을 제안하였다[19]. 제안되는 모델에서는 AD에 대한 유전적 위험의 개인차를 연령대별로 정량화하였다. 하지만, GWAS를 통해 밝혀진 AD 관련 SNP만을 사용함에따라 예측 성능의 한계점을 보여주었다. Park 외 서로 다른 계층에 존재하는 오믹스 데이터를 통합하여 AD 특이적 유전자 네트워크를 구축하였고, 이를 통해 네트워크를 기반으로 한 오믹스 데이터에서 AD 예측이 가능함을 확인하였다[20]. 또한, AD를 효과적으로 분류하기 위해, 비침습적(Non-invasive) 영상 바이오마커를 제공하는 양성자-자기 공명(Proton-magnetic) 분과 기술이 활용되고 있다. Munteanu 외 다층 퍼셉트론(MLP, Multi-Layer Perceptron)을 사용하여 모델의 예측 성능을 향상한 모델을 제안하였다[21]. 하지만, 해당 논문에서는 유전자 발현량 같은 생물 분자층 간의 상호작용을 고려하지 않았다는 한계점을 지니고 있다.
본 논문에서는 오믹스(Omics) 데이터를 활용하여 딥러닝 모델 기반 AD 예측 모델을 제안하고자 한다. 기계학습 기반 모델의 성능을 높이는 가장 직관적인 방법은 각 개체를 수치형 데이터로 나타내는 표현 학습(Representation learning)을 적용하는 것이다. 이러한 관점에서, 본 논문에서는 AD의 상태를 표현할 수 있는 환자들의 유전자 발현 값을 벡터값으로 사용하였고, 질병 네트워크를 기반으로 그래프 임베딩(Graph embedding) 기법인 Node2vec 알고리즘을 적용하여 AD 벡터를 효과적으로 추출하였다. 오믹스 데이터로 구분되는 유전자 발현량 값을 사용함에 있어서 가장 큰 이점은 생물학적 메커니즘을 기계학습 모델에 적용할 수 있다는 것이다. 딥러닝 기반 그래프 임베딩 기법으로 추출된 벡터와 유전자 발현량 값을 하나의 벡터로 결합(Concatenate)하여 질병을 예측할 수 있는 딥러닝 예측 모델을 훈련하는 입력값으로 사용하였다.
본 논문의 구성은 다음과 같다. 1장에서는 AD에 대한 특성을 언급하고 관련 연구들을 소개한다. 2장에서는 딥러닝 기반 AD 예측 모델을 훈련하기 위한 데이터를 소개하고, 모델 아키텍처에 대하여 자세히 기술한다. 3장에서는 예측 모델의 성능을 측정하는 실험 결과에 대하여 설명 및 분석하고, 4장에서는 추후 연구 계획 및 한계점에 대해 언급한다.
Ⅱ. 본 론
2.1 데이터셋
본 논문에서는 유전자 발현량 값을 활용하여 생물 메커니즘을 기계학습 모델에 반영하였다. 두 개의 전두엽 피질(Prefontal cortex) 유전자 발현 데이터(GSE33000, GSE44770)를 통합하여 모델 훈련을 위한 충분한 데이터 샘플 사이즈를 확보하였다. 표 1과 같이 유전자 발현량 값 데이터는 257명의 정상 환자와 439명의 AD환자로 구성되어 있다. 확보된 유전자 발현 값을 토대로 Z 점수(Z-score)를 적용하여 정규화를 실행하였다.
2.2 질병 네트워크 구축
생물 정보학에서 질병 메커니즘을 정확하게 파악하기 위해, 많은 네트워크 기법들이 제안되어왔다[15]. 본 논문에서는 AD를 수치형 데이터로 정확하게 표현할 수 있는 그래프 임베딩(Graph embedding) 기법인 Node2vec을 적용하기 위해, 질병 네트워크를 구축하였다. 그래프 임베딩이란 그래프, 네트워크로 표현되는 여러 복합적 관계를 벡터 또는 벡터의 집합으로 변환하는 방법론이다. 네트워크 기반 분석 방법을 통해 우리는 다양한 관계에서 파생되는 여러 문제를 직관적으로 쉽게 파악할 수 있으며, 새로운 관계 또한 효과적으로 예측할 수 있다. 더 나아가서는 네트워크를 벡터공간에 표현하여 더 다양하고, 정확한 문제 해결 방법을 제시할 수 있다. 본 논문에서는 질병 네트워크를 구축하여 AD 벡터를 추출하고자 한다. 질병 네트워크란 각 노드(Node)는 질병으로 표현되고, 노드 사이에 이어진 에지(Edge) 정보는 질병 간의 유사성(Similarity) 정보로 표현된 네트워크이다. 이에 정확한 네트워크를 구축하기 위해서는 노드 사이의 관계를 표현하는 에지 정보, 즉 유사성 정보를 얼마나 효과적으로 계산하는지에 달려있다.
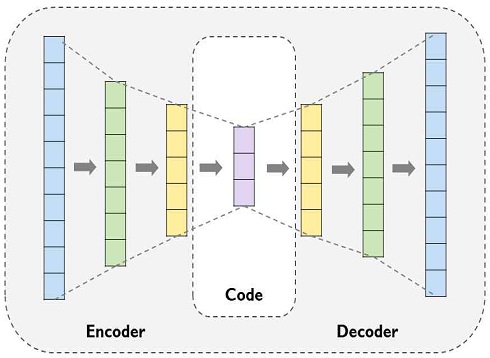
본 논문에서는 오토인코더를 적용하여 잠재 공간(Latent space)을 학습하고, 이를 가우시안 상호작용 프로필 커널(Gaussain interaction profile kernel)에 적용하여 질병 간의 유사성 정보를 계산하였다. 오토인코더(Auto-encoder)는 딥러닝을 활용한 차원 축소(Dimension reduction) 기법으로서 비지도 학습(Unsupervised learning)을 기반으로 모델을 훈련한다. 이는 입력 데이터값보다 적은 차원으로 축소를 시키고, 이를 다시 원본 데이터로 복원하도록 네트워크를 학습시키는 방법이다. 오토인코더는 크게 두 부분으로 구성되는데, 차원을 축소 시키는 인코더(Encoder) 부분과 이를 다시 원래의 데이터 값으로 복원하는 디코더(Decoder) 부분으로 나누어진다(그림 1).
먼저, 원-핫(One-hot) 벡터로 표현된 질병 벡터 x를 인코더를 통해 차원 축소를 진행하였다(식 (1)).
| (1) |
여기서 W는 인코더 부분의 가중치 값을 의미하며, s는 비선형(Non-linear)함수로 표현된다. 디코더 부분에서는 다시 디코딩 과정을 통해 저차원으로 표현된 d 벡터를 원래의 차원값으로 복원을 시킨다(식 (2)).
| (2) |
새롭게 복원된 x'를 통해, 오토인코더의 에러값을 줄이는 방향으로 모델을 학습하며, 가중치W,W',b,b' 값들을 수정해나간다(식 (3)).
| (3) |
오토인코더로 추출된 벡터를 기반으로 질병 i와 질병 j가 주어졌을 때, d(i), d(j)는 각각의 질병 벡터를 의미한다. 질병 i와 질병 j 간의 가우시안 커널 유사도 GS(d(i), d(j))는 다음 식을 통해 계산하였다(식 (4)).
| (4) |
여기서 GS는 가우시안 상호작용 프로필 커널(Gaussain interaction profile kernel)을 뜻하며, rd는 커널의 대역폭(Bandwidth)를 결정하는 하이퍼 파라미터(Hyper-parameter)로서 다음 식을 통해 계산하였다(식 (5)).
| (5) |
파라미터는 질병 당 상호작용의 수로 나누어 정규화를 수행하였다. 이를 통해, 커널 값은 데이터 크기에 영향을 받지 않도록 세팅하였다. 는 통상적으로 1로 설정되며, 본 논문에서도 해당 값을 사용하였다.
가우시안 상호적용 프로필 커널을 통해 질병 사이의 유사성 정보를 효과적으로 계산하였으며, 이를 질병 네트워크 에지(Edge) 정보로 활용하여 질병 네트워크를 구축하였다. 구축된 질병 네트워크를 통해 그래프 임베딩 기법인 Node2vec 알고리즘을 적용하여 각각의 질병 벡터를 효과적으로 추출하였다.
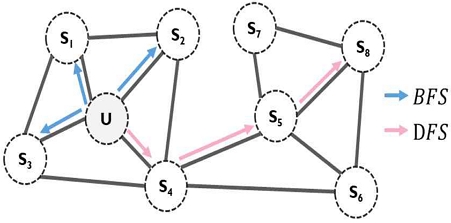
Node2vec 알고리즘은 그림 2와 같이 DFS(Depth-First-Sampling)와 BFS(Breadth-First-Sampling) 개념을 도입하여 기존 그래프 임베딩에 존재하였던 한계점을 극복한 알고리즘이다[22]. DFS는 랜덤워크(Random)를 수행할 때 시작 노드로 다시 되돌아올 확률을 작게 설정하여, 시작 노드로부터 최대한 멀리 떨어져 있는 노드까지 잘 반영할 수 있도록 학습하는 방법이다. 이에 반해, BFS는 시작 노드로 되돌아올 확률 값을 크게 설정하여, 최대한 지역적인 정보인 서브 그래프 구조를 표현하는 방법이다. Node2vec 알고리즘을 활용하여 질병 네트워크를 기반으로 정확한 질병 벡터를 계산하였다.
2.3 차원 축소 알고리즘
본 논문에서는 효과적으로 유전자 발현량 데이터를 처리하기 위해, 주성분 분석(PCA, Principal Component Analysis)을 적용하여 특성(Feature)들의 차원을 축소 시켰다. PCA는 특징 선택(Selection)의 대표적인 방법으로 패턴이 지니는 다수의 특징 중에서 유의미한 소수의 특징을 선별하는 기법이다.
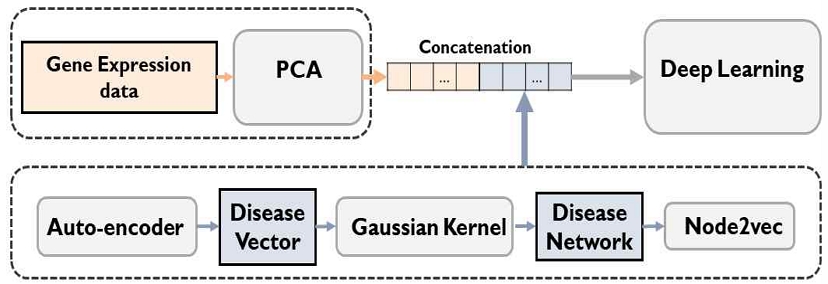
본 논문에서는 PCA를 적용하여, 고차원의 입력 데이터에서 유의미한 특징을 필터링(Filtering)하고, 이를 저차원으로 투영(Projection)하였다. PCA 기법을 통해 유의미한 특징을 추출함으로써 모델의 예측 성능을 높일 수 있도록 입력 데이터를 정제하였다. 최종적으로 PCA를 통해 정제된 유전자 발현 값 벡터 hg와 Node2vec을 통해 추출된 AD 벡터 hd를 결합(Concatenate)하여 하나의 벡터ht = [hm;hd]로 표현하였고, 이를 딥러닝 기반 AD 예측 모델의 입력 데이터값으로 사용하였다.
2.4 딥러닝 기반 AD 예측 모델
Node2vec 알고리즘을 통해 추출된 AD 벡터와 질병 벡터 정보를 결합하여 ht벡터를 생성하였고, 이를 통해 딥러닝 예측 모델을 효과적으로 훈련하는 입력 데이터값으로 사용하였다(그림3). 딥러닝 기술은 컴퓨터 비전(Computer vision), 자연어 처리(NLP, Natural Language Processing), 생물정보학 등 다양한 분야에서 막강한 성능을 자랑하는 인공지능 모델이다[9][10]. 이에 본 논문에서는 여러 개의 퍼셉트론 뉴런(MLP, Multi-Layer Perceptron)을 여러 층으로 쌓아 올린 다층신경망 구조를 적용하였다(식 (9)). 활성화 함수(Activation function)로는 ReLU를 사용하였으며, 훈련 방법으로는 드롭아웃(Dropout) 기법을 적용하였다.
식 (9)에서 wx, ax, 그리고 bx는 각각 x번째 레이어(Layer)의 가중치 행렬, 퍼셉트론 활성화 함수, 바이어스(Bias) 벡터를 의미하며, 는 AD 관련성을 나타내는 예측 변수를 의미한다.
| (9) |
Ⅲ. 실험 결과
3.1 비교 실험
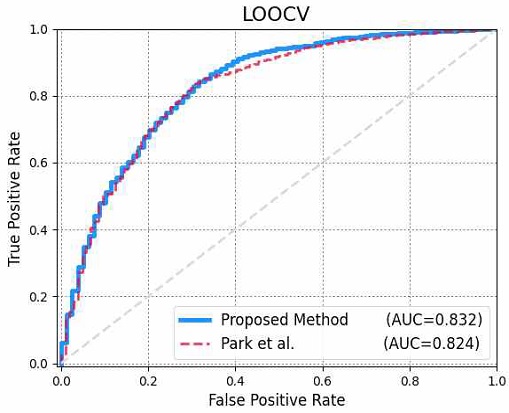
제안하는 예측 모델의 성능을 평가하기 위해 기존 논문과의 비교 실험을 실행하였다[6]. 본 논문에서는 모델의 성능을 정확하게 평가하기 위해, LOOCV(Leave-One-Out Cross-Validation) 검증 방법을 채택하였다. LOOCV는 n개의 데이터 샘플이 존재할 때, n-1개의 데이터 샘플을 무작위적으로 선택하여 이를 훈련데이터로 사용하고, 나머지 한 개의 데이터를 검증데이터로 사용하는 방법이다. 이를 n회 반복하고, 평균을 계산하여 무작위성(Randomness)의 바이어스를 고려하였다. 성능평가 지표로는 AUC(Area Under the receiver operating characteristic Curve)를 채택하였다.
ROC 커브의 아래 면적을 의미하는 AUC 평가 지표는 일반적으로 분류 및 예측 모델의 성능을 측정할 때, 널리 사용된다. AUC 값이 1에 가까울수록, 분류 모델의 성능은 뛰어나다는 것을 의미하며, 0.5는 모델의 분류 성능은 거의 없다는 것을 뜻한다. 즉, 본 논문에서 제안하는 모델이 AD를 정확히 예측할수록, AUC 값은 1에 가깝게 나오게 된다.
그림 4에서 확인할 수 있듯이, 본 논문에서 제안하는 예측 모델의 성능이 (AUC=0.832) AUC 측면에서 기존 모델보다 (AUC=0.824) 성능이 더 뛰어난 것을 확인할 수 있었다. 이는 곧 본 논문에서 제안하는 딥러닝 기반 예측 모델이 AD를 예측하는 데 있어서 효과적이라는 것을 증명한다.
3.2 Node2vec 알고리즘 성능 평가
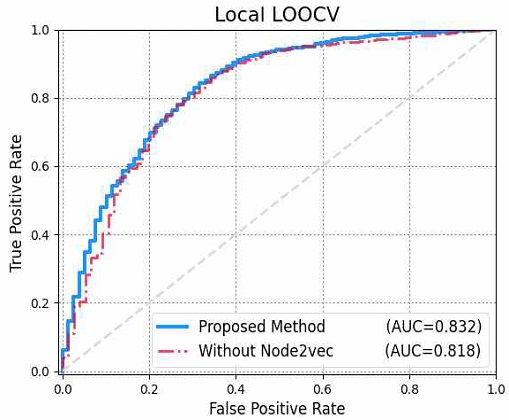
AD 벡터를 정확하게 표현하기 위해, 본 논문에서는 그래프 임베딩 기반 Node2vec을 적용하였다. 정확한 벡터 표현 기법을 통해 추출된 벡터는 예측 모델을 정확하게 훈련하는 데 있어서 중요한 입력 데이터가 된다. 본 실험에서는 Node2vec을 통해 추출한 질병 벡터를 유전자 발현 값과 같이 사용하였을 경우와 유전자 발현 값만 사용하였을 경우로 나누어 비교 실험을 진행하였다.
그림 5에서 확인할 수 있듯이, 유전자 발현 값으로만 입력 데이터를 사용하였을 때보다(AUC=0.818), Node2vec을 적용하여 추출한 질병 벡터를 입력 값으로 함께 사용하였을 때(AUC=0.832), 예측 모델의 성능이 AUC 측면에서 더 나은 것을 확인할 수 있었다. 이는 곧, 그래프 임베딩 기법인 Node2vec 알고리즘이 네트워크 특성을 반영하여 질병 벡터를 수치형 데이터로 정확하게 표현하였다는 것을 증명한다.
Ⅳ. 결론 및 향후 과제
AD는 인간의 치매를 일으키는 하나의 형태로서, 뇌의 구조적 변화로 인해 인지능력 저하와 행동 장애를 일으키는 퇴행성 뇌질환이다. 인간의 기대 수명이 늘어남에 따라, AD의 조기 진단은 인간의 삶에 있어서 중요한 요소가 되어가고 있으며, 많은 연구를 통해 이를 예측하기 위한 다양한 전산 모델들이 제안되고 있다. 최근 인공지능(AI)의 기술이 급격히 발전함에 따라, 다양한 분야에서 이를 통하여 막강한 성능 향상을 도출하고 있다. 이에 본 논문에서는 대표적인 인공지능 기법인 딥러닝 기술을 활용하여 AD 예측 모델을 제안하였다. 먼저, 효과적으로 딥러닝 모델을 훈련하기 위해 그래프 임베딩 기법을 적용하여 입력 데이터 전처리 과정을 수행하였다. 그래프 임베딩의 가장 일반적인 기본 가정은 각 노드 벡터가 인접한 네트워크의 구조를 보존해야 한다는 것이다. 그래프 임베딩 기법인 Node2vec을 활용하여 질병 네트워크 속성을 반영한 질병 벡터를 추출하였다. 또한, 유전자 발현 값을 기반으로 주성분 분석(PCA, Principal Component Analysis)을 적용하여 특성(Feature)들의 차원을 축소 시켰다. 이를 통해, 고차원의 입력 데이터에서 유의미한 특징을 필터링(Filtering)하고, 유의미한 입력값을 추출하는 과정을 수행하였다. 네트워크 속성을 반영한 질병 벡터와 PCA 기법을 수행한 유전체 벡터를 결합(Concatenate)하여 딥러닝 모델을 훈련하는 입력값으로 사용하였다. 정확한 입력 벡터값을 통해서 모델을 효과적으로 훈련하였으며, 이를 통해 AD를 정확하게 예측할 수 있는 모델의 기반을 만들었다. 결과적으로 다양한 비교 실험을 통해 모델의 성능을 평가하였으며, 이를 통해, 제안된 AD 예측 모델의 우수성을 증빙할 수 있었다. 추후 연구에서는 더 정교한 인공지능 기법을 적용하여 질병 벡터값을 추출하고자 하며, 질병의 특성을 정확하게 반영할 수 있는 인공지능 모델을 제안하고자 한다.
Acknowledgments
이 논문은 2022년 부경대학교 국립대학육성사업 지원비에 의하여 연구되었음.
이 성과는 정부(과학기술정보통신부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임(2022R1G1A1003616)
References
-
A. Kumar and A. Singh, "A review on Alzheimer’s disease pathophysiology and its management: An update", Pharmacol. Rep. Vol. 67, No. 2, pp. 195-203, Sep. 2015.
[https://doi.org/10.1016/j.pharep.2014.09.004]

-
K. S. Vetrivel and G. Thinakaran, "Amyloidogenic processing of betaamyloid precursor protein in intracellular compartments", Neurology, Vol. 66, No. 2, pp. 69-73, Dec. 2005.
[https://doi.org/10.1212/01.wnl.0000192107.17175.39]

-
V. M. Lee and J. Q. Trojanowski, "Mechanisms of parkinson's disease linked to pathological alpha-synuclein: New targets for drug discovery", Neuron, Vol. 52, No. 1, pp. 33-38, Oct. 2006.
[https://doi.org/10.1016/j.neuron.2006.09.026]

-
M. R. Roberson, K. Kolasa, D. S. Parsons, and L. E. Harrell, "Cholinergic denervation and sympathetic ingrowth result in persistent changes in hippocampal muscarinic receptors", Neuroscience, Vol. 80, No. 2, pp. 413-418, Jul. 1997.
[https://doi.org/10.1016/S0306-4522(97)00153-X]

-
E. Lin, C. H. Lin, and H. Y. Lane, "Deep Learning with Neuroimaging and Genomics in Alzheimer’s Disease", Int. J. Mol. Sci., Vol. 22, No. 15, pp. 7911, Jul. 2021.
[https://doi.org/10.3390/ijms22157911]

-
J. Ha, C. Park, and S. Park, "Prediction of Alzheimer's disease based on deep neural network by integrating gene expression and DNA methylation dataset", Expert Syst Appl, Vol. 140, pp. 112873, Fed. 2020.
[https://doi.org/10.1016/j.eswa.2019.112873]

-
H. Nawaz, M. Maqsood, S. fzal, F. adil, I. Mehmood, and S. Rho, "A deep feature-based real-time system for Alzheimer disease stage detection", Multimedia Tools and Applications, Vol. 80, pp. 35789-35807, Jun. 2020.
[https://doi.org/10.1007/s11042-020-09087-y]

-
M. Raza, M. Awais, W. Ellahi, N. Aslam, H. Nguyen, and H. Le-Minh, "Diagnosis and monitoring of alzheimer’s patients using classical and deep learning techniques", Expert Syst. Appl., Vol. 136, pp. 353-364, Dec. 2019.
[https://doi.org/10.1016/j.eswa.2019.06.038]

-
S. Basheer, S. Bhatia, and S. B. Sakri, "Computational Modeling of Dementia Prediction Using Deep Neural Network: Analysis on OASIS Dataset", IEEE Access, Vol. 9, pp. 42449-42462, Mar. 2021.
[https://doi.org/10.1109/ACCESS.2021.3066213]

-
S. Kaur, S. Gupta, S. Singh, and I. Gupta, "Detection of Alzheimer’s Disease Using Deep Convolutional Neural Network", International Journal of Image and Graphics, Vol. 22, No. 03, pp. 2140012, Jan. 2021.
[https://doi.org/10.1142/S021946782140012X]

-
J. Ha and C. Park, "MLMD: Metric Learning for Predicting MiRNA-Disease Associations", IEEE Access, Vol. 9, pp. 78847-78858, May 2021.
[https://doi.org/10.1109/ACCESS.2021.3084148]

-
M. Tanveer, B. Richhariya, R. Khan, A. Rashid, P. Khanna, M. Prasad, and C. Lin, "Machine learning techniques for the diagnosis of Alzheimer’s disease: A review", ACM Trans. Multimed. Comput. Commun. Appl. (TOMM), Vol. 16, No. 30, pp. 1-35, Apr. 2020.
[https://doi.org/10.1145/3344998]

-
J. Ha, C. Park, C. Park, and S. Park, "IMIPMF: Inferring miRNA-disease interactions using probabilistic matrix factorization", Journal of Biomedical Informatics, Vol. 102, pp. 103358, Feb. 2020.
[https://doi.org/10.1016/j.jbi.2019.103358]

-
P. Khan, M. F. Kader, S. R. Islam, A. B. Rahman, M. S. Kamal, M. U. Toha, and K. S. Kwak, "Machine Learning and Deep Learning Approaches for Brain Disease Diagnosis: Principles and Recent Advances", IEEE Access, Vol. 9, pp. 37622-37655, Feb. 2021.
[https://doi.org/10.1109/ACCESS.2021.3062484]

-
J. Ha, C. Park, C. Park, and S. Park, "Improved prediction of miRNA-disease associations based on matrix completion with network regularization", Cells, Vol. 9, No. 4, pp. 881, Apr. 2020.
[https://doi.org/10.3390/cells9040881]

-
S. Mørk, S. Pletscher-Frankild, A. Palleja Caro, J. Gorodkin, and L. J. Jensen, "Protein-driven inference of miRNA-disease associations", Bioinformatics, Vol. 30, No. 3, pp. 392-397, Nov. 2013.
[https://doi.org/10.1093/bioinformatics/btt677]

-
J. Ha, C. Park, and S. Park, "PMAMCA: prediction of microRNA-disease association utilizing a matrix completion approach", BMC Systems Biology, Vol. 13, No. 1, pp. 1-13, Mar. 2019.
[https://doi.org/10.1186/s12918-019-0700-4]

-
J. Ha, "MDMF: Predicting miRNA–Disease Association Based on Matrix Factorization with Disease Similarity Constraint", Journal of Personalized Medicine, Vol. 12, No. 6, pp. 885, 2022.
[https://doi.org/10.3390/jpm12060885]

-
R. S. Desikan, C. C. Fan, Y. Wang, A. J. Schork, H. J. Cabral, and L. A. Cupples, "Genetic assessment of age-associated Alzheimer disease risk: Development and validation of a polygenic hazard score", PLoS medicine, Vol. 14, pp. e1002258, Mar. 2017.
[https://doi.org/10.1371/journal.pmed.1002289]

-
C. Park, Y. Yoon, O. Min, S. J. Yu, and J. Ahn, "Systematic identification of differential gene network to elucidate Alzheimer’s disease", Expert Syst Appl, Vol. 85, pp. 249-260, Nov. 2017.
[https://doi.org/10.1016/j.eswa.2017.05.042]

-
C. R. Munteanu, C. Fernandez-Lozano, V. M. Abad, S. P. Fernández, J. Álvarez-Linera, and J. A. Hernández-Tamames, "Classification of mild cognitive impairment and Alzheimer’s Disease with machine-learning techniques using 1H Magnetic Resonance Spectroscopy data", Expert Systems with Applications, Vol. 42, No. 15-16, pp. 6205-6214, Sep. 2015.
[https://doi.org/10.1016/j.eswa.2015.03.011]

-
A. Grover and J. Leskovec, "Node2vec: Scalable feature learning for networks", in In Proceedings of the 22nd ACM SIGKDD international conference on Knowledge discovery and data mining, pp. 855-864, Aug. 2016.
[https://doi.org/10.1145/2939672.2939754]


2013년 8월 : 부산대학교 바이오정보전자공학과(공학사)
2015년 8월 : 연세대학교 컴퓨터과학과(공학석사)
2020년 8월 : 연세대학교 컴퓨터과학과(공학박사)
2020년 ~ 2021년 : 하와이 암센터 포스닥 연구원
2021년 ~ 현재 : 부경대학교 데이터정보과학부 빅데이터융합전공 조교수
관심분야 : 기계학습, 생물정보학, 데이터마이닝, 딥러닝

2015년 2월 : 서강대학교 전자공학과(공학사)
2017년 2월 : 포항공과대학교 전자전기공학과(공학석사)
2020년 8월 : 포항공과대학교 전자전기공학과(공학박사)
2021년 8월 : 포항공과대학교 전자전기공학과 박사후연구원
2021년 ~ 현재 : 부경대학교 미디어커뮤니케이션 학부 휴먼ICT융합전공 조교수
관심분야 : 멀티미디어 영상신호처리, 컴퓨터 비전, 딥러닝 시스템 설계

2012년 12월 : 퍼듀대학교 산업공학과(공학사)
2020년 8월 : 서울대학교 산업공학과(공학박사)
2020년 9월 ~ 2021년 8월 : 삼성전자 무선사업부 Core UX그룹 Senior Designer
2021년 9월 ~ 현재 : 부경대학교 미디어커뮤니케이션학부 휴먼ICT융합전공 조교수
관심분야 : Human-Computer Interaction, Ergonomic Design, User Experience