뇌 MRI 신호 감쇠 모델링을 통해 일반화된 QQ 모델의 클러스터링 기반 OEF 계산 알고리즘
초록
뇌 MRI 신호에 QQ 모델을 통해 혈액 내 산소추출률(OEF)을 계산하여 뇌 질환을 진단할 수 있다. 기존 QQ 모델 기반의 OEF 계산 방법은 MRI 촬영 설정에 민감하다. 본 논문은 MRI 신호 감쇠 모델링을 통해 QQ 모델의 출력값을 일반화하고 클러스터링과 정합을 통해 OEF를 계산하는 방법을 제안한다. QQ 모델에 실례 입력값을 적용하여 신호 감쇠값을 계산하고, 제안한 모델로 파라미터값을 추정한다. 그 후 입력값, 파라미터값 및 자화율의 클러스터링으로 대표 클러스터를 계산한다. 미지의 MRI 신호에 대해 제안한 모델로 추정하고 대표 클러스터와 정합과 보간으로 입력값을 추정해 OEF를 계산한다. 미국 코넬의대 17명 환자 데이터를 이용하여 제안한 방법을 기존 CAT 방법과 비교하였고, 5.67 RMSE를 달성하여 7.15 RMSE의 CAT 방법보다 우수함을 보였다. 또한 제안한 방법이 MRI 촬영 설정 변경에도 안정적이고 높은 정확도로 동작하는 것을 보였다.
Abstract
Calculating oxygen extraction rate(OEF) in blood through QQ model for brain MRI signal can be used for brain disease diagnosis. Previous QQ model-based OEF calculation methods are sensitive to MRI imaging settings. This paper proposes a method to generalize the output value of QQ model through MRI signal decay modeling and calculate OEF with clustering and matching. Signal decay is calculated by applying real input values to QQ model, and parameter values are estimated with the proposed model. Representative clusters are calculated through clustering with input, parameter, and susceptibility values. After estimating the proposed model for an unknown MRI signal, the input value is estimated through cluster matching and interpolation, and OEF is calculated. Using 17 patients data from Weill Cornell Medicine in USA, the proposed method was compared with the previous CAT method, and 5.67 RMSE was achieved, which was superior to the CAT method of 7.15 RMSE. In addition, it was shown that the proposed method performs reliably and with high accuracy even when MRI imaging settings are changed.
Keywords:
oxygen extraction fraction, brain MRI, signal decay modeling, clustering, QQ modelⅠ. 서 론
뇌경색, 뇌종양, 뇌출혈 등 뇌 질환은 현대 사회에서 많이 발생하고 높은 사망률의 원인이다. 이들 질환의 예방 및 진단을 위해 다양한 검사 방법 중 인체에 미치는 영향이 적은 자기공명영상(MRI, Magnetic Resonance Imaging)이 많이 활용된다[1][2].
뇌 질환의 진단을 위해 조직의 생존력이나 기능 분석이 필요하며, 각 조직에서 혈액 내의 산소를 소모한 정도인 산소추출률(OEF, Oxygen Extraction Fraction)을 계산하여 정상적 활동을 분석할 수 있다. 이와 같은 OEF 계산을 위하여 연구들이 진행되었지만, 특정한 조건에서만 계산이 올바르게 수행되는 문제가 있고, 특정한 조건 제약을 약화한 QQ 모델이 유용하지만 MRI 신호에 포함된 잡음에 대한 민감한 문제가 있다[3][4]. 또한, QQ 모델을 기반으로 MRI 신호 잡음에도 불구하고 안정적으로 OEF 계산이 가능한 방법도 있지만 Echo Time(TE)과 같은 MRI 촬영 설정에 의존적인 문제가 있다[4][5].
본 논문에서는 뇌 MRI 신호 감쇠 모델링을 통하여 QQ 모델의 출력값을 표현하고 클러스터링과 정합을 통해 OEF를 계산하는 방법을 제안한다. 특히, 제안한 모델을 이용하여 TE와 같은 MRI 촬영 설정에 의존적인 문제를 해결하였고, 안정적이고 높은 정확도로 OEF를 계산할 수 있음을 보였다.
본 논문은 다음과 같이 구성되어 있다. 2절에서 관련 연구를 요약하고, 3절에서 MRI 신호 감쇠 모델링 기반 클러스터링과 정합 방법을 제안한다. 실험 결과를 4절에서 제시하고 5절에서 결론 짓는다.
Ⅱ. 관련 연구
뇌 MRI로 혈액에서 산소를 방출하는 디옥시헤모글로빈 효과의 연구들이 진행되었다. QUIXOTIC, Calibrated fMRI, qBOLD 같은 세기 신호 모델링 기반 방법과 뇌 전체 CMRO2나 복셀 단위 Quantitative Susceptibility Mapping(QSM)을 이용한 CMRO2 값을 통해 위상 신호 모델링 기반 방법들이 있다[5].
OEF 계산을 위해 Bolar et. al의 QUIXOTIC (Quantitative imaging of extraction of oxygen and tissue consumption) 방법[6], Gauthieret and Hoge의 fMRI를 이용하여 hypercapnia 및 hyperoxia를 위한 generalized calibration model을 이용한 방법[7], Wise et. al의 fMRI를 기반으로 interleaved 및 combined hypercapnia 및 hyperoxia를 이용한 방법[8], He and Yablonskiy의 quantitative BOLD(qBOLD) 방법[9]이 있다. 그러나 이 방법들은 특정한 가정에서 OEF 계산을 수행하기 때문에, 가정이 다른 경우 계산 결과가 올바르지 않은 문제가 있다.
특정한 가정의 제약을 완화하기 위하여, Cho et. al는 QSM과 qBOLD 모델을 결합한 QQ 모델을 제안하여 OEF를 계산하였다[3]. 이 모델은 임상에서 가정하기 어려운 혈관에서의 문제들을 고려할 필요 없이 OEF 계산이 가능하지만, QQ 모델에 포함된 qBOLD 역변환이 잡음에 민감한 문제가 있다.
2.1 QQ 모델
QQ 모델은 식 (1)과 같이 정의되며 QSM 모델과 qBOLD 모델을 결합함으로써 각 모델에서 필요한 파라미터를 상호 보완하여 OEF 계산에서 특정 가정이 필요하지 않은 장점이 있다[4].
| (1) |
여기서 Y는 정맥 산소포화도, v는 정맥 혈액량, R2는 BOLD 효과가 없는 조직의 가로이완, S0는 TE0에서 신호세기, xnb는 비혈류 물질 자화율, w는 QSM 가중치 및 λ는 정규화 가중치이다.
QSM 모델은 정맥 혈액의 디옥시헤모글로빈 영향과 비혈액 조직의 자화율을 구분할 수 있고, qBOLD 모델은 MRI multi-echo gradient echo(mGRE) 시퀀스 영상의 세기를 이용하여 OEF 효과를 모델링한다. 과 의 계산 방법은 Cho et. al과 Lee et. al에서 제시되어 있다[4][5].
QQ 모델은 이론적으로 mGRE 시퀀스의 MRI 신호 크기와 위상을 이용해 계산할 수 있지만, Y와 v의 연관성, qBOLD 역변환에서 잡음 민감성으로 인하여 실제 계산이 쉽지 않고, 정확성이 낮다.
2.2 CAT 방법
QQ 모델의 잡음 민감성을 개선하기 위해 Cho et al은 CAT(Cluster Analysis of Time evolution) 방법을 통해 OEF를 계산하였다[4]. mGRE 시퀀스에서 유사한 신호 감쇠를 하는 복셀들을 같은 모델링 파라미터값을 갖는 클러스터로 가정하였다. 그 후에 각 클러스터의 복셀들에 대해 평균화하고 파라미터를 추정하여 SNR을 증가시키고 견고하게 OEF를 계산하였다. 그러나 QQ 모델이 그래디언트 기반 반복적 문제 해결 과정에서 초기 추정값 설정과 파라미터 스케일링 등의 최적화를 위한 다양한 요소들에 영향받는 문제가 있다.
2.3 Lee et. al 방법
Lee et. al은 QQ 모델의 입력값에 대한 출력값의 쌍들에 대하여 클러스터 기반 분류를 수행하여 대표 클러스터 데이터베이스를 생성하고, 미지의 mGRE 시퀀스 출력값에 대하여 클러스터 데이터베이스의 유사한 클러스터에 대한 정합과 보간을 통하여 안정적으로 OEF를 계산하는 방법을 제안하였다[5]. 그러나 이 방법은 고정된 TE에서의 출력값에 대해서만 적용 가능한 문제가 있다.
Ⅲ. 제안하는 신호 감쇠 모델링을 이용한 클러스터링 기반 OEF 계산 방법
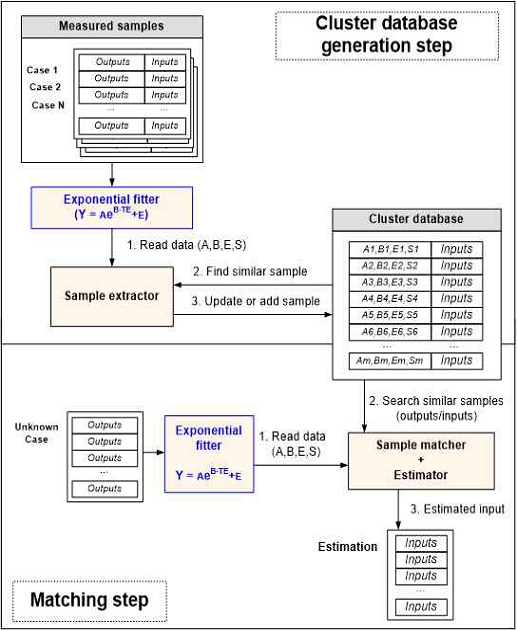
기존 방법의 문제점을 해결하고 안정적인 OEF 계산을 위하여, 본 절에서는 MRI 신호 감쇠 모델링을 통해 QQ 모델의 출력값을 표현하고 클러스터링과 정합을 통해 OEF를 계산하는 방법을 설명한다. 제안하는 방법의 블록 다이어그램은 그림 1과 같으며, 대표 클러스터 데이터베이스를 생성하는 단계와 미지의 출력값에 대하여 정합하고 입력값을 추정하는 2단계로 구성되어 있다.
대표 클러스터 데이터베이스를 생성하는 단계에서는 다양한 샘플들로부터 제안하는 신호 감쇠 모델링을 통하여 파라미터들을 추정한 후에 대표 클러스터 데이터베이스의 클러스터들과 비교하여 등록하거나 갱신하는 처리를 수행한다.
미지의 출력값에 대하여 대표 클러스터 데이터베이스와 정합하여 입력값을 추정하는 단계에서는 미시의 신호에 대하여 제안하는 신호 감쇠 모델링을 통하여 파라미터들을 추정하여 대표 클러스터 데이터베이스와 정합하여 유사한 클러스터를 찾아내고 보간을 통하여 OEF를 계산하는 처리를 수행한다.
Lee et. al 방법과 유사한 구조를 갖지만 MRI 신호 감쇠 추정 모델을 도입하였고, 그로 인해 클러스터 데이터베이스의 원소값들 구조가 다르다.
3.1 MRI 신호 감쇠 모델링
MRI는 강한 자기장 안에서 고주파로 수소 원자핵을 공명시킨 후에, 수소 원자핵이 안정적인 상태로 변화하며 발생하는 신호 에너지를 측정하여 획득된다. mGRE 시퀀스의 시간에 따른 측정 에너지값 즉 출력값은 잡음에 영향을 받으며, 동일한 입력에 대해 TE 설정에 따라 출력값이 다르다. 따라서 Lee et. al 방법의 경우 특정 TE에서의 출력값에 의존적으로 동작하는 문제가 있다.
mGRE 시퀀스 출력값의 잡음 영향과 다양한 TE 설정에 대한 출력값의 차이를 극복하기 위하여, 제안하는 방법에 대하여 식 (2)와 같이 출력값의 변화를 지수 함수 형태로 모델링하였고 A, B, E의 3개 파라미터로 표현을 하였다. A는 제안한 모델에서 지수 함수의 계수이며, B는 지수 가중치이다.
| (2) |
E는 식 (3)과 같이 측정된 mGRE 시퀀스 출력값 M과 지수 함수 모델링 값과의 RMSE이다.
| (3) |
이와 같이 MRI 신호 감쇠에 대한 모델을 통하여 다양한 출력값에 대하여 (A, B, E)의 간소화된 3개 파라미터로 표현할 수 있다.
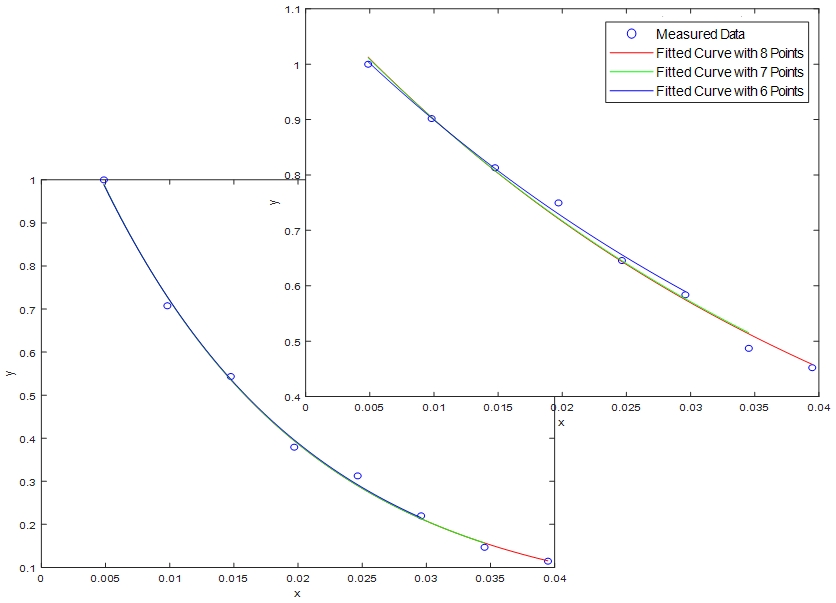
그림 2에는 측정된 8개 신호에 대하여 8개 측정값, 7개 측정값, 6개 측정값을 사용하여 제안한 모델의 파라미터를 추정하여 도시한 그래프이다. 사용한 측정값의 개수가 다르지만, 그래프가 유사한 것을 확인할 수 있다.
3.2 대표 클러스터 데이터베이스 생성 단계
대표 클러스터 데이터베이스는 실례 입력값들의 범위로 제한하여 QQ 모델로 생성한 출력 데이터에 대해, MRI 신호 감쇠 모델링(Exponential fitter)으로 파라미터를 추정하여, 입력값 및 추정 파라미터값과 자화율 쌍에 대해 클러스터링으로 생성한다. 이 쌍은 입력값(Y, v, R2, S0, xnb)과 QQ 모델의 출력값을 이용한 모델링 파라미터(A, B, E)와 자화율(S)로 구성된다. QQ 모델의 출력값은 각 TE에서 MRI 신호값과 자화율이다. 특히, 실제로 측정된 MRI 신호값들은 잡음이 필연적으로 포함되어 있으므로, 출력값에 대해 SNR 100 수준의 가우시안 잡음을 추가하여 QQ 모델 출력 데이터의 현실성을 높였다.
샘플 추출기(Sample extractor)에서는 새로운 샘플 쌍(Y, v, R2, S0, xnb, A, B, E, S)에 대하여 대표 클러스터 데이터베이스에 유사한 샘플이 존재하면, 해당 샘플에 새로운 샘플값을 추가하여 갱신하거나, 유사한 샘플이 없으면 샘플을 추가하여 클러스터 데이터베이스를 생성한다.
샘플 유사성을 결정하기 위해서는 새로운 샘플 데이터와 클러스터 데이터베이스에 포함된 샘플들의 파라미터(A, B, E)와 자화율(S)에 대해 식 (4)의 가중치 L2-norm을 이용하였고, 유사도가 지정한 임계치 이하면 유사한 것으로 판정하였다.
| (4) |
여기서 w는 가중치, Ro는 클러스터 데이터베이스에 포함된 샘플의 파라미터값, No는 새로운 샘플의 파라미터값이며, i는 값의 위치를 의미한다.
클러스터 데이터베이스에 포함되는 샘플 수는 유사도 판정의 임계치 값에 의하여 크게 좌우된다.
3.3 미지의 샘플 정합 및 OEF 계산 단계
미지의 샘플 정합 단계 및 OEF 계산 단계에서는 임의의 TE에서 측정한 MRI 신호를 이용하여 MRI 신호 감쇠 모델링을 통하여 파라미터를 추정하고 클러스터 정합을 통해 유사 샘플을 찾아 입력값(Y, v, R2, S0, xnb)를 추정한 후 OEF를 계산한다.
입력 MRI 신호에 대해 MRI 신호 감쇠 모델링을 통해 파라미터를 추정한 후에, 샘플 정합기(Sample matcher)는 해당 파라미터와 자화율을 클러스터 데이터베이스의 샘플들과 비교하여 가장 유사한 샘플을 검색한다. 이 과정에서 유사도는 3.2절의 식 (4)와 동일한 가중치 L2-norm을 활용하였다.
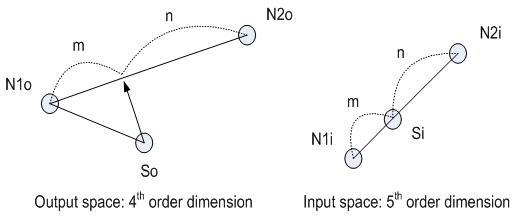
샘플 정합에서 정합된 샘플의 입력값을 그대로 사용할 수 있지만, 실제 측정된 값과 정합된 샘플의 값이 차이가 있다. 이 차이를 보상하기 위해 샘플 정합 과정에서 그림 3과 같이 2개의 유사한 샘플(N1, N2)를 검출하고 두 샘플값(N1o, N2o)로 추정한 벡터(So)에 대해 양방향 보간으로 m:n 비율을 계산하고, 입력값(N1i, N2i)에 대해서 해당 비율에 따라 보간하여 좀 더 정확히 입력값(Si)을 추정하였다.
입력값 추정으로 Y, v, R2, S0 및 xnb을 얻을 수 있고, OEF는 OEF = 1 – Y/Ya로 계산한다. Y는 정맥 산소포화도이고, Ya는 동맥 산소화값으로 0.98이다. v, R2, S0 및 xnb도 임상에서 질환 분석에 활용되지만, 본 연구는 OEF 계산만 초점을 두었다.
Ⅳ. 실험 결과 및 분석
4.1 데이터 획득 및 처리
실험은 미국 코넬의대의 CAT 방법과 Lee et. al 방법에서 사용한 데이터를 활용하였다[4][5]. 이 데이터는 32채널 수신기 코일로 3T GE 스캐너에서 3D 동맥스핀 라벨링 및 다중에코 그레디언트 에코, 확산강조영상 기법으로 편측 대뇌 동맥 영역에 병변이 위치한 허혈성 뇌졸중 환자를 촬영한 것이다.
3D ASL 시퀀스 파라미터, 3D mGRE 시퀀스 파라미터, DWI 시퀀스 파라미터는 CAT 방법과 Lee et. al 방법에서 사용한 것과 동일하다. 또한, 촬영한 신호에 대해 QSM 재구성을 적용하였다.
4.2 CAT 방법과 비교 분석
제안하는 방법은 유사성 판단에 가중치 L2-norm을 사용하여 가중치 w 및 유사도 판단 임계치 등 설정이 필요하며, 클러스터 데이터베이스 생성에 샘플 데이터도 필요하다. 따라서, 다양한 실험과 분석으로 가중치, 임계치 및 샘플 데이터를 설정하였다.
샘플들은 입력값 외에 3개 모델 파라미터(A, B, E)와 자화율(S)로 구성되고 가중치 w를 (5.0, 1.0, 5.0, 20)로 설정하였고, 임계치는 가중치 L2-norm 값이 0.04보다 작은 경우 유사한 것으로 판단하였다.
표 1은 CAT 방법과 제안한 방법으로 데이터 중 3개, 4개, 6개 데이터를 선택해 클러스터 데이터베이스를 생성하고, 다른 데이터에 적용해 OEF를 계산하여 RMSE(Root Mean Square Error)를 분석한 결과이다. 실제 MRI 측정값에는 잡음이 필연적으로 포함되기에 SNR=100 가우시안 잡음을 추가한 데이터를 활용하여 실험하였다.
표에는 17명 데이터의 뇌 영역 샘플값들의 개수도 제시되었고, 환자마다 뇌 크기가 달라 차이가 있지만 약 140~220만개 샘플이 존재한다. 또한, 3개, 4개, 6개 데이터를 선택하여 학습에 제안한 방법을 적용했을 때 클러스터 데이터베이스에 저장된 샘플은 4,024개, 4,546개, 4,892개로 유사한 신호값을 갖는 데이터들이 평균화되어서 많이 축소되었다. 또한, 사용하는 환자 데이터의 수를 늘려도 샘플수가 급격히 증가하지 않는다.
3개, 4개, 6개의 환자 데이터로 학습한 후에 계산한 OEF의 평균 RMSE는 각각 6.00, 5.86, 5.67이며, 클러스터 데이터베이스 생성에 사용한 것을 제외하면 6.13, 5.89, 5.35의 RMSE를 보였다. 클러스터 데이터베이스 생성에 사용하는 데이터 수가 증가하면 정확도가 높아지는 것을 알 수 있다. 또한 CAT 방법보다 우수한 정확도를 보이는 것을 알 수 있다.
4.3 TE 변화에 따른 성능 분석
제안하는 방법은 MRI 신호 감쇠 모델을 활용하여 TE 변화에도 불구하고 강인하게 OEF를 계산할 수 있다. 이를 위하여 TE 간격 및 샘플수를 변화하고 잡음을 추가한 신호들에 대하여 성능을 분석하였다. 표 2에는 실험에 사용한 다양한 TE 간격과 샘플수를 정리하였다.
표 2의 다양한 모델로 SNR=100 잡음을 고려해 생성한 MRI 신호에 대해 제안한 방법의 성능을 표 3에 정리하였다. 이때 4.2절 6개 환자 데이터를 이용해 생성한 클러스터 데이터베이스를 활용하였다.
클러스터 데이터베이스 생성에 활용된 것과 다른 TE와 샘플수를 활용하였음에도 불구하고 OEF에 대한 RMSE가 크게 차이가 없는 것을 확인할 수 있고, 이를 통하여 MRI 신호 감쇠 모델링 기법을 통하여 파라미터 추정을 활용하는 제안하는 방법이 효율적인 것을 알 수 있다.
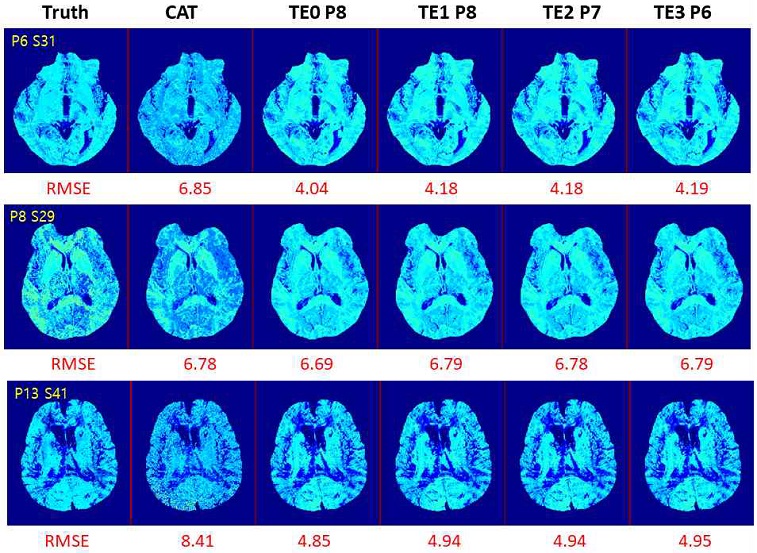
그림 3에는 원본 OEF 영상, CAT 방법의 OEF 영상, 제안하는 방법의 다양한 TE 및 샘플수 모델로 계산한 OEF 영상을 도시하였다. 그림에서 CAT 방법과 제안하는 방법이 원본 OEF 영상과 거의 유사한 것을 알 수 있으며, 일부 영역에 있어서 제안하는 방법이 더 유사한 것을 확인할 수 있다.
Ⅴ. 결론 및 향후 과제
다양한 뇌 질환으로 인한 사망률이 높으며, 이를 검사하기 위한 기술들이 개발되고 있고 MRI는 인체에 해가 없어 임상에서 많이 활용된다.
본 논문에서는 뇌 MRI를 통해 뇌 조직의 혈액 내 OEF를 계산하여 정상적 활동을 분석하는 방법을 제안하였다. QQ 모델을 기반으로 하여 가정 조건을 완화하였고, 잡음 민감성 문제도 해결하였다. 특히, MRI 신호 감쇠 모델링을 통한 파라미터를 활용함으로써 TE 변경이나 샘플수가 다른 다양한 촬영 조건에서 동작하기 어려운 기존 방법의 문제들를 해결하여 안정적으로 OEF를 계산하였다.
최근 딥러닝 기술이 다양한 분야에서 활용되고 있다. 따라서 QQ 모델 기반의 OEF 계산에 있어서 딥러닝을 도입하여 성능 향상 등의 모색이 차후 연구로 필요할 것으로 생각된다. 제안하는 방법은 일반화된 처리 구조로 설계되어, 뇌 외에도 타 조직 질환 분석에 활용이 가능할 것으로 판단된다.
Acknowledgments
This research was supported by Kumoh National Institute of Technology(202001940001)
References
-
M. D. Luca and J. Olesen, "The Cost of Brain Diseases: A Burden or a Challenge?", Neuron, Vol. 82, No. 6, pp. 1205-1208, Jun. 2014.
[https://doi.org/10.1016/j.neuron.2014.05.044]

-
H. Y. Lee, N. C. F. Codella, M. D. Cham, J. W. Weinsaft, and Y. Wang, "Automatic left ventricle segmentation using iterative thresholding and an active contour model with adaptation on short-axis cardiac MRI", IEEE Trans. on Biomedical Engineering, Vol. 57, No. 4, pp. 905-913, Apr. 2010.
[https://doi.org/10.1109/TBME.2009.2014545]

-
J. Cho, Y. Kee, P. Spincemaille, T. D. Nguyen, J. Zhang, A. Gupta, S. Zhang, and Y. Wang, "Cerebral metabolic rate of oxygen (CMRO2) mapping by combining quantitative susceptibility mapping (QSM) and quantitative blood oxygenation level-dependent imaging (qBOLD)", Magnetic Resonance in Medicine, Vol. 80, No. 4, pp. 1595-1604, Mar. 2018.
[https://doi.org/10.1002/mrm.27135]

-
J. Cho, S. Zhang, Y. Kee, P. Spincemaille, T. D. Nguyen, S. Hubertus, A. Gupta, and Y. Wang, "Cluster analysis of time evolution (CAT) for quantitative susceptibility mapping (QSM) and quantitative blood oxygen level‐dependent magnitude (qBOLD)‐based oxygen extraction fraction (OEF) and cerebral metabolic rate of oxygen (CMRO2) mapping", Magnetic Resonance in Medicine, Vol. 83, pp. 844-857, Sep. 2019.
[https://doi.org/10.1002/mrm.27967]

-
H. Y. Lee, "OEF Calculation Method through Cluster-based Classification and Matching of QQ Model on Brain MRI Images", Journal of KIIT, Vol. 18, No. 10, pp. 15-23, Oct. 2020.
[https://doi.org/10.14801/jkiit.2020.18.10.15]

-
D. S. Bolar, B. R. Rosen, A. Sorensen, and E. Adalsteinsson, "Quantitative imaging of extraction of oxygen and tissue consumption (QUIXOTIC) using venular-targeted velocity-selective spin labeling", Magnetic Resonance in Medicine, Vol. 66, No. 6, pp. 1550-1562, Jun. 2011.
[https://doi.org/10.1002/mrm.22946]

-
C. J. Gauthier and R. D. Hoge, "Magnetic resonance imaging of resting OEF and CMRO2 using a generalized calibration model for hypercapnia and hyperoxia", NeuroImage, Vol. 60, No. 2, pp. 1212-1225, Apr. 2012.
[https://doi.org/10.1016/j.neuroimage.2011.12.056]

-
R. G. Wise, A. D. Harris, A. J. Stone, and K. Murphy, "Measurement of OEF and absolute CMRO2: MRI-based methods using interleaved and combined hypercapnia and hyperoxia", NeuroImage, Vol. 83, pp. 135-147, Dec. 2013.
[https://doi.org/10.1016/j.neuroimage.2013.06.008]

-
X. He and D. A. Yablonskiy, "Quantitative BOLD: mapping of human cerebral deoxygenated blood volume and oxygen extraction fraction: default state", Magnetic Resonance in Medicine, Vol. 57, No. 1, pp. 115-126, 2007.
[https://doi.org/10.1002/mrm.21108]


1997년 : 성균관대학교 정보공학과 (학사)
1999년 : KAIST 전산학과 (공학석사)
2006년 : KAIST 전자전산학과 (공학박사)
2008년 ~ 현재 : 금오공과대학교 컴퓨터소프트웨어공학과 교수
관심분야 : Digital Forensics, Image Processing, IoT