딥러닝 기법을 이용한 망막 혈관 분할
초록
당뇨망막증은 망막의 말초혈관에 순환장애가 일어나 발생하는 당뇨병의 합병증으로, 이를 진단하기 위하여 미세혈관류를 분할하였다. 기존 필터와 특징을 사용한 혈관분할은 두꺼운 혈관은 비교적 잘 분할을 하나, 미세한 혈관에 대해서는 정확도가 떨어진다는 단점이 있다. 그리하여 전처리로 노이즈 제거를 위한 필터, 영상 대비를 위한 히스토그램 평활화를 사용하였으며, 픽셀 단위 분할을 위해 딥러닝 기법을 이용하였다. 기존 방법의 정확도는 90% ~ 94%이며, 제안한 방법의 정확도는 95%이다. 결과 영상에서 시신경 유두 및 삼출몰 주변에서 분할 오류가 나타나는 문제점이 있으나, 이는 네트워크 깊이가 얕음에 의한 오류로 향후 네트워크 변경을 통해 정확도를 개선할 수 있다.
Abstract
Diabetic retinopathy is a complicated form of diabetes due to circulatory disorder in the peripheral blood vessels of the retina. We segment the microvessel for diagnosing diabetic retinophathy. The conventional methods using filter and features can segment the thick blood vessels, but it has relatively weak for segmenting fine blood vessels. In pre-processing step, noise reduction filter and histogram equalization are applied to suppress the noise and enhance the image contrast. Then, deep learning technique is used for pixel-by-pixel segmentation. The accuracy of conventional methods is between 90% to 94%, while the proposed method has improved as 95% accuracy. There is a problem of segmentation error around the optic disc and exudate due to the network depth. However the accuracy can be improved by modifying the network architecture in the future.
Keywords:
retinal imaging, blood vessel, deep learning, segmentationⅠ. 서 론
과거 헬스 케어는 전염병을 예방하고 질병 치료를 중심으로 하였으나, 최근에는 인구 고령화와 소비자 맞춤형 의료 서비스에 대한 관심이 커지면서 예방 및 관리의 시대를 맞이하고 있다. 또한 기존 의료 기술에 ICT 기술을 융합하여 저비용, 고효율의 의료 서비스와 고부가가치를 창출하는 유-헬스케어(u-Health) 기술의 중요성이 더욱 커지고 있다.
당뇨망막증(Diabetic Retinopathy)[1][2]은 망막의 말초혈관에 순환장애가 일어나 발생하는 당뇨병의 합병증으로서, 당뇨병의 경우 2030년에는 세계 인구 10명중 1명이 당뇨병을 앓을 것으로 예상된다.
안저 검사는 망막질환을 진단하기 위한 검사로써, 이를 진단하기 위해서는 시신경유두(Optic Disc), 혈관(Blood Vessel), 삼출물(Exudate)등을 검출 및 분할하여야 한다.
기존 고전적인 필터와 특징 추출기를 사용하여 혈관 분할을 할 경우 두꺼운 혈관은 비교적 잘 분할을 하나, 미세한 혈관에 대해서는 정확도가 떨어진다는 단점이 있었다. 그리하여 미세한 혈관에 대해서도 정확히 분할할 수 있는 방법을 고려하여 딥러닝을 사용하였다.
Ⅱ. 제안방법
2.1 전처리
기존 컬러 망막 영상 중 초록(Green) 채널 영상만을 입력으로 사용하는 단일 채널 흑백 영상 방식과 달리 제안한 방법은 빨강, 초록, 파랑 채널 중 빨강과 초록 채널의 평균으로 사용하였다.
본 논문에서 여러 데이터셋에서 입력 영상을 사용하기 때문에 영상을 정규화하기 위해 픽셀 값을 [0, 1]로 만들어주어 일반화 하였다. 일반적으로 영상의 전처리 과정인 노이즈 제거를 위하여 가우시안 분포에 기초한 필터인 양방향 필터[3]를 이용하여 노이즈를 제거하는 동시에 혈관 경계를 최대한 보존하였다.
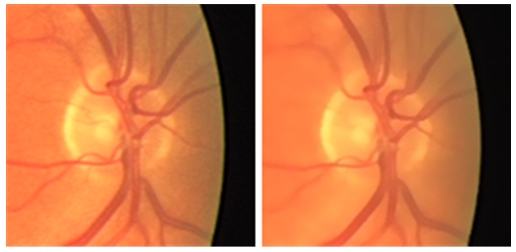
또한 영상 중심부 또는 주변부의 미세 혈관을 정확하게 분할하기 위하여 CLAHE(Contrast Limited Adaptive Histogram Equalization) 방법을 이용한 히스토그램 평활화를 하였는데, 이는 영역기반으로 나누어 평활화를 한 것으로 그림 1과 같이 일반적인 평활화에 비해 전반적으로 대비를 좋게 하였다.
2.2 픽셀단위 분할을 위한 네트워크
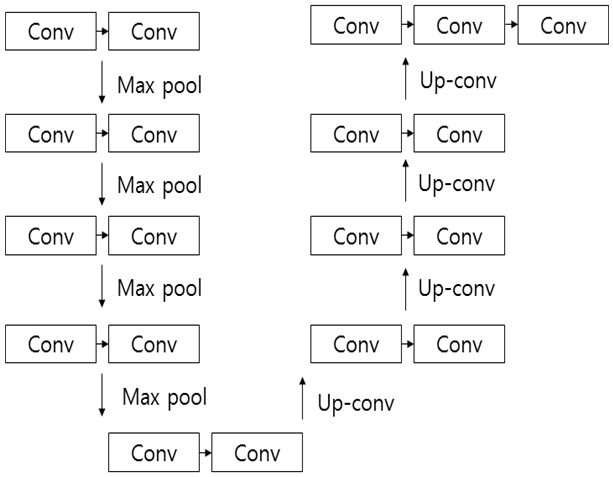
Fully convolutional 레이어[4]는 픽셀단위로 클래스가 라벨링되어 픽셀 단위 예측이 가능하고 지도 라벨링된 영상을 통해 지도학습이 가능하게 되었다. 하지만 Fully convolutional 레이어의 추론 결과는 대상 물체의 크기에 의존적이다. 물체가 큰 경우 파편화되거나, 작은 경우는 배경으로 무시되는 경향이 있다. 이를 위해 그림 2와 같은 UNET[5]이라는 학습 모델을 사용하였는데, 이는 자동 인코더와 유사하지만 마지막 단에 Fully convolutional 레이어를 대신하여 컨벌루션(Convolution)을 사이즈 1×1로 사용하고 결과물에 softmax한 결과가 출력된다.
또한 차원 축소 엔코더(Dimension Reduction Encoder)와 차원 증가 디코더(Dimension Increment Decoder)를 사용하여 차원공간이 보존된다. 차원 축소 엔코더는 컨볼루션 신경망(Convolutional Neural Network)과 같은 형식으로 사용되며, 다운 샘플링을 하고, 입력 영상을 이용하여 특징맵을 만들며 입력 시 여러 노드가 단일 출력결과를 생성한다. 차원 증가 디코더의 업샘플링은 업컨벌루션(Up-convolution)을 사용하여 위의 차원 축소 엔코더를 통해 필터의 계수를 학습하는 과정을 통해서 결정된 필터를 사용하여 결과물을 얻는다.
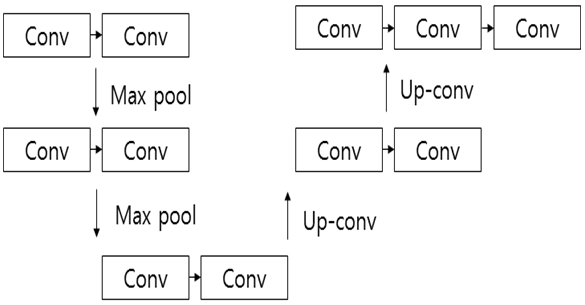
기존 UNET은 23개의 컨벌루션 레이어를 사용하는데 반해, 제안한 알고리즘에서는 그림 3과 같이 13개의 레이어로 축소하여 설계하였다.
학습 과정 배치 정규화(Batch Normalization)[6]을 사용하는데, 기존에는 y = weight × x + bias를 이용한 것으로, 학습 시 초기의 값이 뒤로 갈수록 공분산에 의해 의미가 줄어들어 학습 효과를 감소시킨다.
| (1) |
그에 반해 위 식인 배치 정규화를 사용하여 공분산에 의한 초깃값이 흐려지는 것을 방지해주었다.
2.3 네트워크 인코더 구조
각각의 컨벌루션 블록은 3×3 컨벌루션 2개로 이루어져있고 활성함수는 ReLU를 사용하며, dropout 0.2 이다. 이후 2×2 맥스 풀링(Max Pooling)하여 2배의 특징 채널을 사용하며, 이러한 컨벌루션 과정을 4번 진행함으로 최소 입력 사이즈는 32×32이다. 이 과정에서 입력 영상의 세부 객체의 특징을 포함하는 특징맵을 출력한다.
2.4 네트워크 디코더 구조
반면 디코더는 인코더이서 학습된 필터를 이용하여 디컨벌루션 연산한 것 즉, 맥스 풀링 되기 전의 특징맵을 크롭(Crop)하여 디컨벌루션 할 때 연결하여 업샘플링을 하게 된다. 디코더 또한 인코더와 마찬가지로 3×3 컨벌루션을 사용하고 활성함수로 ReLU를 사용하였다.
2.5 학습
학습은 최적의 매개변수를 찾기 위해 확률적 경사 하강법(Stochastic Gradient Descent)[7]를 사용하였는데, 이때 손실함수의 기울기를 계산하고, 학습률(η=0.001)을 기반을 두어 기존의 가중치 값을 업데이트 하였다.
| (2) |
그래픽 처리장치 메모리의 사용량을 최대화시켜 빠른 학습 보다는 정확한 학습을 위해 입력 사이즈(원본 영상의 1/5)를 크게 주고 배치 사이즈를 줄여 학습시켰다.
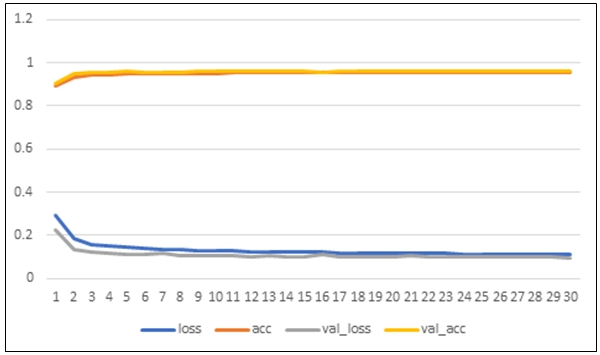
그림 4와 같은 손실함수는 틀린 데이터에 치중하는 특성을 가진 평균 제곱근 편차(Mean of Sum of Square Error)를 대신하여 옳은 결과와 그른 결과에 동등한 영향을 주는 크로스 엔트로피(Cross Entropy)를 이용하여 구현하였다.
| (3) |
여기서 P는 데이터 X와 네트워크 가중치 W를 바탕으로 기준값을 얻을 확률이고 Q는 P에 근사하도록 만들고 싶은 즉, 모델이 예측하는 결과이다. 학습 반복 횟수는 20회로 고정하여 실험을 진행하였다.
학습 과정에서 최대한 오버피팅(Over-fitting)을 줄이기 위하여 검증 손실 함수(Validation Lss Function)의 결과가 이전의 결과 값에 대하여 줄지 않으면 학습 모델의 가중치 데이터를 초기화하지 않았다.
2.6 후처리
제안된 딥러닝 시스템의 출력은 아래와 같이 softmax 함수를 사용하였다.
| (4) |
결론적으로 출력 영상의 픽셀값은 혈관일 확률이다. 따라서 일정한 확률값 이상일 때 존재함을 결정하기 위해 본 논문에서는 Otsu thresholding[8]을 사용하여 자동으로 라벨링을 결정하였다.
Ⅲ. 실험 결과
본 실험에서는 실험 장비의 한계(GTX 960M)에 의해서 UNET 네트워크를 축소하여 사용하였으며, 실험에 사용된 데이터셋은 DRIVE(https://www.isi.uu.nl/Research/Databases/DRIVE/)[9], STARE(http://cecas.clemson.edu/ahoover/stare/)[10], 데이터셋이다. DRIVE는 총 40장의 영상(정상 영상 33장)이며, 영상의 크기는 565×584 이다. STARE는 총 81장의 영상(정상 영상 31장)이며, 영상의 크기는 605×700 이다.
각각의 데이터 셋은 서로 다른 전문가에 의해 만들어진 두 가지 기준값이 있다. 두 가지 중 첫 번째 기준값 영상을 사용하여 실험을 평가하였다. 실험 결과에서의 정확도 평가는 다음의 식을 활용하여 산출하였다.
| (5) |
TP(True Positive)는 실제 True인 정답을 True라고 분류, FP(False Positive)는 실제 False인 정답을 True라고 분류, FN(False Negative)는 실제 True인 정답을 False라고 분류, TN(True Negative)는 실제 False인 정답을 False라고 분류하는 것을 의미한다. 훈련과 평가는 데이터 셋에서 7: 3의 비율로 랜덤으로 뽑아서 평가하였으며, 수치적인 정확도(Accuracy)는 전처리 이전과 이후로 구분하였다.
표 1은 기존 방법과 제안한 방법의 정확도를 비교이며, 딥러닝과 앙상블 러닝의 하이브리드 방법(93.27%), 딥 컨벌루션 뉴렬 네트워크(94.70%), 멀티 스케일 컨벌루션 뉴렬 네트워크(90.70%), 그리고 제안한 방법(95.23%)이 기존 방법에 비해 높은 정확도를 나타내었다. 이는 두꺼운 혈관 보다 미세한 혈관에 대한 정확도 향상을 의미한다. 또한 그림 5에서도 볼 수 있듯이 비교적 정확하게 혈관을 분할하였다.
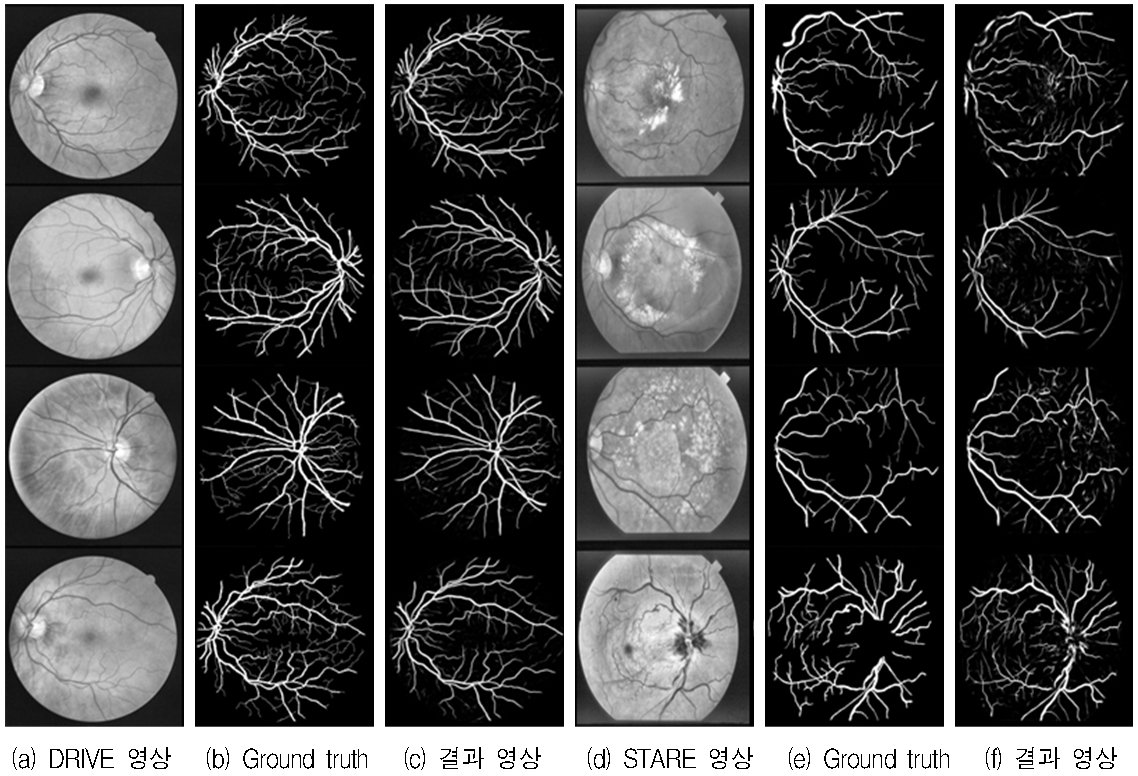
Output image comparison Ⅰ: (a) DRIVE image, (b) Ground truth, (c) Output image, (d) STARE image, (e) Ground truth, (f) Output image
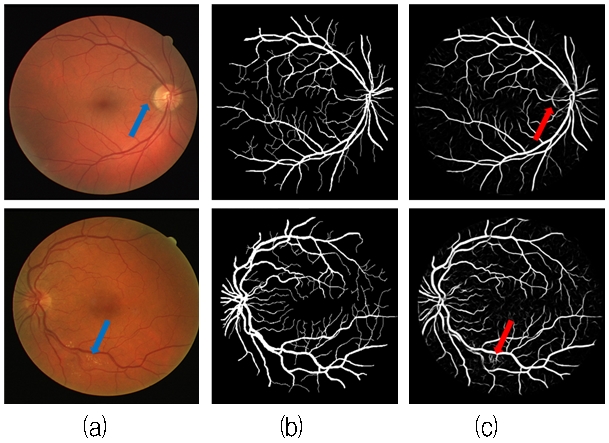
그림 6과 같은 결과 영상에서의 혈관 분할에서 시신경 유두의 가장자리 부분이나 삼출물의 가장자리 부분이 노이즈로 포함되어 같이 검출되는 오류가 있다. 이 부분은 네트워크의 깊이가 얕음에 의해 발생한 오류로 간주된다.
Ⅳ. 결 론
본 논문에서는 당뇨망막증 진단을 위한 혈관 분할 연구를 수행하였다. 제안한 방법은 분할 정확도가 약 95% 이다. 향후 미세혈관의 분할 정확도 개선을 통해 최종 정확도를 개선할 수 있다. 네트워크를 충분히 깊게 설계한다면 텍스처(Texture)와 미세혈관 등에도 충분히 개선될 여지가 있다고 판단한다.
Acknowledgments
이 논문은 2018년도 한국산업기술대학교 학술연구진흥사업에 의하여 연구되었으며, 정부의 재원으로 한국연구재단의 지원을 받아 수행된 사업임(No. 2017R1D1A1B03033526, NRF-2017R1A6A1A03015562)
References
-
V. Gulshan, L. Peng, M. Coram, M. Stumpe, D. Wu, A. Narayanaswamy, S. S. Venugopalan, K. Widner, T. Madams, and J. Cuadros, "Development and validation of a deep learning algorithm for detection of diabetic retinopathy in retinal fundus photographs", JAMA, 316(22), p2402-2410, Dec.), (2016.
[https://doi.org/10.1001/jama.2016.17216]

-
R. Gargeya, and T. Leng, "Automated identification of diabetic retinopathy using deep learning", Ophthalmology, 124(7), p962-969, Jul.), (2017.
[https://doi.org/10.1016/j.ophtha.2017.02.008]

-
C. Tomasi, and R. Manduchi, "Bilateral Filtering for Gray and Color Images", In Proceedings of the IEEE International Conference on Computer Vision, Bombay, India, India, p839-846, Jan.), (1998.
[https://doi.org/10.1109/iccv.1998.710815]

-
J. Long, E. Shelhamer, and T. Darrell, "Fully convolutional networks for semantic segmentation", Proceedings of the IEEE conference on computer vision and pattern recognition, p3431-3440, (2015).
[https://doi.org/10.1109/cvpr.2015.7298965]

-
O. Ronneberger, P. Fischer, and T. Brox, "U-Net: Convolutional Networks for Biomedical Image Segmentation", International Conference on Medical image computing and computer-assisted intervention, p234-241, May), (2015.
[https://doi.org/10.1007/978-3-319-24574-4_28]

- S. Ioffe, and C. Szegedy, "Batch normalization : Accelerating deep network training by reducing internal covariate shift", Proceedings of the ICML, p448-456, Mar.), (2015.
-
L Bottou, "Large-scale machine learning with stochastic gradient descent", Proceedings of COMPSTAT, p177-186, (2010).
[https://doi.org/10.1007/978-3-7908-2604-3_16]

-
N. Otsu, "A threshold selection method from gray-level histograms", IEEE Transactions on Systems, Man, and Cybernetics, 9(1), p62-66, Jan.), (1979.
[https://doi.org/10.1109/tsmc.1979.4310076]

-
J. Staal, M. Abr`amoff, M. Niemeijer, M. Viergever, B. van Ginneken, "Ridge based vessel segmentation in color images of the retina", IEEE Transactions on Medical Imaging, 23(4), p501-509, Apr.), (2004.
[https://doi.org/10.1109/tmi.2004.825627]

-
A. Hoover, V. Kouznetsova, and M. Goldbaum, "Locating blood vessels in retinal images by piecewise threshold probing of a matched filter response", IEEE Transactions on Medical Imaging, 19(3), p203-210, Mar.), (2000.
[https://doi.org/10.1109/42.845178]

-
D. Maji, A. Santara, S. Ghosh, D. Sheet, and P. Mitra, "Deep neural network and random forest hybrid architecture for learning to detect retinal vessels in fundus images", 37th annual international conference of the IEEE Engineering in Medicine and Biology Society (EMBC), p3029-3032, Aug.), (2015.
[https://doi.org/10.1109/embc.2015.7319030]

- D. Maji, A. Santara, P. Mitra, and D. Sheet, "Ensemble of deep convolutional neural networks for learning to detect retinal vessels in fundus images", arXiv preprint arXiv:1603.04833, (2016).
-
M. Li, Q. Yin, and M. Lu, "Retinal Blood Vessel Segmentation Based on Multi-Scale Deep Learning", 2018 Federated Conference on Computer Science and Information Systems (FedCSIS), p1-7, Sep.), (2018.
[https://doi.org/10.15439/2018f127]


2019년 : 한국산업기술대학교 메카트로닉스공학과(학사)
2019년 : 탑엔지니어링
관심분야 : 머신비전, 딥러닝

2004년 2월 : 고려대학교 제어계측 공학과(공학사)
2008년 2월 : 광주과학기술원 정보기전 공학부(공학석사)
2013년 2월 : 광주과학기술원 기전공학부(공학박사)
2019년 5월 현재 : 한국산업기술학교 메카트로닉스공학과 교수
관심분야 : 영상처리, 컴퓨터 비전, 정합, 융합